MI Little – Wikipedia
Mimivirus Est un genre de virus dans la famille de Mimiviridae , [6] servir d’hôtes naturels. Avec la famille Mimiviridae entendu Mimivirus Aux virus géants dans le phylum du Nucléocytoviricota (aussi Anglais Virus d’ADN nucléocytoplasmique , NCLDV; proposition originale ” Nucléocytoplasmaviricota “;; Une proposition encore antérieure est une commande ” Mégavirales «S. l. [7] [5] ).
Dans le genre Mimivirus Il y a un seul type confirmé d’ici mars 2019 auprès du Comité international de la taxonomie des virus (ICTV) Acanthamoeba Polyphaga Mimivirus ( À propos ), qui est donc aussi le type de type. [8] Dans le langage familier et dans la littérature plus ancienne, l’APMV est généralement uniquement Mimivirus désigné. Cependant, il existe toute une série de grands virus liés phylogénétiquement qui sont proposés comme autres membres de ce genre.
Le Mimivirus L’APMV a été découvert dans une tour de refroidissement industrielle à Bradford (Angleterre) en 1992 lors de la recherche sur la maladie du légionnaire (Légionellose), et il a été constaté qu’il a été trouvé dans l’Amöbe Acanthamoeba Polyphaga augmenté. En 2003 c’était au Université de la Méditerranée identifié à Marseille d’un groupe de travail autour de Didier Raoult. [9] Avec un diamètre de 400 nm, les particules virales (virions) de l’APMV ont la taille de petites bactéries. [dix] En raison de cette taille et de cette similitude externe avec les bactéries sphériques (COCCI), il a été initialement considéré comme une bactérie positive à Gram et l’a appelé Bradfordcoccus . Lorsque vous avez reconnu l’erreur, le virus nouvellement découvert a été nommé en allusion à ses propriétés de taille et de coloration Imitation du virus , virus trompeur. Enfin, il en est sorti Mimivirus , avec la section du nom moi Comme abréviation pour Anglais imitation de microbe . En octobre 2004, Didier Raoult et ses collègues étaient la structure de son génome dans le journal Science publié. [11] Mimivirus Soit dit en passant, ce n’était pas le seul virus qui s’est produit: un autre exemple est que Milieu de microport (proposé pour Pithoviridae ), Zunächst Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, a déclaré Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena, Felipe Assise, Fabio Dornas, Jacques , Christelle Denes, Anthony Levasseur, Philip Pe Colson, Jonatas Abrahão, Bernard La Scola. [douzième]
La même équipe que l’APMV a découvert plus tard a découvert un virus légèrement plus grand qui Acanthamoeba Castellanii Mamavirus (ACMV, Kurz Mamavirus ) Avec les virophages Spoutnik qui l’ont infecté. ACMV et APMV sont si étroitement liés qu’ils sont généralement dans le même genre Mimivirus être placé. [13]
Jusqu’en 2013, en tant que virus encore plus grand, que Pandoravirus , le genre a montré les virus Mimivirus le plus grand diamètre de capuchon de tous les virus connus; En attendant, cependant, il est également de parents proches Mégavirus chilensis , Tupanvirus , Platanovirus , ainsi que d’autres virus géants comme celui Pithovirus dépassé.
La première connaissance Mimivirus -Wirt est l’amibe Acanthamoeba Polyphaga (Genre Acanthamoeba , Amoebozoa). Jusqu’à présent, seuls les représentants du genre ont pu Acanthamoeba , sauf A. polyphaga toujours A. Castellanii et A. Mauritaniensis , lors de l’hébergement de ce virus, aucune cellule d’autres organismes individuels ou multiples. [14] Les hôtes naturels sont inconnus (en 2015). [15]

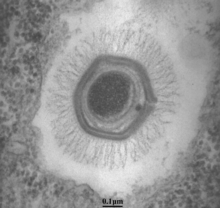
Les capsides des virions (particules virales) d’APMV apparaissent sous un microscope électronique hexagonal, donc la géométrie du capuchon est
Ikosaedrisch. [17] Il ne semble pas y avoir de coquille virale externe, ce qui indique que le virus ne laisse pas la cellule hôte par exocytose. [14]
La principale protéine du Mimivirus -Kapsids se compose de deux domaines du type de rouleau de biscuit ( Anglais Pli de rouleau de gelée ). Cette protéine forme un capsomère homo -rimer en tant qu’unité organisationnelle du kapside. Les capsomers sont emballés par hexagonale sous la forme de “marguerites”: six capsomers entourent un approfondissement entre eux. [17] [18]
Les virions de Mimivirus Avoir un diamètre de capuchon de 400 nm.
Fibrilles [ Modifier | Modifier le texte source ]]

B : Enregistrement AFM de deux fibres de surface détachées du Mimivirus .
C: Cryo-em-image of One Mimivirus Après démantèlement partiel des fibrilles à l’aide de bromélaïne.
D: Image AFM des fibres internes du Mimivirus .
Le Kapsid est recouvert d’une couche compacte de fibrilles (tégument).
Les filaments protéiques (fibrilles) exceptionnels de la surface du capuchon ont une longueur d’environ 100 nm (80–125 nm) et portent ainsi la longueur totale d’un virion à 600 nm. [5] [15] Les écarts dans la littérature scientifique rendent les nombres très imprécis lorsque, par exemple, la «taille» du virion est parfois spécifiée comme entre 400 et 800 nm. En dehors des différences entre les arbres viraux individuels dans le genre Mimivirus Parfois, la taille totale est spécifiée avec des filaments, et parfois le diamètre du capuchon pur.
Des études de ces filaments par Klose et al. (2010) à l’aide d’un microscope à force de balayage montrent que ceux-ci sont souvent attachés à une structure de support commune.
À ce moment-là, cependant, il ne pouvait pas être trouvé aux parties de la surface du capuchon que ces sangles sont attachées. [17] Chaque fibrille se termine par un petit capuchon sphérique d’une protéine avec une fonction inconnue. [15] [17] Les fichiers protéiques se sont révélés résistants aux protéases à moins qu’ils soient traités avec du lysozyme. Les filaments semblaient donc recouverts de peptidoglycane. C’était en bon accord avec le fait que Mimivirus À travers le Méthode de gramme permet. [19]
Avec leur surface fortement glycosylée, les filaments jouent apparemment un rôle important dans le rapprochement avec les maisons économiques et l’infection ultérieure. [19] [15] Le composant principal des fibres est la protéine R135 (à côté de L725 et L829). Sa structure est similaire aux protéines de la famille de la glucose-méthanol-cholin-oxydo-durase (GMC oxydoréductase), qui ont un domaine de liaison à la FAD N-terminal et un domaine d’étiquette de substrat C-terminal.
Le prochain homologue R135 est une alcool aryl oxydase [20] qui est impliqué dans l’extraction de lignine biologique des parois des cellules végétales. R135 pourrait ainsi être impliqué dans la perforation de la paroi cellulaire de ses hôtes naturels, en particulier les algues contenant des algues. [15] Dans des conditions de laboratoire, cependant, aucune des trois protéines mentionnées n’est absolument nécessaire à l’infectiosité. [15]
Stargate [ Modifier | Modifier le texte source ]]
![TEM-Aufnahme eines Mimivirus-Virions (APMV) mit Stargate und Filamenten.[16][Anm. 1]](https://upload.wikimedia.org/wikipedia/commons/thumb/4/4f/F21-01-9780123846846_mimivirus_particle_stargate.png/175px-F21-01-9780123846846_mimivirus_particle_stargate.png)


Ce qui est frappant, c’est la structure pentagonale en forme d’étoile dans l’un des coins du capuchon, le «Stargate», appelé So, tire-étoile , [21] Anglais pour “Star Gate”, allemand également appelé «structure étoile» ou «structure navale»). [22] Si vous regardez directement ce point d’angle (le centre de l’étoile), il semble y avoir cinq zones triangulaires entre ses rayons.
Les rayons ont une largeur d’environ 50 nm, une épaisseur de 40 nm et une longueur de 200 nm; Vous atteignez presque les pierres angulaires voisines des Kapsids Ikosaedrian. La porte d’étoile n’est pas couverte par des fibrilles. [15] La présence de cette structure modifie la géométrie du capuchon en déviant de la forme idéale en cuir ICOSA: conformément à une inspection plus approfondie, un seul axe avec une symétrie à cinq faisceaux traverse le virion, qui traverse le centre de l’étoile (appelé sommet). [17] [23]
La symétrie du capuchon est spécifiée différemment avec un numéro de triangulation t = 972–1141 ou t = 1200. [6]
Étant donné qu’aucune dépression ordonnée par hexagonale ne peut être observée à la surface de la structure de l’étoile, il est supposé qu’il s’agit d’une protéine qui diffère de la protéine principale du capuchon. [17]
Le Stargate joue un rôle spécial dans l’infection de la cellule hôte:
Pendant l’infection, le “fermoir” s’ouvre sur l’apex et le noyau viral est libéré (avec l’ADN et les protéines préfabriquées) du Kapsid dans le cytosol de la cellule hôte (via la phagocytose).
C’est la raison pour laquelle la structure des étoiles comme une “porte d’étoile” ( Anglais stargate ) appelé. [24]
Nucléocapses [ Modifier | Modifier le texte source ]]
Le Mimivirus A plusieurs caractéristiques morphologiques avec tous les membres du groupe de virus NCLDV.
Immédiatement sous le capuchon du Mimivirus Par exemple, il existe deux couches de densité électronique qui sont interprétées comme des membranes. [19] Sous ces membranes, il y a une couverture de protéine d’environ 7 nm d’épaisseur dans laquelle l’ADN à double parcours linéaire du virus est enfermé.
Ce noyau central condensé du virion apparaît sous le microscope électronique comme une zone sombre, le «nucléokapside» si appelé.
Le grand génome du virus est situé dans cette zone, et les ARNm et les protéines préfabriquées.
Étant donné que tous les autres NCLDV ont une couche lipidique interne qui entoure le noyau central, vous soupçonnez également que Mimivirus .
Les parois du nucléocapside sont à environ 30 nm derrière les parois du capuchon, dans la zone de la structure des étoiles (le Stargate), la surface du nucléocapside est également abaissée. [17] On pense que l’espace entre la pointe de la structure d’étoile et le nucléocapside est rempli d’enzymes hydrolytiques nécessaires à la pénétration du virus dans la cellule hôte.
Des brins de protéines internes ont été découverts entre le kapside et le nucléokapside, qui stabilisent apparemment le positionnement spatial mutuel des deux parties. [19]
Par rapport à la plupart des autres virus, l’APMV a un génome important et complexe qui se compose d’un seul brin à double brin d’ADN linéaire (ADNdb). La longueur du génome du type sauvage APMV (variant de sortie Mimivirus M1) était de Raoult et al. (2004) spécifiés avec 1 181 404 pb, [11] Cette valeur a été faite par Bäckström et al. (2019) légèrement corrigés à 1 181 594 pb. [25] Cela correspond à environ 800 nm. Le mimivirus variant sans fibre M4 n’a que 0,993 MBP, entre M2 avec 1,10 MBP et M3 avec 1,10 MBP. [26] Le contenu GC de l’APMV est de 28%.
Chez APMV 1260, il y a des cadres de lecture ouverts (ORFS, Anglais cadres de lecture ouverts ), [11] Parmi eux prévoyaient 979 gènes de codage. [25] [15] [27] Cela va bien au-delà de l’équipement minimum de 4 gènes nécessaires à un virus, tels que les phages MS2 et Qβ. [28] Des études détaillées sur le génome corrigent à plusieurs reprises les erreurs de séquence et peuvent découvrir de nouveaux cadres de lecture. [29]
La proportion d’ADN non codante n’est donc qu’environ 9,5 à 10%. Les cadres de lecteur ouvert sont séparés par des lacunes d’environ 157 paires nucléotides. Deux sections d’ADN avec le nom R ( Anglais droite – à droite) et l ( Anglais gauche – à gauche) code à peu près le même nombre de gènes (450 ou 465, selon les données de 2010). Le contenu GC est 28% bas. Près des extrémités de la molécule d’ADN se trouvaient des «répétitions inversées» ( Anglais répétitions inversées ) trouvé avec 617 paires nucléotides. On pense que l’interaction mutuelle de ces lieux peut conduire à la formation d’un ADN circulaire Q Q avec deux petites extensions. [29]
Au cours de l’analyse, au moins 21 gènes homologies, y compris ceux qui ont jusqu’à présent été connus de aucun autre virus, mais uniquement des organismes cellulaires, ont été trouvés. [11] [9] [30] 43 gènes sont homologues à ces autres virus géants (NCCLDV). [15] Comme les autres virus géants contient Mimivirus Plusieurs gènes pour le métabolisme du sucre, des lipides et des acides aminés. Il y avait également des gènes métaboliques qui n’ont été trouvés dans aucun autre virus. [14]
Plusieurs transcrits d’ARNm ont pu être obtenus à partir de virions nettoyés.
Comme pour les autres NCLDV, les transcrits de l’ADN polymérase, une protéine de capuche et un facteur de transcription de type TFII ont été trouvés en particulier. Cependant, trois transcrits synthétiques aminoacyl-ARNt différents et quatre molécules d’ARNm inconnues ont été trouvées pour cela Mimivirus sont spécifiques. Ces transcriptions pré-préparées peuvent être traduites sans expression du gène viral et sont susceptibles de se répliquer Mimivirus nécessaire. D’autres virus d’ADN comme ça Cytomégalievievus humain et le Virus herpes simplex Le type 1, contenait également des transcrits d’ARNm emballés. [14]
Mimivirus est l’un des rares virus d’ADNdb dans le génome duquel une séquence codant pour l’inteine a été démontrée. Les Inteins sont un domaine protéique qui catalyse leur propre distance d’une molécule porteuse et de la liaison ultérieure des extrémités formées.
Une telle séquence est dans Mimivirus -Gen pour l’ADN polymérase B disponible. [trente et un]
En raison de l’équipement génétique exceptionnellement complexe du virus vivant et nature inanimée Bien sûr, c’est-à-dire que les «êtres vivants» doivent être définis.
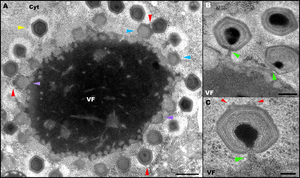
UN) Enregistrement TEM d’une usine de virus intracellulaire, avec des particules virales (virions) de Mimivirus Dans divers stades de montage
– VF: Virus Factory (viroplasma)
– CYT: Cytoplasme
– Arrows violets: Virions vides et encore sans fibres dans les premiers stades de la composition, à proximité immédiate de la périphérie de l’usine de virus qui se produit
– Arrows bleus: Virions vides en partie assemblés et sans fibres
– Flèches jaunes: Virions matures et couverts de fibres qui sont maintenant plus loin de l’usine de virus que les particules immatures
– Flèches rouges: Dans le cas de plusieurs particules de virus, un «Stargate» qui est régulièrement situé au point distal de l’usine peut être vu
B) et C) Enregistrement TEM du Mimivirus -Particule
– Arrows verts: Mimivirus -Artikel sur la scène de «l’emballage d’ADN» (emballage d’ADN)
– Flèches rouges: deux bords d’une porte d’étoile en face du point de l’emballage d’ADN

Les détails et les différentes étapes du cycle de réplication de Mimivirus , comme la liaison évidente à la surface cellulaire et l’entrée dans la cellule, la libération du noyau virus, la réplication de l’ADN, la transcription, la traduction et enfin l’assemblage et la libération de virions de fille, ne sont pas encore suffisamment connus. Cependant, les scientifiques ont créé l’aperçu général donné ci-dessus en utilisant des enregistrements microscopiques électroniques de cellules infectées.
Toutes les étapes du cycle de propagation fonctionnent dans le cytoplasme de la cellule hôte. [32]
L’infection de l’amibe avec un mimivirus a probablement lieu selon le scénario suivant:
- Le Mimivirus -Les vivons sont similaires dans leur taille et la présence de polysaccharides caractéristiques à la surface (voir la coloration Gram, nom). Ils sont donc absorbés par l’amibe comme nourriture pendant un processus d’endocytose. Les polysaccharides agissent comme un récepteur chimique et initient l’attachement. À la suite de l’endocytose, les virions sont dans les endosomes dans la cellule.
- Les filaments de protéines sont partiellement lysés dans les endosomes, ce qui signifie que le capuchon peut interagir avec la membrane d’endosome.
- ~ 2 heures après l’infection: le Kapsid s’ouvre dans la zone de la structure de l’étoile (Stargate), la membrane intérieure fusionnée avec la membrane endosome et le contenu du capuchon sont libérés dans le cytoplasme.
- Après la particule centrale (la partie intérieure du nucléokapside) a quitté le cytoplasme, la synthèse de l’ARNm viral commence en raison de la présence de l’appareil de transcription virale. Ces ARNm s’accumulent à l’intérieur de la particule centrale sous forme de granule. [32] De l’extérieur, le virus semble disparaître et tout dans la cellule semble normal (phase sombre, Anglais phase d’éclipse ).
- 4 à 5 heures après l’infection: l’ADN viral quitte la particule centrale et est déballé pour que la réplication puisse commencer. En conséquence, en plus de la coquille vide de la particule centrale, une «usine de virus» si appelée – un lieu de synthèse des composants individuels des virions et de son assemblage ultérieur est créé. Si plusieurs particules de virus sont entrées dans la cellule, les «usines» qu’elles forment fusionnent en un seul. Vous pouvez maintenant voir une petite accumulation dans certaines zones de la cellule.
- 6 à 9 heures après l’infection: composition (assemblage) du kapside avec un pack d’ADN simultané à la périphérie des “usines de virus”. Une propriété inhabituelle du Mimivirus Est-ce que l’ADN est emballé. [24] Le Mimivirus -Les vivons sont clairement visibles dans la cellule.
- 14-24 heures après l’infection: les cellules amiba sont lysées, c’est-à-dire H. Ils ont éclaté et les virions sont libérés. Plus de 300 unités sont générées par cellule hôte. [29] [14]
La transmission est effectuée par diffusion passive. [6]
Il a été supposé que Mimivirus La cour de certaines formes de pneumonie (pneumonie) pourrait l’être. Ceci est principalement basé sur des preuves indirectes sous la forme d’anticorps contre le virus découvert dans la pneumonie. [33] En raison des quelques publications précédentes, la classification du Mimivirus Actuellement difficile comme pathogène possible. Une grande partie des cas de pneumonie se déroule sans cause déterminable. [34] Bien qu’un Mimivirus En isolé dans le cas d’un morceau souffrant de pneumonie, isolé, [35] Et il y a des indications de cultures cellulaires qui Mimivirus Les macrophages peuvent s’infecter et y être reproduits. [36] Il a donc été observé dans des conditions expérimentales que Mimivirus Les macrophages humains peuvent infecter, c’est-à-dire H. pénétrer les cellules via la phagocytose et peut y s’yor. [37] [36] De plus, des anticorps contre le mimivirus ont été trouvés dans plusieurs études chez un petit nombre de patients atteints de pneumonie. [33] [38] Un cas individuel de pneumonie d’un assistant de laboratoire qui a travaillé avec des cultures de ce virus a également été décrit. Le contenu des anticorps contre le mimivirus dans son sang a également augmenté. [39] Cependant, la présence d’anticorps contre le virus lui-même n’est pas une indication de sa pathogénicité. Il est possible que Mimivirus a simplement de fortes propriétés immunogènes, c’est-à-dire H. déclenche une réponse immunitaire claire. [29] Dans l’un des cas enregistrés, il a également été possible d’isoler le virus sous sa forme pure à partir d’échantillons de liquides reçus par les patients. [40]
Les protéines de fibre R135 et L829 étaient le principal vétéran du Mimivirus Identifié: Cependant, le mimivirus de variant sans fibre M4 n’a montré aucune réactivité avec les sérums de patients humains, ce qui confirme que ces protéines manquent dans M4. [15]
Le Compant-virus Est un virus satellite que les affections mimy linéaires B et C, mais pas les mimivirus de la ligne A. Celles-ci montrent un Mimivire, Anglais élément de résistance à la virophage mimivirus , mentionné la résistance qui fonctionne de manière similaire au système CRISPR / CAS. [41] [42]
Soit dit en passant, il a été prouvé qu’il n’y a pas seulement un gentransfer entre les hôtes Amöboid et les virus géants comme endocytobion virale intracellulaire (les organismes qui vivent ou se multiplient dans les cellules d’autres organismes) [43] , mais même entre les virus et en même temps les endocytobiontes bactériennes existantes. [44] [26] [45]
| Virus | Aminoacyl-trnce-synthétase | MUTS de type octocorallia | Filaments de protéines (long) | Stargate [24] | Bien connu virophagien [quarante-six] | Cytoplasmique Virion |
Se dérober |
|---|---|---|---|---|---|---|---|
| Mégavirus chilensis | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | et | et (75 nm) | et | Non | et | Acanthamöben (sommeil, amibozoa) |
| Mamavirus ACMV | 4 (Tyr, en colère, Met, Cys) | et | et (120 nm) | et | et | et | Acanthamöben (sommeil, amibozoa) |
| Mimivirus À propos (Wildtyp M1) |
4 (Tyr, en colère, Met, Cys) | et | et (120 nm) | et | et | et | Acanthamöben (sommeil, amibozoa) |
| Mimivirus M4 (variante chauve / sans fibre) |
2 (Met, Cys) | Non | Non | et | Reporter | et | Acanthamöben (sommeil, amibozoa) |
| Cafétéria-roenbergensis-virus | 1 (avec) | et | Non | Non | et | et | Phogotrophie Protrozos (hétéroxema, trinsolets) |
Système externe [ Modifier | Modifier le texte source ]]
Le genre Mimivirus Et quelques autres – pas encore confirmés par l’ICTV en mars 2019 – Genres génétiquement similaires et espèces de la famille Mimiviridae (tel que ” Mamavirus “,” Mégavirus ” et ” Moumouvirus «) Formez une klade du mimivirus dans le sens plus large.
Pour cela, il a été proposé d’être ” Mégamivirininae “, [3] ‘ Mégavirinae ” [2] Ou ” Maintenina ” [d’abord] se soulever dans le rang de sous-famille et donc des autres sous-familles du Mimiviridae à différencier. Il en résulte les revêtements suivants:
- CafeteriAVirus Group (groupe II)
- Klosneuvirus groupe – comme une sous-famille possible ” Klosneupirinae ”
- Mimiviridae -GROUP I -Les lignes suivantes émergent en ceci: [2]
- A Lignes: Mimivirus Groupe: Mimivirus dans un sens plus étroit
- Lignes B: Moumouvirus -Groups (Mouuoupies)
- Ligne C: groupe Courdo11 avec ” Mégavirus chilensis «(Espèces, plus Courdo1-Virus)
- Tupanvirus Groupe (virus Tupan)
Depuis jusqu’à présent (mars 2019) de l’ICTV, seul le genre Mimivirus Il reste à confirmer au moment où les candidats sont affectés à cette sous-famille proposée de ce genre et pour lesquelles les propres genres doivent être mis en place. Cependant, il semble certain que les représentants de la ligne A Mimivirus appartenir.
Celui de Wilson et al. En 2017, décrit le candidat ” GVSAG AB-566-O17 “(Du NCBI en tant qu’espèce” Mimivirus AB-566-O17 “désigné [47] ) est plus lié aux auteurs (Fig. 2) à APMV que CROV (mais plus proche que les représentants du groupe OLPG). Il ne doit donc être attribué à aucun des groupes ci-dessus (surtout pas au genre Mimivirus ). [48]
Système intérieur [ Modifier | Modifier le texte source ]]
Systématique de la ligne A (Mimivirus dans le sens plus étroit):
- genre Mimivirus
- Espèces: Acanthamoeba Polyphaga Mimivirus (À propos de) [49] (Confirmé par l’ICTV en mars 2018) – Lieu: Bradford, Angleterre, Royaume-Uni [5] (informel Mimivirus s. s. [50] )
- Mimivirus M1 ( Anglais type sauvage ) [26] – Type sauvage (variante naturelle d’origine) de Bradford
- Mimivirus m1 et m2 – formes intermédiaires entre M1 et M4 avec des fibrilles plus courtes que le type sauvage [26]
- Mimivirus M4 ( Anglais Variante chauve / sans fibre ) – La variante sans feu n’est pas infectée par les virophages Spoutnik [15] [26]
- Mimivirus Bombay (MVB, alias Bombay-Virus) [49] [5] [51] [52] [53] [54] – Emplacement: Mumbai, Inde
- Mimivirus shirakomae (alias shirakomae-virus) [53] – Fundort: Shirakoma Pond, Nagano, Japon [5] [55]
- Mimivirus kasaii (Alias Kasaii Virus) -Location: Arakawa (rivière), Tokyo, Japon [5] [55] [53]
- Virus de samba [49] [56] [51] – Emplacement: Rio Negro, Brésil [5] [57] [58] [59]
- Mimivirus Amazonia (Alias Amazonia Virus, Virus amazonien) [5] [60] [51] – Emplacement: Rio Negro, Brésil [5]
- Virus d’huîtres – Lieu: Florianópolis, Brésil [5] [60]
- Virus en couronne [soixante-et-un] [60] – Emplacement: Lagoa Santa, Brésil [5]
- Le NCBI mène toujours le Moumouvirus saoudien (SDMV) sous APMV, [62] Cependant, comme référence, les travaux de Bajrai et al. (2016) que dans le titre Saudi Moumouvirus, le premier groupe B Mimivirus isolé de l’Asie [63] Correctement sur le groupe d. H. La ligne B (groupe Moumouvirus) fait référence.
- Espèces: ” Acanthamoeba Castellanii Mamavirus «(ACMV, alias Acanthamoeba Castellanii Mimivirus ) [soixante-quatre] [65] [66] – Emplacement: Paris, France [5]
- Espèces: ” Hirudovirus ” [soixante-sept] [68] – Emplacement: Tunisie [5]
- Espèces: Acanthamoeba Polyphaga Mimivirus (À propos de) [49] (Confirmé par l’ICTV en mars 2018) – Lieu: Bradford, Angleterre, Royaume-Uni [5] (informel Mimivirus s. s. [50] )



-
- Espèces: ” Niemeyer-virus «(NYMV) [71] [69] -Location: Pampulha-See, Belo Horizonte, Brésil (nommé en l’honneur de l’architecte Oscar Niemeyer) [5] [72]
- Espèces: ” Mimivirus Battle6 ” [soixante-treize] [51]
- Espèces: ” Mimivirus Battle7 ” [74] [51]
- Espèces: ” Mimivirus Battle19 ” [75] [51]
- Espèces: ” Mimivirus Battle27 ” [76] [51]
- Espèces: ” Mimivirus Battle57 ” [77] [51]
- Espèces: ” Mimivirus Battle66 ” [78] [51]
- Espèces: ” Mimivirus Battle83 ” [79] [51]
- Espèces: ” Mimivirus Battle86 ” [80] [81]
- Espèces: ” Mimivirus Cher ” [82] [81]
- Espèces: ” Mimivirus dakar4 ” [83] [51]
- Espèces: Mimivirus fauteuil (Alias „ Fauteuil-Virus “) [84] [85] [5] [51] – voir. Fauteuil virus FD [quatre-vingt six]
- Espèces: ” Mimivirus huitre A06 ” [quatre-vingt sept] [51]
- Espèces: ” Mimivirus lactour «(Alias„ Imivirus lactours “, [51] Lactours-virus ) [88] [85] – voir. Virus lactours lt2 [89]
- Espèces: ” Mimivirus lentille «(Alias„ Lentille-Virus “,” Acanthamoeba polyphaga lentillevirus “) [90] [85] [51] [66] – voir. Lentille virus CL [91]
- Espèces: ” Mimivirus Longchamps «(Alias„ Longchamps-Virus “) [92] [85] [5] [51] – voir. Virus Longchamps FPL [93]
- Espèces: ” Mimivirus marais «(Alias„ Marais-virus “) [quatre-vingt-quatorze] [85] [51]
- Espèces: ” Mimivirus Montadette2 ” [95] [81]
- Espèces: ” Mimivirus pointerouge1 ” [96] – voir. Virus de Pointerouge 1 (alias Virus pointe-rouge 1 ) [97] [5] [51] – Emplacement: Marseille, France [5]
- Espèces: ” Mimivirus pointerouge2 ” [98] – voir. Virus de Pointerouge 2 (alias Virus Pointe-Rouge 2 ) [99] [5] [51] – Emplacement: Marseille, France [5]
- Espèces: Mimivirus SR1 (Alias „ Virus de type mimivirus SR1 “) [100] – Emplacement: Serendah Village, Peninsule malaise – cascade [65]
- Espèces: Mimivirus SR4 (Alias „ Virus de type mimivirus SR4 “) [101] – Emplacement: Serendah Village, péninsule malaise – Bascade et village moyens [65]
- Espèces: ” Mimivirus SR9 «(Alias„ Virus de type mimivirus SR9 “) [102] – Emplacement: Village de Serendah, péninsule malaise – Bouche de l’afflux dans le lac [65]
- Espèces: ” Mimivirus T2 ” [103] [51]
- Espèces: ” Mimivirus T3 ” [104] [51]
- Espèces: ” Mimivirus terra2 «(Alias Terra2-virus ) [105] [85] [51] – Emplacement: Marseille, France [5]
- Espèces: ” Mimivirus Univirus ” [106] [81]
Kladogramm [ Modifier | Modifier le texte source ]]
Arbre phylogénétique proposé du genre Mimivirus Nach Abrahão et al. (2018), Fig. 4: [107]
| Mimivirus |
|
||||||||||||||||||||||||||||||||||||||||||
Remarque: italique pour les espèces proposées.
- ↑ un b c d C’est F Le matériau a été copié par cette source, qui sous un Creative Commons Attribution 4.0 Licence internationale est disponible.
- Stefanie Reinberger: Révolution des virus géants . Dans: Spectre de la science. Spectrum of Science Verlagsgesellschaft, Heidelberg 2012, pp. 14-16.
- Graziele Oliveira, Bernard La Scola, Jonathan Abrahão: Virus géant vs amibe: combattre pour la suprématie . Dans: Virol J , 16, 126, 4. novembre 2019, Deux: 10.1186 / s12985-019-1244-3 , Researchgate.net (PDF)
- Ana Cláudia Dos S. P. Andrade, Thalita S. Arantes, Rodrigo A. L. Rodrigues, Talita B. Machado, Fábio P. Dornas, Melissa F. Landell, Cinthia Furst, Luiz G. A. Borges, Lara A. L. Dutra, Gabriel Almeida, Giliane De S. Iveira, Erna G. Kroon, Jonathan S. Abrahão: Géants omniprésents: une pléthore de virus géants trouvés au Brésil et en Antarctique . Dans: Revue virologique , Bande 15, nr. 22, 24 janvier 2018, doi: 10.1186 / s12985-018-0930-x
- Philippe Colson, Bernard La Scola, Anthony Levasseur, Gustavo Caetano-Anollés,& Didier Raoult: Mimivirus: ouvrir la voie à la découverte de virus géants d’amibes . Dans: La nature revue microbiologie , Band 15, pp. 243–254, 27 février 2017, doi: 10.1038 / nrmicro.2016.197
- Jean-Michel Clangie (Hrsg.): Photos du Mimivirus. Laboratoire d’information structurelle et génomique CNRS UPR, Marseille
- Séquence de référence NC_006450. NCBI
- David R. Wessner: Découverte du mimivirus géant . Masterclasses de la nature
- Mimivirus – un aperçu , AUF: ScienceDirect, 2012
- Scinexx: Énormes virus à la frontière à la vie , à partir du 28 février 2018
- Andreas Jahn: Le virus du virus , ON: Spektrum.de du 6 août 2008
- Page de biologie: Mimivirus
- À lui: Questions de taille. La vie est en direct. , ON: Biologie de la taille des octets, blog du 1er mai 2009, avec une image du Stargate qui s’ouvre, identique à Magicic et al. (2008), Fig. 5. [24]
- Rookie Adrian: Une nouvelle étude met en lumière les virus géants mystérieux . Phys.org, 8 mai 2020 (l’illustration montre apparemment un représentant typique du genre Mimivirus avec Stargate (ouverture pour l’ADN) et Tegument (couverture en fibrilles).
- Nicoletta Lanese: Les virus géants crachent leur ADN à travers un «Stargate». Maintenant, les scientifiques savent ce qui les déclenche . LiveScience, 26. Mai 2020
- ↑ un b Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: Le virus de Bodo Saltans (BSV) infectant la kinétoplastide, une fenêtre sur les virus géants les plus abondants de la mer… , dans: Elife Sciences 7, mars 2018, Deux: 10.7554 / Elife.33014
- ↑ un b c Centre national de la recherche scientifique: Liste des principaux virus «géants» connus sous le nom d’aujourd’hui (mars 2019) (PDF) Universally Aix Mieselee, mirs, 2019.
- ↑ un b Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Diversité cachée des virus géants du sol . Dans: Communications de la nature. Volume 9, numéro d’article: 4881 (2018), 19. novembre 2018, Deux: 10.1038 / S41467-018-07335-2
- ↑ un b c d C’est ICTV: Histoire de la taxonomie ICTV: Acanthamoeba Polyphaga Mimivirus , EC 51, Berlin, Allemagne, juillet 2019; Ratification par e-mail mars 2020 (MSL # 35)
- ↑ un b c d C’est F g H je J k l m n O p q r s t dans dans Jan Diesterend, Janis Kruse, Monica Hagedorn, Christian Hammann: Les amibes, les virus géants et les virophages constituent un trio complexe et multicouche , dans: Frontiers in Cellular and Infection Microbiology 7, janvier 2018, Deux: 10.3389 / fcimb.2017.00527 , via Researchgate , Fig. 1 (NCLDVS et ‘ Mégavirales «Ne sont pas appelés« familles »correctement dans ce travail, signifiaient« groupes »).
- ↑ un b c Viralzone: Mimivirus. Expasy, Récupéré le 8 juillet 2019 .
- ↑ ICTV: Liste des espèces maîtres 2018b.v2. Consulté le 6 août 2019 . MSL # 34V
- ↑ un b Berrnhaire Latez, S. Audil, C. Robert, L. Jamer, R. T. Basslerie, D. Goung: Un virus géant dans les amibes. Dans: Science. 299, 2003, S. 2033. PMID 12663918
- ↑ Laurie O’Keefe: Dimensionnement des virus . (PDF; 103 Ko) Le scientifique . Illustration à Didier Raoult: Virus reconsidérés , ibid du 28 février 2014
- ↑ un b c d Dedier Raoult, S. Audic, C. Robert, C. Abergel, P. Reneso, H. Ogata, B. La Scola, M. Suzan, J. M. Claverie: La séquence du génome de 1,2 mégabase du mimivirus. Dans: Science. 306 (5700), 19. novembre 2004, S. 1344–1350, doi: 10.1126 / science.1101485 . PMID 15486256
- ↑ Découverte et autres études sur les virus géants à l’infection IHU Mediterranee qui a modifié la perception de la virosphère . Dans: Virus , 11 (4), mars / avril 2019, PII: E312, doi: 10.3390 / v11040312 , PMC 6520786 (Texte intégral gratuit), PMID 30935049
- ↑ H. Pearson: “ Virophage ” suggère que les virus sont vivants . Dans: Nature . Groupe 454 , Non. 7205 , 2008, ISSN 0028-0836 , S. 677–677 , est ce que je: 10.1038 / 454677a , Bibcode: 2008Nature.454..677p .
- ↑ un b c d C’est M. Suzan-Monti, B. La Scola, D. Raink: Aspects génomiques et évolutifs de Mimivirus . Dans: Recherche de virus . Groupe 117 , Non. d’abord , Avril 2006, S. 145–155 , est ce que je: 10.1016 / j.virusres.2005.07.011 , PMID 16181700 .
- ↑ un b c d C’est F g H je J k Thomas Klose, Dominik A. Herbst, Hanyu Zhu, Joann P. Max, Hilkka I. Field, Michael G. Rossmann: Une enzyme de mimivirus qui participe à l’entrée virale , dans: Structure Band 23, Nr. 6, 2. juin 2015, S. 1058–1065, Deux: 10.1016 / j.str.2015.03.023
- ↑ un b c ICTV: Mimiviridae – Figures. Dans: ICTV 9th Report (2011).
- ↑ un b c d C’est F g H Xiao C., Kuznetsov Y. G., Sun S., Hafenstein S. L., Kostyuchenko V. A., Chipman P. R., Suzan-Monti M., Raoult D., McPherson A., Rossmann M. G.: Études structurelles du mimivirus géant . Dans: PLOS BIOL . Groupe 7 , Non. 4 , 2009, S. E92 , est ce que je: 10.1371 / journal.pbio.1000092 , PMID 19402750 , PMC 2671561 (Texte complet gratuit).
- ↑ Jean-Michel Claverie, Chantal Abergel, Hiroyuki Ogata: Mimivirus . Dans: Curr Top Microbiol Immunol . Groupe 328 , 2009, S. 89–121 , est ce que je: 10,1007 / 978-3-540-68618-7_3 , PMID 19216436 .
- ↑ un b c d Klose Thomas, Kuznetsov Y. G., Xiao C., Sun S., McPherson A., Rossmann M. G.: La structure tridimensionnelle du Mimivirus . Dans: Intervironnement . Groupe 53 , Non. 5 , 2010, S. 268-273 , est ce que je: 10.1159 / 000312911 , PMID 20551678 , PMC 2895761 (Texte complet gratuit).
- ↑ Voir aussi vanillyl alcool oxydase
- ↑ Chuan Xiao, Matthias G. Fischer, Sleep M. Bolotaulo, Nancy Ulloa-Randeau, Gustavo A. Avila & Curtis A. Suttle: La reconstruction cryo-em de la capside du virus de la cafétéria Roenbergensis suggère une nouvelle voie d’assemblage pour les virus géants . Dans: Rapports scientifiques de la nature , Bande 7, nr. 5484, 14 juillet 2017, Doi: 10.1038 / s41598-017-05824-W .
- ↑ James L. mais je ne le fais pas: Virus géants . Aud: Scientifique américain , Volume 89, n ° 4, juillet / août 2011. Voir en particulier la figure 3.
- ↑ En physique, ce principe géométrique est connu comme une symétrie (réfraction d’une symétrie discrète).
- ↑ un b c d C’est Autothan Nitan Zauterman, Y. Mussafia, D. B. Halli, E. Shimoni, E. Klein, C. Xio, S. Sunky, A. Minsky: Minsky: Portails de sortie et d’emballage d’ADN distincts dans le virus ACanthamoeba Polyphaga Mimivirus . Dans: PLOS BIOL . Groupe 6 , Non. 5 , 2008, S. E114 , est ce que je: 10.1371 / journal.pbio.0060114 , PMID 18479185 , PMC 2430901 (Texte complet gratuit).
- ↑ un b Disa Bäckström, Natalya Yutin, Stifen L. Jøgensen, Jennah Dharamshi, Felix Homa, Catzyna Zaremba-Niedwiedzka, Anja Spang, Yuri I. Wolf, Eugene V. Contise, Thijs J. G. G. G. G. G. G. G. Richard P. Novick (HRSG.): Les génomes du virus des sédiments en mer profonde élargissent le mégavirome de l’océan et soutiennent les origines indépendantes du gigantisme viral. Dans: MBIO Vol. 10, n ° 2, mars – avril 2019, S. E02497 – E18, Pdf ( Mémento des Originaux à partir du 29 juin 2019 Archives Internet ) Info: Le lien d’archive a été utilisé automatiquement et non encore vérifié. Veuillez vérifier le lien d’origine et d’archiver en fonction des instructions, puis supprimez cette note. (PDF) Doi: 10.1128 / race.02497-18 , PMC 6401483 (Texte complet gratuit). PMID 30837339 , Researchgate
- ↑ un b c d C’est Mickaël Boyer, Saïd Azza, Lina Barrassi, Thomas Klose, A. Campocasso, I. Pagnier, G. Fournous, A. Borg, C. Robert, Xhang, C. DeTtus, B. Henrissat, M. Rossat, D. Raoult: Mimivirus montre une réduction spectaculaire du génome après culture intraamoebal. Dans: Proc Natl Acad Sci (PNAS) USA 108 (25), 21. Juni 2011, S. 10296–10301, doi: 10.1073 / pnas.1101118108 . PMID 21646533 , PMC 3121840 (Texte complet gratuit).
- ↑ David M. Needham, Alexandra Z. Worden et al .: Une lignée distincte de virus géants apporte un photosystème de rhodopsine aux prédateurs marins unicellulaires , dans: PNAS, 23. septembre 2019, doi: 10.1073 / pnas.1907517116 , ISSN 0027-8424 , ici: Supplément 1 (xlsx)
- ↑ Lansing M. Prescott: Microbiologie . Wm. C. Brown Publishers, Dubuque, IA 1993, ISBN 0-697-01372-3.
- ↑ un b c d Jean-Michel Claverie, Chantal Abergel: Mimivirus et son virophage . Dans: Genet Genet de 6 ans . Groupe 43 , Non. 49–66 , 2009, doi: 10.1146 / Annurev-Genet-102108-134255 , PMID 19653859 .
- ↑ Jean-Michel Claverie, Hiroyuki Ogata, Stéphane Audic, Chantal Abergel, Pierre-Edouard Fournier, Karsten Suhre: Mimivirus et le concept émergent du virus «géant» . Dans: Recherche de virus . Groupe 117 , Non. d’abord , 2006, S. 133–144 , est ce que je: 10.1016 / j.virusres.2006.01.008 , PMID 16469402 , Arxiv: Q-BIO / 0506007 .
- ↑ Hiroyuki Ogata, Didier Raoult, Jean-Michel Claverie: Un nouvel exemple d’intein viral dans Mimivirus . Dans: Virol J . Groupe 2 , Non. 8 , 2005, PMID 15707490 , PMC 549080 (Texte complet gratuit).
- ↑ un b Musshi, Jabert n. Jaeman I. Replication cytoplasmique de type vaccina du mimivirus géant . Dans: Proc Natl Acad Sci USA (PNAS) . Groupe 107 , Non. 13 , 2010, S. 5978–5982 , est ce que je: 10.1073 / pnas.0912737107 , PMID 20231474 , PMC 2851855 (Texte complet gratuit).
- ↑ un b B. La Scola, T. J. Marrie, J. P. Auffray, D. Raoult: Mimivirus chez les patients atteints de pneumonie . Dans: Émerger infecte dis. 11e année, Non. 3 , 2005, S. 449–452 , PMID 15757563 , PMC 3298252 (Texte complet gratuit).
- ↑ Marrie TJ, Durant H, Yates L: Pneumonie acquise par la communauté nécessitant une hospitalisation: étude prospective à 5 ans . Dans: Examens des maladies infectieuses . 11e année, Non. 4 , 1989, S. 586–99 , est ce que je: 10.1093 / CLINIDS / 11.4.586 , PMID 2772465 .
- ↑ Hanene Saadi, Isabelle Pagnier, Philippe Colson, Jouda Kanoun Cherif, Majed Beji, Mondheer Bugalmi, Saïd Azza, Nicholas Armstrong, Catherine Robert, Ghlain Funnoous, Bernard La Scola, Diier Raoult: Première isolement du mimivirus chez un patient atteint de pneumonie . Dans: Maladies infectieuses cliniques . 57e année, Non. 4 , Août 2013, S. E127-34 , est ce que je: 10.1093 / CA / CIT354 , PMID 23709652 .
- ↑ un b E. Ghigo, J. Kartenbeck, P. Lien, L. Pelkmans, C. Capo, J. L. Mense, D. Raoult: Le pathogène ameobal Mimivirus infecte les macrophages par phagocytose. Dans: Pathogènes PLOS. Bande 4, numéro 6, juin 2008, S. E1000087, Doi: 10.1371 / journal.ppat.1000087 , PMID 18551172 , PMC 2398789 (Texte complet gratuit).
- ↑ Vamin A.Cre Iovo B., Pipazian L. Progrès de la pathogénicité de l’immivirus . Dans: Intervironnement . Groupe 53 , Non. 5 , 2010, S. 304–309 , est ce que je: 10.1159 / 000312915 , PMID 20551682 .
- ↑ P. Berger, L. Papazian, M. Drancourt, B. La Scola, J. P. Auffray, D. Raoult: Micro-organismes associés à l’AMEBA et diagnostic de pneumonie nosocomiale . Dans: Émerger infecter dis . 12e année, Non. 2 , 2006, S. 248–255 , PMID 16494750 , PMC 3373093 (Texte complet gratuit).
- ↑ D. Raoult, P. Reneso, P. Brouqui: Infection en laboratoire d’un technicien par Mimivirus . Dans: Ann interne avec . 144e année, Non. 9 , 2006, S. 702–703 , PMID 16670147 ( Annals.org ).
- ↑ M. J. Vanspauwen et al .: Infections par Mimivirus chez les patients atteints de maladie pulmonaire obstructive chronique . Dans: Médecine respiratoire . 106e année, Non. douzième , 2012, S. 1690-1694 , est ce que je: 10.1016 / j.rmed.2012.08.019 ( ScienceDirect.com ).
- ↑ Anthony Lephera, Winzigurs, Ern Cabrageby, Pierre Post Porteciti, Berier Loodti, date de Rooel. Mimivire est un système de défense dans Mimivirus qui confère une résistance au virophage. Dans: Nature , 2016, doi: 10.1038 / nature17146 .
- ↑ Ewen Callaway: Système «immunitaire» de type CRISPR découvert dans le virus géant , dans: Nature: News, du 29 juin 2016
- ↑ Nisrine Chelkha, Anthony Levasseur, Pierre Pontarotti, Didier Raoult et al.: Une étude phylogénomique des séquences du génome de la brouillon Acanthamoeba Polyphaga suggère des échanges génétiques avec des virus géants , dans: Frontiers in Microbiology 9: 2098, septembre 2018, Deux: 10.3389 / FMICB.2018.02098
- ↑ Patrick L. Scheid: Les amibes libres et leurs impacts multiples sur la santé environnementale , dans: Module de référence dans les systèmes de terre et les sciences de l’environnement, 27. Février 2018, doi: 10.1016 / b978-0-12-409548-9.10969-8 , ici: Texte après la figure 8 (colonne de droite)
- ↑ Une homologie entre CRISPR et Mimivire ne semble pas impossible, mais nécessite certainement des examens supplémentaires.
- ↑ M. G. Fischer, C. A. Suttle: Un virophage à l’origine de grands transposons d’ADN . Dans: Science . Groupe 332 , Non. 6026 , 2011, S. 231–234 , est ce que je: 10.1126 / science.1199412 , PMID 21385722 .
- ↑ NCBI: Mimivirus AB-566-O17 (acronyme: GVSAG AB-566-O17) (Espèces)
- ↑ William H Wilson, Ilana C Gilg, Mohammad Moniruzzaman, Erin K Field, Sergey Koren, Gary R LeCleir, Joaquín Martínez Martínez, Nicole J Poulton, Brandon K Swan, Steven Wilhelm: Exploration génomique des virus géants de l’océan individuels. Dans: ISM Journal. 11 (8), août 2017, S. 1736–1745, Deux: 10.1038 / Ismej.2017.61 , PMC 5520044 (Texte complet gratuit). PMID 28498373
- ↑ un b c NCBI: Acanthamoeba Polyphaga Mimivirus (Espèces)
- ↑ Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Deia Sahmi, a déclaré Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Fabio Dornas, Jacques Yaacoub Boub Khalna, Islelele, Jacques Yaacoub Ier, Anthony, Anthony Levassur, Philippe Colson, Jonathan Abrahão, Bernard La Scola: Découverte et autres études sur les virus géants à l’infection IHU Mediterranee qui a modifié la perception de la virosphère . Dans: Virus , 11 (4), mars / avril 2019, PII: E312, doi: 10.3390 / v11040312 , PMC 6520786 (Texte intégral gratuit), PMID 30935049
- ↑ un b c d C’est F g H je J k l m n O p q r s t dans dans Didier Raoult, Anthony Levasseur, Bernard La Scola: Détection de PCR de Mimivirus, dans: Emerging Infectious Diseases, Juni 2017, Band 23, Nr. 6, S. 1044–1045, Doi: 10.3201 / eid2306.161896 , Pdf (PDF)
- ↑ Haansika chhabra: Virus géants trouvés dans les échantillons d’eau de Mumbai , dans: Businessline: Science, Bangalore, 9. Mai 2019
- ↑ un b c d Sans départements: Une famille de nouvelles cyclophilines, conservées dans le genre Mimivirus des virus ADN géants , dans: Computational and Structural Biotechnology Journal, Band 16, Juli 2018, S. 231–236, doi:10.1016/j.csbj.2018.07.001
- ↑ Anirvan Chatterjee, Thyas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Les signatures géantes et métagénomiques des virus géants sont omniprésentes dans les échantillons d’eau provenant des eaux usées, du lac intérieur, de l’usine de traitement des eaux usées et de l’approvisionnement municipal en eau à Mumbai, en Inde . Dans: Rapports scientifiques , Volume 9, n ° 3690, 6 mars 2019, Doi: 10.1038 / s41598-019-40171-y , PMID 30842490 , PMC 6403294 (Texte intégral gratuit)
- ↑ un b Masaharu Takemura, Tatsuya Mikami, Shingo Murono: Presque concourir la séquence du génome de deux Mimivirus Des souches isolées d’un étang d’eau douce japonaise et de la bouche de la rivière, dans: génome annonce. 4 (6), novembre / décembre 2016, E01378-16, Deux: 10.1128 / Genomea.01378-16 , PMC 5146454 (Texte complet gratuit). PMID 27932662 . Les deux lignes virales sont étroitement liées aux auteurs avec Mimivirus Bombay, ils appartiennent donc également aux Spezies APMV.
- ↑ Rafael K Campos, Paulo V Boratto, Felipe L Assis, Eric Rgr Aguiar, Lorena CF Silva, Jonas D Albarnaz, Fabio P Dornas, Giliane S Trindade, Paulo P Ferreira, João T Marques, Catherine Robert, Division Raoult, Erna Gl Virus de samba: un nouveau mimivirus d’une forêt tropicale géante, l’Amazonie brésilienne. Dans: Virology Journal 2014 11:95, Deux: 10.1186 / 1743-422x-11-95
- ↑ Jason R. Schrad, Jonathan S. Abrahão, Juliana R. Cortines et Kristin N. Parent: Caractérisation structurelle et protéomique de l’initiation d’une infection du virus géant (PDF) dans: cellule VOM 8. Mai 2020, doi: 10.1016 / j.cell.2020.04.032
- ↑ Jason R. Schrad, Jonathan S. Abrahão, Juliana R. Cortines, Kristin N. Parent: L’acide bouillant imite la libération du géant géant intracellulaire du géant (PDF) sur: Biorxiv du 20 septembre 2019, doi: 10.1101 / 777854 (Préimprimée)
- ↑ Virus géants mystérieux: en taille et en complexité gargantuesennes , ON: ScitechDaily à partir du 9 mai 2020, source: Michigan State University
- ↑ un b c Felipe L. Assis, Leena Bajrai, Jonathan S. Abraham, Erna G. Kroon, Fabio P. Dornas, Kétylen R. Andrade, Paulo V. B. Boratto, Mariana R. Pilotto, Catherine, Bernard The Penlope. Analyse du panénome de la lignée brésilienne A Mimivirus d’amie , dans: Virus 7 (7), 2015, S. 3483–3499, Doi: 10.3390 / v7072782 . Les candidats traités ici représentent un groupe de parenté selon les auteurs, selon lesquels les autres sources montrent que le virus de la samba et du kroon appartient aux espèces APMV. Cela s’applique alors aux deux autres.
- ↑ Paulo Victor Miranda Boratto, Fábio Pio Dornas, Lorena Christine Ferreira da Silva, Rodrigo Araújo Lima Rodrigues, Graziele Pereira Oliveira, Juliana Reis Cortines, Betânia Paiva Drumond, Jonatas Santos Abrahão: Les analyses du gène de capside majeur du virus Kroon et de sa transcription mettent en évidence un schéma distinct d’évolution des gènes et d’épissage parmi les mimivirus. Dans: J Virol. 92 (2), 15. janvier 2018, E01782-17, Doi: 10.1128 / jvi.01782-17 , PMC 5752926 (Texte complet gratuit). PMID 29118120
- ↑ NCBI: Moumouvirus saoudien
- ↑ Leena H. Bajrai, Felipe L. de Assis, Esam I. Azhar, Priscilla Jardot, Catherine Robert, Jonathan Abrahão, Didier Raoult, Bernard La Scola: Saudi Moumouvirus, le premier groupe B Mimivirus isolé de l’Asie , devant. Microbiol., 20 décembre 2016, Deux: 10.3389 / FMICB.2016.02029
- ↑ NCBI: Acanthamoeba Castellanii Mamavirus (Espèces)
- ↑ un b c d Tan Yeh Fong, Chai Ying Lim, Chun Wie Chong, Patricia Kim Chooi Lim, Ivan K. S. Yap, Pooi Pooi Leong, Kenny Voon: Isolement et quantification des virus de type mimivirus et de type Marseillevirus à partir d’échantillons de sol dans un village aborigène (Orang Asli) en Malaisie péninsulaire , dans: Interirology 61 (2), S. 1–4, août 2018, doi: 10.1159 / 000491602 , Medscape , Pdf (PDF; 523 kb) Fig. 2 – Sr1, Sr4 et Sr9 sont étroitement liés les uns aux autres, mais sont beaucoup plus proches des autres candidats à la ligne A que cela Moumouvirus saoudien (Ligne B). L’attribution à la ligne A est donc probable.
- ↑ un b Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: pnas.org , dans: PNAS 116 (39), 10./24. Septembre 2019, S. 19585 –19592, doi: 10.1073 / pnas.1912006116 . PMID 31506349 , Fig. 2 . En outre:
Julien Guglielmini, Anthony Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: La diversification des virus géants et de l’ADNdb eucaryote a précédé l’origine des eucaryotes modernes , ON: Biorxiv du 29 octobre 2018 (préparation), doi: 10.1101 / 455816 - ↑ NCBI: Herudovirus Strain Sangsue
- ↑ Mondher Boughalmi, Isabelle Pagnier, Sarah Aherfi, Philippe Colson, Didier Raoult, Bernard La Scola: Première isolement d’un virus géant de sauvage Hirud médicalisé Sangsue: Mimiviridae isolement dans Hirud médicalisé (PDF) Dans: Virus 2013, 5, S. 2920-2930, doi: 10.3390 / v5122920
- ↑ un b Paulo V. M. Boratto, Thalita S. Arantes, Lorena C. F. Silva, Felipe L. Assis, Erna G. Kroon, Bernard La Scola, Jonathan S. Abrahão: Virus Niemeyer: un nouveau mimivirus groupe A isolat hébergeant un ensemble de gènes aminoacyl-ARNtas dupliqués. Dans: Microbiol avant. Bande 6, 2015, S. 1256. Deux: 10.3389 / FMICB.2015.01256 , PMC 4639698 (Texte complet gratuit). PMID 26635738 .
- ↑ un b Gabriel Augusto Pires de Souza, Victoria Fulgencio Queiroz, Maurício Teixeira Lima, Erik Vinicius de Sousa Reis, Luiz Felipe Leomil Coelho, Jonatas Santos Abrahão: Le virus devient viral: un kit éducatif pour les cours de virologie. Dans: Revue virologique , Bande 17, nr. 13, 31 janvier 2020, Deux: 10.1186 / s12985-020-1291-9
- ↑ NCBI: Virus Niemeyer (Espèces)
- ↑ Elton Alisson: Nouveau virus géant trouvé au Brésil , Bei: Agence Fapesp, Brasilien, 4. Juni 2014
- ↑ NCBI: Mimivirus Battle6 (Espèces)
- ↑ NCBI: Mimivirus Battle7 (Espèces)
- ↑ NCBI: Mimivirus Battle19 (Espèces)
- ↑ NCBI: Mimivirus Battle27 (Espèces)
- ↑ NCBI: Mimivirus Battle57 (Espèces)
- ↑ NCBI: Mimivirus Battle66 (Espèces)
- ↑ NCBI: Mimivirus Battle83 (Espèces)
- ↑ NCBI: Mimivirus Battle86 (Espèces)
- ↑ un b c d C’est Anirvan Chatterjee, Thyas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Isolement et séquençage complet du génome de Mimivirus Bombay, un virus géant dans les eaux usées de Mumbai, Inde . Dans: Données génomiques. 9 c), mai 2016, doi:10.1016/j.gdata.2016.05.013 , Fig. 2
- ↑ NCBI: Mimivirus Cher (Espèces)
- ↑ NCBI: Mimivirus dakar4 (Espèces)
- ↑ NCBI: Mimivirus fauteuil
- ↑ un b c d C’est F Christelle Desnues, Bernard La Scola, Natalya Yutin, Ghislain Fournous et al.: Provirophages and transpovirons as the diverse mobilome of giant viruses, in: PNAS 109(44), 30. Oktober 2012, S. 18078–18083, doi: 10.1073 / pnas.1208835109
- ↑ NCBI: Fauteuil virus FD
- ↑ NCBI: Mimivirus huitre A06 (Espèces)
- ↑ NCBI: Mimivirus lactour (Espèces)
- ↑ NCBI: Virus lactours lt2 (Espèces)
- ↑ NCBI: Mimivirus lentille (Espèces)
- ↑ NCBI: Lentille virus CL (Espèces)
- ↑ NCBI: Mimivirus Longchamps (Espèces)
- ↑ NCBI: Virus Longchamps FPL (Espèces)
- ↑ NCBI: Mimivirus marais (Espèces)
- ↑ NCBI: Mimivirus Montadette2 (Espèces)
- ↑ NCBI: Mimivirus pointerouge1 (Espèces)
- ↑ NCBI: Virus de Pointerouge 1 (Espèces)
- ↑ NCBI: Mimivirus pointerouge1 (Espèces)
- ↑ NCBI: Virus de Pointerouge 1 (Espèces)
- ↑ NCBI: Mimivirus SR1 (Espèces)
- ↑ NCBI: Mimivirus SR4 (Espèces)
- ↑ NCBI: Mimivirus SR9 (Espèces)
- ↑ NCBI: Mimivirus T2 (Espèces)
- ↑ NCBI: Mimivirus T3 (Espèces)
- ↑ NCBI: Mimivirus terra2 (Espèces)
- ↑ NCBI: Mimivirus Univirus (Espèces)
- ↑ Jonathan Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assisi, Paulo Boratto Colson, Anthony Levisur, Guido Kroemer, Guido Kroemer Raoult, Bernard La Scola: Le tupanvirus géant à queue possède l’appareil de traduction le plus complet de la Virosphère connue . Dans: Communications de la nature . 9e année, Non. d’abord , 27. février 2018, deux: 10.1038 / s41467-018-03168-1 ( Nature.com ).
Recent Comments