Thermomètre ARN – Wikipedia wiki
Structure d’ARN dépendante de la température
Un Thermomètre ARN (ou Thermocapteur ARN ) est une molécule d’ARN non codante sensible à la température qui régule l’expression des gènes. Les thermomètres d’ARN régulent souvent les gènes requis lors d’un choc thermique ou d’une réponse au choc froid, mais ont été impliqués dans d’autres rôles de régulation tels que dans la pathogénicité et la famine. [d’abord]
En général, les thermomètres d’ARN fonctionnent en modifiant leur structure secondaire en réponse aux fluctuations de température. Cette transition structurelle peut ensuite exposer ou occlure des régions importantes d’ARN telles qu’un site de liaison des ribosomes, qui affecte ensuite le taux de traduction d’un gène de codage protéique à proximité.
Les thermomètres d’ARN, ainsi que les riboswitches, sont utilisés comme exemples à l’appui de l’hypothèse du monde de l’ARN. Cette théorie propose que l’ARN était autrefois le seul acide nucléique présent dans les cellules et a été remplacé par le système actuel de l’ADN → ARN → protéine. [2]
Des exemples de thermomètres d’ARN comprennent FourU, [3] le HSP90 cis -ELLEME DE RÉGULATION, [4] l’élément rose, [5] Le thermomètre ARN LIG, [6] et le thermomètre HSP17. [7]
Découverte [ modifier ]]
Le premier élément d’ARN sensible à la température a été signalé en 1989. [8] Avant cette recherche, les mutations en amont du site de début de transcription dans un phage CIII de phage Lambda (λ) se sont révélées affecter le niveau de traduction de la protéine CIII. [9] Cette protéine est impliquée dans la sélection d’un cycle de vie lytique ou lysogène dans le phage λ, avec des concentrations élevées de CIII favorisant la lysogénie. [9] Une étude plus approfondie de cette région d’ARN en amont a identifié deux structures secondaires alternatives; L’étude expérimentale a révélé que les structures étaient interchangeables et dépendent à la fois de la concentration et de la température des ions de magnésium. [8] [dix] Ce thermomètre ARN est maintenant censé encourager l’entrée à un cycle lytique sous contrainte thermique afin que le bactériophage se réplique rapidement et échappe à la cellule hôte. [d’abord]
Le terme “thermomètre ARN” n’a été inventé qu’en 1999, [11] Quand il a été appliqué à l’élément ARN RPOH identifié dans Escherichia coli . [douzième] Plus récemment, des recherches en bioinformatique ont été utilisées pour découvrir plusieurs nouveaux thermomètres d’ARN candidats. [13] Les recherches basées sur la séquence traditionnelle sont cependant inefficaces, car la structure secondaire de l’élément est beaucoup plus conservée que la séquence d’acide nucléique. [13]
Distribution [ modifier ]]
La plupart des thermomètres d’ARN connus sont situés dans la région non traduite (UTR) 5 dans les données génomiques. [14] [15]
Bien que principalement trouvé chez les procaryotes, un thermomètre ARN potentiel a été trouvé chez les mammifères, y compris les humains. [16] L’ARN-1 du choc thermique du thermocapteur candidat (HSR1) active le facteur de transcription du choc thermique 1 (HSF1) et induit des protéines protectrices lorsque la température cellulaire dépasse 37 ° C (température corporelle), empêchant ainsi les cellules de surchauffer. [16]
Structure [ modifier ]]

Les thermomètres d’ARN sont structurellement simples et peuvent être fabriqués à partir de séquences d’ARN courtes; Le plus petit n’est que de 44 nucléotides et se trouve dans l’ARNm d’une protéine de choc thermique, Hsp17, en Synechocystis Espèce PCC 6803. [18] [19] Généralement, ces éléments d’ARN varient de 60 à 110 nucléotides [20] et ils contiennent généralement une épingle à cheveux avec un petit nombre de paires de bases incompatibles qui réduisent la stabilité de la structure, permettant ainsi un dépliage plus facile en réponse à une augmentation de la température. [21]
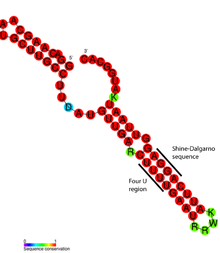
L’analyse structurelle détaillée du thermomètre ARN rose a révélé que les bases incompatibles sont en fait engagées dans des épanouissements non standard qui préserve la structure hélicoïdale de l’ARN (voir figure). Les paires de bases inhabituelles sont constituées de paires G-G, U-U et UC-U. Étant donné que ces paires de bases non canoniques sont relativement instables, l’augmentation de la température provoque la fusion locale de la structure de l’ARN dans cette région, exposant la séquence Shine-Dalgarno. [17]
Certains thermomètres d’ARN sont significativement plus complexes qu’une seule épingle à cheveux, comme dans le cas d’une région trouvée dans l’ARNm de l’ACSP qui contiendait un pseudoknot, ainsi que de multiples épingles à cheveux. [22] [23]
Les thermomètres à ARN synthétiques ont été conçus avec une simple structure en épingle à cheveux simple. [24] Cependant, la structure secondaire de ces thermomètres d’ARN courts peut être sensible à la mutation, car un seul changement de base peut rendre l’épingle à cheveux inactive en vain . [25]
Mécanisme [ modifier ]]

Les thermomètres d’ARN se trouvent dans le 5 ‘UTR de l’ARN messager, en amont d’un gène codant pour les protéines. [d’abord] Ici, ils sont capables d’obtenir le site de liaison des ribosomes (RBS) et d’empêcher la traduction de l’ARNm en protéine. [14] À mesure que la température augmente, la structure en épingle à cheveux peut «faire fondre» et exposer la séquence RBS ou Shine-Dalgarno pour permettre la liaison de la petite sous-unité ribosomale (30S), qui assemble ensuite d’autres machines de traduction. [d’abord] Le codon de démarrage, a généralement trouvé 8 nucléotides en aval de la séquence Shine-Dalgarno, [14] signale le début d’un gène codant pour les protéines qui est ensuite traduit par un produit peptidique par le ribosome. De plus cis -Actrage mécanisme, un seul exemple de trans -Actrage du thermomètre ARN a été trouvé dans l’ARNm de RPOS où il est considéré comme impliqué dans la réponse de famine. [d’abord]
Un exemple spécifique d’un motif de thermomètre ARN est le thermomètre Fouru trouvé dans Salmonella enterica . [3] Lorsqu’elle est exposée à des températures supérieures à 45 ° C, la boucle de tige qui se pose en face de la séquence Shine-Dalgarno devient non appariée et permet à l’ARNm d’entrer le ribosome pour que la traduction se produise. [25] Mg 2+ Il a également été démontré que la concentration en ions affecte la stabilité de FourU. [26] Le thermomètre ARN le plus étudié se trouve dans le rpoh gène dans Escherichia coli . [27] Ce thermocapteur régule positivement les protéines de choc thermique à des températures élevées à travers σ 32 , un facteur sigma spécialisé du choc thermique. [11]
Bien que généralement associés à l’expression des protéines induites par la chaleur, les thermomètres d’ARN peuvent également réguler les protéines du choc froid. [22] Par exemple, l’expression de deux protéines 7kda est régulée par un thermomètre ARN dans la bactérie thermophile Thermus thermophilus [28] et un mécanisme similaire a été identifié dans les entérobactéries. [23]
Les thermomètres d’ARN sensibles à des températures de 37 ° C peuvent être utilisés par des agents pathogènes pour activer les gènes spécifiques à l’infection. [14] Par exemple, la régulation positive de test , codant pour un régulateur transcriptionnel clé des gènes de virulence dans Listeria monocytogenes , a été démontré en fusionnant l’ADN 5 ‘de test au gène de la protéine fluorescente verte; La fusion de gènes a ensuite été transcrite à partir du promoteur T7 dans E. coli et la fluorescence a été observée à 37 ° C mais pas à 30 ° C. [29]
Implications pour l’hypothèse du monde de l’ARN [ modifier ]]
L’hypothèse du monde de l’ARN indique que l’ARN était autrefois le porteur des informations héréditaires et enzymatiquement actifs, avec différentes séquences agissant comme biocatalystes, régulateurs et capteurs. [30] L’hypothèse propose ensuite que l’ADN moderne, l’ARN et la vie à base de protéines ont évolué et que la sélection ait remplacé la majorité des rôles de l’ARN par d’autres biomolécules. [2]
On pense que les thermomètres d’ARN et les riboswitches sont évolutivement anciens en raison de leur distribution à grande échelle dans les organismes liés à la distance. [trente et un] Il a été proposé que, dans le monde de l’ARN, les thermocapteurs d’ARN auraient été responsables de la régulation dépendante de la température des autres molécules d’ARN. [2] [32] Les thermomètres d’ARN dans les organismes modernes peuvent être des fossiles moléculaires qui pourraient faire allusion à une importance auparavant plus répandue dans un monde d’ARN. [2]
Autres exemples [ modifier ]]
Les références [ modifier ]]
- ^ un b c d C’est F Narberhaus F, Waldminghaus T, Chowdhury S (janvier 2006). “Thermomètres d’ARN” . FEMS Microbiol. Tour . 30 (1): 3–16. est ce que je: 10.1111 / j.1574-6976.2005.004.x . PMID 16438677 .
- ^ un b c d Atkins JF, Metherand RF, Cach T (2006). Le monde de l’ARN: la nature de l’ARN moderne suggère un monde d’ARN prébiotique . Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-739-6 .
- ^ un b Waldminghaus T, Heidrich N, Brantl S, Narberhaus F (juillet 2007). “Fouru: un nouveau type de thermomètre ARN dans Salmonelle ” . Mol. Microbiol . 65 (2): 413–424. est ce que je: 10.1111 / j.1365-2958.2007.05794.x . PMID 17630972 .
- ^ un b Ahmed R, Duncan RF (2004). “Régulation translationnelle de l’ARNm de Hsp90. Éléments de région non traduits à 5 ‘AUG-proximal 5’s essentiels pour la traduction préférentielle des chocs thermiques” . J Biol Chem . 279 (48): 49919–49930. est ce que je: 10.1074 / jbc.m404681200 . PMID 15347681 .
- ^ un b Nocker A, Hausherr T, Balsiger S, Krstulovic NP, Hennecke H, Sarberhaus F (2001). “Un thermocapteur à base d’ARNm contrôle l’expression des gènes de choc thermique rhizobial” . Acides nucléiques res . 29 (23): 4800–4807. est ce que je: 10.1093 / nar / 29.23.4800 . PMC 96696 . PMID 11726689 .
- ^ Matsunaga J, Schlax PJ, Haake DA (2013-11-15). “Rôle pour les séquences d’ARN à action cis dans l’expression dépendante de la température des protéines LIG multiadhésives chez Leptospira interrogans” . Journal of Bactériologie . 195 (22): 5092–5101. est ce que je: 10.1128 / jb.00663-13 . ISSN 0021-9193 . PMC 3811586 . PMID 24013626 .
- ^ Kortman J, Sczodrok S, Rinnuthal J, Sloves H, Narb House F (2011). “Traduction à la demande par un simple thermocapteur à base d’ARN” . Acides nucléiques res . 39 (7): 2855–2868. est ce que je: 10.1093 / nar / gkq1252 . PMC 3074152 . PMID 21131278 .
- ^ un b Altuvia S, Kornitzer D, Teff D, Oppenheim AB (1989-11-20). “Les structures d’ARNm alternatives du gène CIII du bactériophage lambda déterminent le taux de son initiation de traduction”. Journal of Molecular Biology . 210 (2): 265-280. est ce que je: 10.1016 / 0022-2836 (89) 90329-X . PMID 2532257 .
- ^ un b Altuvia S, Oppenheim AB (juillet 1986). “Signaux régulateurs de translation dans la région codante du gène bactériophage lambda ciii” . Journal of Bactériologie . 167 (1): 415–419. est ce que je: 10.1128 / jb.167.1.415-419.1986 . PMC 212897 . PMID 2941413 .
- ^ Altuvia S, Corner D, Kobi S, Oppenheim AB (1991-04-20). “Éléments fonctionnels et structurels de l’ARNm du gène CII du bactériophage lambda” Journal of Molecular Biology . 218 (4): 723–733. est ce que je: 10.1016 / 0022-2836 (91) 90261-4 . PMID 1827163 .
- ^ un b Storz G (1999-03-15). “Un thermomètre ARN” . Gènes et développement . 13 (6): 633–636. est ce que je: 10.1101 / gad.13.6.633 . PMID 10090718 .
- ^ Morita MT, Tanaka Y, Kodama TS, Kyogoku Y, Yanagi H, Yura T (1999-03-15). “Induction translationnelle du facteur de transcription des chocs thermiques Sigma32: preuve d’un thermocapteur d’ARN intégré” . Gènes et développement . 13 (6): 655–665. est ce que je: 10.1101 / gad.13.6.655 . PMC 316556 . PMID 10090722 .
- ^ un b Waldminghaus T, Gaubig LC, Narberhaus F (novembre 2007). “Prédiction bioinformatique à l’échelle du génome et évaluation expérimentale des thermomètres potentiels d’ARN”. Génétique et génomique moléculaire . 278 (5): 555–564. est ce que je: 10.1007 / S00438-007-0272-7 . PMID 17647020 . S2cid 24747327 .
- ^ un b c d Narber House F (2010). “Contrôle translationnel des gènes bactériens de choc thermique et de virulence par des ARNm de détection de température” . ARN Biol . 7 (1): 84–89. est ce que je: 10.4161 / RNA.7.1.10501 . PMID 20009504 . Récupéré 2011-04-23 .
- ^ Johansson J (2009). “Thermocapteurs d’ARN dans les agents pathogènes bactériens”. Détection et signalisation bactériennes . Microbiol contribue . Contributions à la microbiologie. Vol. 16. Bâle. pp. 150–160. est ce que je: 10.1159 / 000219378 . ISBN 978-3-8055-9132-4 . PMID 19494584 .
- ^ un b Shamovsky I, Ivannikov M, Kandel ES, Gershon D, Nudler E (mars 2006). “Réponse médiée par l’ARN au choc thermique dans les cellules mammiliennes” Nature . 440 (7083): 556–560. Bibcode: 2006natur.440..556S . est ce que je: 10.1038 / nature04518 . PMID 16554823 . S2cid 4311262 .
- ^ un b Chowdhury S, Maris C, Allain FH, Narberhaus F (2006-06-07). “Base moléculaire pour la détection de température par un thermomètre ARN” . Le journal EMBO . 25 (11): 2487–2497. est ce que je: 10.1038 / sj.emboj.7601128 . PMC 1478195 . PMID 16710302 .
- ^ Kortmann J, Sczodrok S, Rinnanthal J, Schwalbe H, Narberhaus F (avril 2011). “Traduction à la demande par un simple thermocapteur à base d’ARN” . Recherche des acides nucléiques . 39 (7): 2855–2868. est ce que je: 10.1093 / nar / gkq1252 . PMC 3074152 . PMID 21131278 .
- ^ Kortmann J, Sczodrok S, Rinnanthal J, Schwalbe H, Narberhaus F (avril 2011). “Traduction à la demande par un simple thermocapteur à base d’ARN” . Acides nucléiques res . 39 (7): 2855–2868. est ce que je: 10.1093 / nar / gkq1252 . PMC 3074152 . PMID 21131278 .
- ^ Waldminghaus T, Fippinger A, Alfsmann J, Narberhaus F (décembre 2005). “Les thermomètres d’ARN sont communs dans l’alpha- et gamma-protéobactéries”. Biol. Chem . 386 (12): 1279–1286. est ce que je: 10.1515 / bc.2005.145 . PMID 16336122 . S2cid 84557068 .
- ^ Narberhaus F (janvier-février 2010). “Contrôle translationnel des gènes bactériens de choc thermique et de virulence par des ARNm de détection de température” . Biologie de l’ARN . 7 (1): 84–89. est ce que je: 10.4161 / RNA.7.1.10501 . PMID 20009504 .
- ^ un b Breaker RR (janvier 2010). “L’ARN passe dans le froid” . Mol. Cellule . 37 (1): 1–2. est ce que je: 10.1016 / j.molcel.2009.12.032 . PMC 5315359 . PMID 20129048 .
- ^ un b Giuladori AM, Di Pietro F, Marzi S, Masquida B, Wagner R, Romby P, Gualeerzi CO, Pon CL (Janogy 2010). “L’ARNm CSPA est un thermocapteur qui module la traduction de la protéine du choc froid CSPA” . Mol. Cellule . 37 (1): 21–33. est ce que je: 10.1016 / j.molcel.2009.11.033 . PMID 20129052 .
- ^ Nepert J, Kales D, Bock R (novembre 2008). “Conception de thermomètres d’ARN synthétiques simples pour l’expression des gènes contrôlés par température dans Escherichia coli” . Recherche des acides nucléiques . 36 (19): E124. est ce que je: 10.1093 / nar / gkn545 . PMC 2577334 . PMID 18753148 .
- ^ un b Nikolova EN, Al-Hashimi HM (septembre 2010). “Thermodynamique de la fusion de l’ARN, une paire de bases à la fois” . ARN . 16 (9): 1687–1691. est ce que je: 10.1261 / RNA.2235010 . PMC 2924531 . PMID 20660079 .
- ^ Renthal J, Klainte B, Narbauser F, Flumble H (2011-07-04). “Modulation de la stabilité du Salmonelle Thermomètre ARN de type Fouru ” . Recherche des acides nucléiques . 39 (18): 8258–8270. est ce que je: 10.1093 / nar / gkr314 . PMC 3185406 . PMID 21727085 .
- ^ Shah P, Gilchrist MA (2010). Spirin As (éd.). “La propriété thermoussiste des thermomètres d’ARN est-elle unique?” . Plos un . 5 (7): E11308. Bibcode: 2010ploso … 511308S . est ce que je: 10.1371 / journal.pone.0011308 . PMC 2896394 . PMID 20625392 .
- ^ Mega R, Manzoku M, Shinkai A, Nakagawa N, Kuramitsu S, Masui R (août 2010). Thermus thermophilus “. Biochem. Biophys. Res. Commun . 399 (3): 336–340. est ce que je: 10.1016 / j.bbrc.2010.07.065 . PMID 20655297 .
- ^ Johansson J, Mandin P, Renzoni A, Chiaruttini C, Springer M, Cossart P (septembre 2002). “Un thermocapteur d’ARN contrôle l’expression des gènes de virulence dans Listeria monocytogenes ” . Cellule . 110 (5): 551–561. est ce que je: 10.1016 / s0092-8674 (02) 00905-4 . PMID 12230973 .
- ^ Gilbert W (février 1986). “Le monde de l’ARN” . Nature . 319 (6055): 618. Bibcode: 1986natur.319..618G . est ce que je: 10.1038 / 319618A0 .
- ^ Serganov A, Patel DJ (octobre 2007). “Ribozymes, riboswitches et au-delà: régulation de l’expression des gènes sans protéines” . La nature revoit la génétique . 8 (10): 776–790. est ce que je: 10.1038 / nrg2172 . PMC 4689321 . PMID 17846637 .
- ^ Bocobza SE, Aharoni A (octobre 2008). “Changer la lumière sur les riboswitches de l’usine”. Tendances de la science des plantes . 13 (10): 526–533. est ce que je: 10.1016 / j.tplants.2008.07.004 . PMID 18778966 .
- ^ Gaubig LC, Waldminghaus T, Narberhaus F (janvier 2011). “Plusieurs couches de contrôle régissent l’expression de l’opéron de choc thermique d’Escherichia coli ibpab” . Microbiologie . 157 (Pt 1): 66–76. deux: 10.1099 / mic.0.043802-0 . PMID 20864473 .
- ^ Balsiger S, Ragaz C, Baron C, Narberhaus F (2004). “Régulation spécifique au réplicon des petits gènes de choc thermique dans Agrobacterium tumefaciens” . J Bacteriol . 186 (20): 6824–6829. est ce que je: 10.1128 / jb.186.20.6824-6829.2004 . PMC 522190 . PMID 15466035 .
Recent Comments