Một số biến số song song lặp lại (hoặc VNTR ) là một vị trí trong bộ gen, trong đó một chuỗi nucleotide ngắn được tổ chức như một sự lặp lại song song. Chúng có thể được tìm thấy trên nhiều nhiễm sắc thể và thường hiển thị các biến thể về độ dài (số lần lặp lại) giữa các cá thể. Mỗi biến thể hoạt động như một alen di truyền, cho phép chúng được sử dụng để nhận dạng cá nhân hoặc cha mẹ. Phân tích của họ rất hữu ích trong nghiên cứu di truyền và sinh học, pháp y và dấu vân tay DNA.
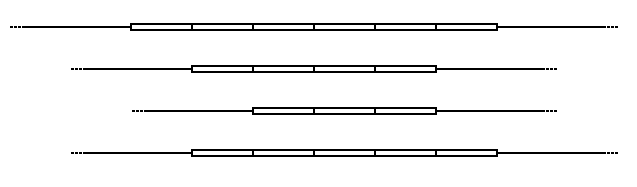

Cấu trúc và biến đổi alen [ 19659006] Trong sơ đồ trên, các khối hình chữ nhật đại diện cho từng chuỗi DNA lặp lại tại một vị trí VNTR cụ thể. Sự lặp lại song song – tức là chúng được nhóm lại với nhau và được định hướng theo cùng một hướng. Lặp lại riêng lẻ có thể được loại bỏ khỏi (hoặc thêm vào) VNTR thông qua các lỗi tái hợp hoặc sao chép, dẫn đến các alen có số lần lặp khác nhau. Bên cạnh các phần lặp lại là các phân đoạn của chuỗi không lặp lại (hiển thị ở đây là các dòng mỏng), cho phép các khối VNTR được chiết xuất bằng các enzyme cắt giới hạn và được phân tích bằng RFLP, hoặc được khuếch đại bằng kỹ thuật phản ứng chuỗi polymerase (PCR) và kích thước của chúng được xác định bằng gel điện di.
Sử dụng trong phân tích di truyền [ chỉnh sửa ]
VNTR là một nguồn quan trọng của các dấu hiệu di truyền RFLP được sử dụng trong phân tích liên kết (ánh xạ) của bộ gen lưỡng bội. Giờ đây, nhiều bộ gen đã được giải trình tự, các VNTR đã trở nên cần thiết cho các cuộc điều tra tội phạm pháp y, thông qua dấu vân tay DNA và cơ sở dữ liệu CODIS. Khi được loại bỏ khỏi DNA xung quanh bằng phương pháp PCR hoặc RFLP, và kích thước của chúng được xác định bằng điện di trên gel hoặc vết rộp phía Nam, chúng tạo ra một mô hình các dải duy nhất cho mỗi cá nhân. Khi được thử nghiệm với một nhóm các dấu hiệu VNTR độc lập, khả năng hai cá thể không liên quan 'có cùng một mô hình allelic là rất thấp. Phân tích VNTR cũng đang được sử dụng để nghiên cứu sự đa dạng di truyền và mô hình chăn nuôi trong quần thể động vật hoang dã hoặc thuần hóa. Như vậy, VNTR có thể được sử dụng để phân biệt các chủng mầm bệnh vi khuẩn. Trong bối cảnh pháp y vi sinh vật này, các xét nghiệm như vậy thường được gọi là Phân tích VNTR nhiều Loci hoặc MLVA.

Vị trí nhiễm sắc thể của 13 vị trí VNTR trong bảng điều khiển CODIS.
Di truyền [ chỉnh sửa ]
Trong phân tích dữ liệu VNTR, có thể sử dụng hai nguyên tắc di truyền cơ bản:
- Kết hợp nhận dạng – cả hai alen VNTR từ một vị trí cụ thể phải khớp. Nếu hai mẫu từ cùng một cá thể, chúng phải hiển thị cùng một kiểu alen.
- Kết hợp di truyền – các alen VNTR phải tuân theo quy tắc di truyền. Để phù hợp với một cá nhân với cha mẹ hoặc con cái của mình, một người phải có một alen phù hợp với một từ mỗi cha mẹ. Nếu mối quan hệ xa hơn, chẳng hạn như ông bà hoặc anh chị em, thì các trận đấu phải phù hợp với mức độ liên quan.
Mối quan hệ với các loại DNA lặp lại khác [ chỉnh sửa ]
DNA lặp đi lặp lại, đại diện cho hơn 40% bộ gen của con người, được sắp xếp trong một loạt các mẫu hoang mang. Lặp lại lần đầu tiên được xác định bằng cách trích xuất DNA vệ tinh, không tiết lộ cách chúng được tổ chức. Việc sử dụng các enzyme cắt giới hạn cho thấy một số khối lặp lại được xen kẽ trong toàn bộ bộ gen. Trình tự DNA sau đó cho thấy các lần lặp lại khác được tập hợp tại các vị trí cụ thể, với sự lặp lại song song là phổ biến hơn so với lặp lại đảo ngược (có thể cản trở sự sao chép DNA). Các VNTR là lớp lặp lại song song cụm thể hiện sự thay đổi allelic theo chiều dài của chúng.
Các lớp [ chỉnh sửa ]
 Điều này cho thấy một ví dụ lý thuyết về một VNTR ở hai cá nhân khác nhau. Một chuỗi DNA từ mỗi cá nhân được hiển thị trong đó có chuỗi lặp lại song song mà các cá nhân chia sẻ. Sự hiện diện trình tự là một VNTR vì một cá nhân có năm lần lặp lại, trong khi người kia có bảy lần lặp lại (số lần lặp lại khác nhau ở các cá nhân khác nhau). Mỗi lần lặp lại là mười nucleotide, làm cho nó trở thành một minisat Vệ tinh, chứ không phải là một kính hiển vi trong đó mỗi lần lặp lại là 1-6 nucleotide.
Điều này cho thấy một ví dụ lý thuyết về một VNTR ở hai cá nhân khác nhau. Một chuỗi DNA từ mỗi cá nhân được hiển thị trong đó có chuỗi lặp lại song song mà các cá nhân chia sẻ. Sự hiện diện trình tự là một VNTR vì một cá nhân có năm lần lặp lại, trong khi người kia có bảy lần lặp lại (số lần lặp lại khác nhau ở các cá nhân khác nhau). Mỗi lần lặp lại là mười nucleotide, làm cho nó trở thành một minisat Vệ tinh, chứ không phải là một kính hiển vi trong đó mỗi lần lặp lại là 1-6 nucleotide.
VNTR là một loại minisatocate trong đó kích thước của chuỗi lặp thường là mười đến một trăm cặp cơ sở . Minisatellites là một loại trình tự lặp lại song song DNA, có nghĩa là các trình tự lặp lại lần lượt mà không có trình tự hoặc nucleotide khác ở giữa chúng. Minisatellites được đặc trưng bởi một chuỗi lặp lại khoảng mười đến một trăm nucleotide, và số lần lặp lại trình tự thay đổi từ khoảng năm đến năm mươi lần. Trình tự của minisatellites lớn hơn so với microsatellites, trong đó trình tự lặp lại thường là 1 đến 6 nucleotide. Hai loại trình tự lặp lại đều song song nhưng được chỉ định bởi độ dài của trình tự lặp lại. Do đó, các VNTR vì chúng có các chuỗi lặp lại từ mười đến một trăm nucleotide trong đó mỗi lần lặp lại giống hệt nhau, được coi là minisatellites. Tuy nhiên, trong khi tất cả các VNTR là minisatellites, không phải tất cả các minisatellites đều là VNTR. Các VNTR có thể khác nhau về số lần lặp lại từ cá nhân này sang cá nhân khác, trong đó một số minisatellites không phải VNTR có trình tự lặp lại lặp lại cùng một số lần trong tất cả các cá nhân có lặp lại song song trong mã di truyền của chúng. [1][2]
Xem thêm [ chỉnh sửa ]
Phần mềm để gõ MLVA [ chỉnh sửa ]
Tài liệu tham khảo [ chỉnh sửa ]
19659005] [ chỉnh sửa ]

