Capsid – Wikipedia


EIN Kapsid ist die Proteinhülle eines Virus, die sein genetisches Material einschließt. Es besteht aus mehreren oligomeren (sich wiederholenden) Strukturuntereinheiten aus Protein, die als Protomere bezeichnet werden. Die beobachtbaren dreidimensionalen morphologischen Untereinheiten, die einzelnen Proteinen entsprechen können oder nicht, werden als Kapsomere bezeichnet. Die Proteine, aus denen das Kapsid besteht, werden genannt Kapsidproteine oder virale Hüllproteine (VCP). Das Kapsid und das innere Genom heißt das Nucleocapsid.
Kapside werden grob nach ihrer Struktur klassifiziert. Die Mehrheit der Viren hat Kapside mit entweder helikalem oder ikosaedrischem Charakter[2][3] Struktur. Einige Viren, wie Bakteriophagen, haben aufgrund von Einschränkungen der Elastizität und Elektrostatik kompliziertere Strukturen entwickelt.[4] Die ikosaedrische Form, die 20 gleichseitige dreieckige Flächen hat, nähert sich einer Kugel an, während die helikale Form der Form einer Feder ähnelt und den Raum eines Zylinders einnimmt, aber selbst kein Zylinder ist.[5] Die Kapsidflächen können aus einem oder mehreren Proteinen bestehen. Beispielsweise hat das Kapsid des Maul- und Klauenseuche-Virus Gesichter, die aus drei Proteinen mit den Namen VP1–3 bestehen.[6]
Einige Viren sind umhülltwas bedeutet, dass das Kapsid mit einer Lipidmembran beschichtet ist, die als das bekannt ist virale Hülle. Die Hülle wird vom Kapsid von einer intrazellulären Membran im Wirt des Virus aufgenommen; Beispiele hierfür sind die innere Kernmembran, die Golgi-Membran und die äußere Membran der Zelle.[7]
Sobald das Virus eine Zelle infiziert hat und sich selbst zu replizieren beginnt, werden neue Kapsiduntereinheiten unter Verwendung des Proteinbiosynthesemechanismus der Zelle synthetisiert. Bei einigen Viren, einschließlich solchen mit helikalen Kapsiden und insbesondere solchen mit RNA-Genomen, bilden die Kapsidproteine zusammen mit ihren Genomen. Bei anderen Viren, insbesondere komplexeren Viren mit doppelsträngigen DNA-Genomen, bilden die Kapsidproteine leere Vorläufer Prokapsiden Dazu gehört eine spezielle Portalstruktur an einem Scheitelpunkt. Über dieses Portal wird virale DNA in das Kapsid transloziert.[8]
Strukturanalysen von MCP-Architekturen (Major Capsid Protein) wurden verwendet, um Viren in Linien zu kategorisieren. Beispielsweise wurden der Bakteriophage PRD1, das Algenvirus Paramecium bursaria Chlorella-Virus (PBCV-1), das Mimivirus und das Säugetier-Adenovirus in dieselbe Linie eingeordnet, während doppelsträngige DNA-Bakteriophagen (Caudovirales) und Herpesvirus zu einer zweiten gehören Abstammung.[9][10][11][12]
Spezifische Formen[edit]
Ikosaeder[edit]


Die ikosaedrische Struktur ist bei Viren äußerst verbreitet. Das Ikosaeder besteht aus 20 dreieckigen Flächen, die durch 12 fünffache Scheitelpunkte begrenzt sind, und besteht aus 60 asymmetrischen Einheiten. Somit besteht ein ikosaedrisches Virus aus 60 N Proteinuntereinheiten. Die Anzahl und Anordnung der Kapsomere in einem ikosaedrischen Kapsid kann unter Verwendung des von Donald Caspar und Aaron Klug vorgeschlagenen “Quasi-Äquivalenzprinzips” klassifiziert werden.[13] Wie die Goldberg-Polyeder kann eine ikosaedrische Struktur als aus Pentameren und Hexameren aufgebaut angesehen werden. Die Strukturen können durch zwei ganze Zahlen indiziert werden h und kmit
und
;; Die Struktur kann als Einnahme betrachtet werden h Schritte vom Rand eines Pentamers, 60 Grad gegen den Uhrzeigersinn drehen und dann nehmen k Schritte, um zum nächsten Pentamer zu gelangen. Die Triangulationsnummer T. für das Kapsid ist definiert als:
In diesem Schema enthalten ikosaedrische Kapside 12 Pentamere plus 10 (T. – 1) Hexamere.[14][15] Das T.-Nummer ist repräsentativ für die Größe und Komplexität der Kapside.[16] Geometrische Beispiele für viele Werte von h, k, und T. finden Sie unter Liste der geodätischen Polyeder und Goldberg-Polyeder.
Es gibt viele Ausnahmen von dieser Regel: Beispielsweise haben die Polyomaviren und Papillomaviren Pentamere anstelle von Hexameren in sechswertigen Positionen auf einem Quasi-T = 7-Gitter. Mitglieder der doppelsträngigen RNA-Viruslinie, einschließlich Reovirus, Rotavirus und Bakteriophage φ6, haben Kapside, die aus 120 Kopien des Kapsidproteins aufgebaut sind, entsprechend einem “T = 2” -Kapsid oder wohl einem T = 1-Kapsid mit einem Dimer in der Asymmetrie Einheit. In ähnlicher Weise haben viele kleine Viren ein Pseudo-T = 3 (oder P = 3) -Kapsid, das gemäß einem T = 3-Gitter organisiert ist, wobei jedoch unterschiedliche Polypeptide die drei quasi äquivalenten Positionen einnehmen [17]
T-Nummern können beispielsweise auf unterschiedliche Weise dargestellt werden T. = 1 kann nur als Ikosaeder oder Dodekaeder dargestellt werden und je nach Art der Quasi-Symmetrie T. = 3 kann als abgeschnittenes Dodekaeder, Ikosidodekaeder oder abgeschnittenes Ikosaeder und ihre jeweiligen Dualen als Triakis-Ikosaeder, rhombisches Triacontaeder oder Pentakis-Dodekaeder dargestellt werden.[18][clarification needed]
Prolate[edit]
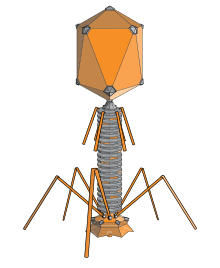
Ein längliches Ikosaeder ist eine häufige Form für die Köpfe von Bakteriophagen. Eine solche Struktur besteht aus einem Zylinder mit einer Kappe an jedem Ende. Der Zylinder besteht aus 10 länglichen dreieckigen Flächen. Die Q-Nummer (oder T.Mitte), die eine beliebige positive ganze Zahl sein kann,[19] Gibt die Anzahl der Dreiecke an, die aus asymmetrischen Untereinheiten bestehen und die 10 Dreiecke des Zylinders bilden. Die Kappen werden durch das T (oder T) klassifiziertEnde) Nummer.[20]
Das Bakterium E coli ist der Wirt für den Bakteriophagen T4, der eine prolate Kopfstruktur aufweist. Das für Bakteriophagen kodierte gp31-Protein scheint funktionell homolog zu sein E coli Chaparon-Protein GroES und in der Lage, es beim Aufbau von Bakteriophagen-T4-Virionen während der Infektion zu ersetzen.[21] Wie GroES bildet gp31 mit GroEL-Chaperonin einen stabilen Komplex, der für das Falten und Zusammenbauen unbedingt erforderlich ist in vivo des Bakteriophagen T4 Hauptkapsidproteins gp23.[21]
Helical[edit]

Viele stäbchenförmige und filamentöse Pflanzenviren haben Kapside mit helikaler Symmetrie.[22] Die helikale Struktur kann als eine Menge von beschrieben werden n 1-D molekulare Helices verwandt mit einem n-fache axiale Symmetrie.[23] Die helikale Transformation wird in zwei Kategorien eingeteilt: eindimensionale und zweidimensionale helikale Systeme.[23] Die Erzeugung einer gesamten helikalen Struktur beruht auf einer Reihe von Translations- und Rotationsmatrizen, die in der Proteindatenbank codiert sind.[23] Die helikale Symmetrie ist durch die Formel gegeben P. = μ x ρ, wo μ ist die Anzahl der Struktureinheiten pro Umdrehung der Helix, ρ ist der axiale Anstieg pro Einheit und P. ist die Tonhöhe der Helix. Die Struktur soll aufgrund der Eigenschaft offen sein, dass jedes Volumen durch Variieren der Länge der Helix eingeschlossen werden kann.[24] Das am meisten verstandene helikale Virus ist das Tabakmosaikvirus.[22] Das Virus ist ein einzelnes Molekül (+) Strang-RNA. Jedes Hüllprotein im Inneren der Helix bindet drei Nukleotide des RNA-Genoms. Influenza-A-Viren unterscheiden sich durch mehrere Ribonukleoproteine. Das virale NP-Protein organisiert die RNA in einer helikalen Struktur. Die Größe ist auch unterschiedlich; Das Tabakmosaikvirus hat 16,33 Proteinuntereinheiten pro helikaler Umdrehung.[22] während das Influenza-A-Virus eine Schwanzschleife mit 28 Aminosäuren aufweist.[25]
Funktionen[edit]
Die Funktionen des Kapsids sind:
- das Genom schützen,
- liefern das Genom, und
- mit dem Host interagieren.
Das Virus muss eine stabile, schützende Proteinhülle bilden, um das Genom vor tödlichen chemischen und physikalischen Einflüssen zu schützen. Dazu gehören Formen natürlicher Strahlung, extreme pH- oder Temperaturwerte sowie proteolytische und nukleolytische Enzyme. Bei nicht umhüllten Viren kann das Kapsid selbst an der Wechselwirkung mit Rezeptoren auf der Wirtszelle beteiligt sein, was zum Eindringen in die Wirtszellmembran und zur Internalisierung des Kapsids führt. Die Abgabe des Genoms erfolgt durch anschließendes Entfernen oder Zerlegen des Kapsids und Freisetzung des Genoms in das Zytoplasma oder durch Ausstoßen des Genoms durch eine spezialisierte Portalstruktur direkt in den Wirtszellkern.
Ursprung und Entwicklung[edit]
Es wurde vermutet, dass sich viele virale Kapsidproteine mehrfach aus funktionell unterschiedlichen zellulären Proteinen entwickelt haben.[26] Die Rekrutierung von zellulären Proteinen scheint in verschiedenen Stadien der Evolution stattgefunden zu haben, so dass einige zelluläre Proteine vor der Divergenz zellulärer Organismen in die drei gegenwärtigen Lebensbereiche eingefangen und refunktionalisiert wurden, während andere vor relativ kurzer Zeit entführt wurden. Infolgedessen sind einige Kapsidproteine in Viren weit verbreitet, die entfernte verwandte Organismen infizieren (z. B. Kapsidproteine mit der Geleerollenfalte), während andere auf eine bestimmte Gruppe von Viren beschränkt sind (z. B. Kapsidproteine von Alphaviren).[26][27]
Ein Rechenmodell (2015) hat gezeigt, dass Viruskapside möglicherweise aus der RNA-Welt stammen und als Mittel für den horizontalen Transfer zwischen Replikatorgemeinschaften dienen, da diese Gemeinschaften nicht überleben könnten, wenn die Anzahl der Genparasiten zunimmt, wobei bestimmte Gene verantwortlich sind für die Bildung dieser Strukturen und jener, die das Überleben selbstreplizierender Gemeinschaften begünstigten.[28] Die Verschiebung dieser Ahnengene zwischen zellulären Organismen könnte das Auftreten neuer Viren während der Evolution begünstigen.[27]
Siehe auch[edit]
Verweise[edit]
- ^ MA Asensio, NM Morella, CM Jakobson, EC Hartman, JE Glasgow, B Sankaran et al. (September 2016). “Eine Auswahl für den Zusammenbau zeigt, dass eine einzelne Aminosäuremutante des Bakteriophagen-MS2-Hüllproteins ein kleineres virusähnliches Teilchen bildet.”. Nano-Briefe. 16 (9): 5944–50. Bibcode:2016NanoL..16.5944A. doi:10.1021 / acs.nanolett.6b02948. PMID 27549001.
- ^ Lidmar J., Mirny L., Nelson DR (November 2003). “Virusformen und Knickübergänge in Kugelschalen”. Körperliche Überprüfung E.. 68 (5 Pt 1): 051910. arXiv:cond-mat / 0306741. Bibcode:2003PhRvE..68e1910L. doi:10.1103 / PhysRevE.68.051910. PMID 14682823. S2CID 6023873.
- ^ Vernizzi G., Olvera de la Cruz M. (November 2007). “Facettieren von Ionenschalen über Elektrostatik in Ikosaeder”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 104 (47): 18382–6. Bibcode:2007PNAS..10418382V. doi:10.1073 / pnas.0703431104. PMC 2141786. PMID 18003933.
- ^ Vernizzi G., Sknepnek R., Olvera de la Cruz M. (März 2011). “Platonische und archimedische Geometrien in elastischen Mehrkomponentenmembranen”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 108 (11): 4292–6. Bibcode:2011PNAS..108.4292V. doi:10.1073 / pnas.1012872108. PMC 3060260. PMID 21368184.
- ^ Branden C, Tooze J (1991). Einführung in die Proteinstruktur. New York: Girlande. S. 161–162. ISBN 978-0-8153-0270-4.
- ^ “Virusstruktur (web-books.com)”.
- ^ Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J. D. (1994). Molekularbiologie der Zelle (4. Aufl.). p. 280.
- ^ Newcomb WW, Homa FL, Brown JC (August 2005). “Beteiligung des Portals an einem frühen Schritt in der Herpes-simplex-Virus-Kapsid-Assemblierung”. Zeitschrift für Virologie. 79 (16): 10540–6. doi:10.1128 / JVI.79.16.10540-10546.2005. PMC 1182615. PMID 16051846.
- ^ Krupovic M, Bamford DH (Dezember 2008). “Virusentwicklung: Wie weit reicht die doppelte Beta-Barrel-Viruslinie?” Natur Bewertungen. Mikrobiologie. 6 (12): 941–8. doi:10.1038 / nrmicro2033. PMID 19008892. S2CID 31542714.
- ^ Forterre P (März 2006). “Drei RNA-Zellen für ribosomale Linien und drei DNA-Viren zur Replikation ihrer Genome: eine Hypothese für den Ursprung der zellulären Domäne”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 103 (10): 3669–74. Bibcode:2006PNAS..103.3669F. doi:10.1073 / pnas.0510333103. PMC 1450140. PMID 16505372.
- ^ Khayat R., Tang L., Larson ET, Lawrence CM, Young M., Johnson JE (Dezember 2005). “Die Struktur eines Archaealvirus-Kapsidproteins zeigt eine gemeinsame Abstammung von eukaryotischen und bakteriellen Viren.”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 102 (52): 18944–9. doi:10.1073 / pnas.0506383102. PMC 1323162. PMID 16357204.
- ^ Laurinmäki PA, Huiskonen JT, Bamford DH, Butcher SJ (Dezember 2005). “Membranproteine modulieren die Doppelschichtkrümmung im Bakterienvirus Bam35”. Struktur (London, England: 1993). 13 (12): 1819–28. doi:10.1016 / j.str.2005.08.020. PMID 16338410.
- ^ Caspar DL, Klug A (1962). “Physikalische Prinzipien beim Aufbau regulärer Viren”. Cold Spring Harbor Symposien zur quantitativen Biologie. 27: 1–24. doi:10.1101 / sqb.1962.027.001.005. PMID 14019094.
- ^ Carrillo-Tripp M., Shepherd CM, Borelli IA, Venkataraman S., Lander G., Natarajan P. et al. (Januar 2009). “VIPERdb2: eine erweiterte und Web-API-fähige relationale Datenbank für strukturelle Virologie”. Nukleinsäureforschung. 37 (Datenbankproblem): D436-42. doi:10.1093 / nar / gkn840. PMC 2686430. PMID 18981051.
- ^ Johnson JE, Speir JA (2009). Schreibtisch-Enzyklopädie der Allgemeinen Virologie. Boston: Akademische Presse. S. 115–123. ISBN 978-0-12-375146-1.
- ^ Mannige RV, Brooks CL (März 2010). “Periodensystem der Viruskapside: Auswirkungen auf die natürliche Selektion und das Design”. PLUS EINS. 5 (3): e9423. Bibcode:2010PLoSO … 5.9423M. doi:10.1371 / journal.pone.0009423. PMC 2831995. PMID 20209096.
- ^ Sgro J. “Virusworld”. Institut für Molekulare Virologie. Universität von Wisconsin-Madison.
- ^ Damodaran KV, Reddy VS, Johnson JE, Brooks CL (Dezember 2002). “Eine allgemeine Methode zur Quantifizierung der Quasi-Äquivalenz in ikosaedrischen Viren”. Journal of Molecular Biology. 324 (4): 723–37. doi:10.1016 / S0022-2836 (02) 01138-5. PMID 12460573.
- ^ Luque A, Reguera D (Juni 2010). “Die Struktur länglicher viraler Kapside”. Biophysical Journal. 98 (12): 2993–3003. Bibcode:2010BpJ …. 98.2993L. doi:10.1016 / j.bpj.2010.02.051. PMC 2884239. PMID 20550912.
- ^ Casjens S (2009). Schreibtisch-Enzyklopädie der Allgemeinen Virologie. Boston: Akademische Presse. S. 167–174. ISBN 978-0-12-375146-1.
- ^ ein b Marusich EI, Kurochkina LP, Mesyanzhinov VV. Chaperone in der Bakteriophagen-T4-Assemblierung. Biochemie (Mosc). 1998; 63 (4): 399 & ndash; 406
- ^ ein b c Yamada S., Matsuzawa T., Yamada K., Yoshioka S., Ono S., Hishinuma T. (Dezember 1986). “Modifiziertes Inversionswiederherstellungsverfahren für die Kernspinresonanztomographie”. Die Wissenschaftsberichte der Forschungsinstitute der Tohoku-Universität. Ser. C, Medizin. Tohoku Daigaku. 33 (1–4): 9–15. PMID 3629216.
- ^ ein b c Aldrich RA (Februar 1987). “Kinder in Städten – das KidsPlace-Programm in Seattle”. Acta Paediatrica Japonica. 29 (1): 84–90. doi:10.1111 / j.1442-200x.1987.tb00013.x. PMID 3144854.
- ^ Racaniello VR, Enquist LW (2008). Principles of Virology. 1: Molekularbiologie. Washington, DC: ASM Press. ISBN 978-1-55581-479-3.
- ^ Ye Q, Guu TS, Mata DA, Kuo RL, Smith B., Krug RM, Tao YJ (26. Dezember 2012). “Biochemische und strukturelle Belege zur Unterstützung eines kohärenten Modells für die Bildung des Doppelhelix-Influenza-A-Virus-Ribonukleoproteins”. mBio. 4 (1): e00467–12. doi:10.1128 / mBio.00467-12. PMC 3531806. PMID 23269829.
- ^ ein b Krupovic M, Koonin EV (März 2017). “Mehrere Ursprünge von viralen Kapsidproteinen von zellulären Vorfahren”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 114 (12): E2401 – E2410. doi:10.1073 / pnas.1621061114. PMC 5373398. PMID 28265094.
- ^ ein b Krupovic M, Dolja VV, Koonin EV (Juli 2019). “Ursprung der Viren: Urreplikatoren, die Kapside von Wirten rekrutieren” (PDF). Natur Bewertungen. Mikrobiologie. 17 (7): 449–458. doi:10.1038 / s41579-019-0205-6. PMID 31142823. S2CID 169035711.
- ^ Jalasvuori M., Mattila S., Hoikkala V. (2015). “Den Ursprung von Viren verfolgen: Kapsidbildende Gene als lebensrettende Voranpassung innerhalb einer Gemeinschaft früher Replikatoren”. PLUS EINS. 10 (5): e0126094. Bibcode:2015PLoSO..1026094J. doi:10.1371 / journal.pone.0126094. PMC 4425637. PMID 25955384.
Weiterführende Literatur[edit]
- Williams R (1. Juni 1979). Die geometrische Grundlage der natürlichen Struktur: Ein Quellbuch des Designs. S. 142–144, Abbildungen 4-49, 50, 51: Cluster aus 12 Kugeln, 42 Kugeln, 92 Kugeln. ISBN 978-0-486-23729-9.
- Pugh A (1. September 1976). Polyeder: Ein visueller Ansatz. Kapitel 6. Die geodätischen Polyeder von R. Buckminster Fuller und verwandten Polyedern. ISBN 978-0-520-02926-2.
- Almansour I, Alhagri M, Alfares R, Alshehri M, Bakhashwain R, Maarouf A (Januar 2019). “IRAM: Virus-Capsid-Datenbank und Analyseressource”. Datenbank: Das Journal of Biological Databases and Curation. 2019. doi:10.1093 / database / baz079. PMC 6637973. PMID 31318422.
Externe Links[edit]
| Wikimedia Commons hat Medien im Zusammenhang mit Kapsid. |
 und
und  ;; Die Struktur kann als Einnahme betrachtet werden h Schritte vom Rand eines Pentamers, 60 Grad gegen den Uhrzeigersinn drehen und dann nehmen k Schritte, um zum nächsten Pentamer zu gelangen. Die Triangulationsnummer T. für das Kapsid ist definiert als:
;; Die Struktur kann als Einnahme betrachtet werden h Schritte vom Rand eines Pentamers, 60 Grad gegen den Uhrzeigersinn drehen und dann nehmen k Schritte, um zum nächsten Pentamer zu gelangen. Die Triangulationsnummer T. für das Kapsid ist definiert als:

Recent Comments