Beta-Blatt – Wikipedia
Gemeinsames Motiv der regelmäßigen Sekundärstruktur in Proteinen; Dehnung der Polypeptidkette, typischerweise 3 bis 10 Aminosäuren lang, mit Rückgrat in einer erweiterten Konformation
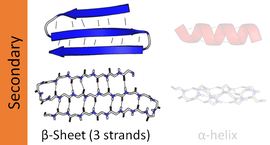
Das β-Faltblatt (ebenfalls β-Faltenblatt) ist ein häufiges Motiv der regulären Sekundärstruktur in Proteinen. Beta-Blätter bestehen aus Beta-Stränge (ebenfalls β-Strang) seitlich durch mindestens zwei oder drei Wasserstoffbrückenbindungen verbunden, die eine im Allgemeinen verdrillte, plissierte Schicht bilden. Ein β-Strang ist ein Abschnitt der Polypeptidkette, der typischerweise 3 bis 10 Aminosäuren lang ist und ein Grundgerüst in einer erweiterten Konformation aufweist. Die supramolekulare Assoziation von β-Faltblättern ist an der Bildung der Proteinaggregate und -fibrillen beteiligt, die bei vielen Erkrankungen des Menschen beobachtet wurden, insbesondere bei Amyloidosen wie der Alzheimer-Krankheit.
Geschichte[edit]

Die erste β-Faltblattstruktur wurde in den 1930er Jahren von William Astbury vorgeschlagen. Er schlug die Idee der Wasserstoffbindung zwischen den Peptidbindungen paralleler oder antiparallel verlängerter β-Stränge vor. Astbury verfügte jedoch nicht über die erforderlichen Daten zur Bindungsgeometrie der Aminosäuren, um genaue Modelle zu erstellen, zumal er damals nicht wusste, dass die Peptidbindung planar war. Eine verfeinerte Version wurde 1951 von Linus Pauling und Robert Corey vorgeschlagen. Ihr Modell enthielt die Planarität der Peptidbindung, die sie zuvor als Ergebnis der Keto-Enol-Tautomerisierung erklärt hatten.
Struktur und Orientierung[edit]
Geometrie[edit]
Die Mehrzahl der β-Stränge ist neben anderen Strängen angeordnet und bildet mit ihren Nachbarn ein ausgedehntes Wasserstoffbindungsnetzwerk, in dem die NH-Gruppen im Grundgerüst eines Strangs Wasserstoffbrücken mit den C = O-Gruppen im Grundgerüst des Nachbarstrangs herstellen Stränge. In dem vollständig ausgedehnten β-Strang zeigen aufeinanderfolgende Seitenketten gerade nach oben, dann gerade nach unten, dann gerade nach oben usw. Benachbarte β-Stränge in einem β-Faltblatt sind so ausgerichtet, dass ihr C.α Atome sind benachbart und ihre Seitenketten zeigen in die gleiche Richtung. Das “plissierte” Erscheinungsbild von β-Strängen ergibt sich aus der tetraedrischen chemischen Bindung am C.α Atom; Wenn beispielsweise eine Seitenkette gerade nach oben zeigt, müssen die Bindungen an C ‘leicht nach unten zeigen, da ihr Bindungswinkel ungefähr 109,5 ° beträgt. Die Faltung bewirkt den Abstand zwischen C.α
ich und Cα
ich + 2 ungefähr 6 Å (0,60 nm) sein, anstatt der 7,6 Å (0,76 nm), die von zwei vollständig ausgedehnten erwartet werden trans Peptide. Der “seitliche” Abstand zwischen benachbarten C.α Atome in wasserstoffgebundenen β-Strängen betragen ungefähr 5 Å (0,50 nm).
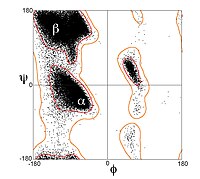
Β-Stränge sind jedoch selten perfekt verlängert; Vielmehr weisen sie eine Wendung auf. Die energetisch bevorzugten Diederwinkel nahe (φ, ψ) = (–135 °, 135 °) (im Großen und Ganzen der obere linke Bereich des Ramachandran-Diagramms) weicht signifikant von der vollständig erweiterten Konformation ab (φ, ψ) = (–180 °, 180 °).[1] Die Verdrehung ist häufig mit abwechselnden Schwankungen der Diederwinkel verbunden, um zu verhindern, dass die einzelnen β-Stränge in einem größeren Blatt auseinander spreizen. Ein gutes Beispiel für eine stark verdrehte β-Haarnadel ist das Protein BPTI.
Die Seitenketten zeigen von den Falten der Falten nach außen, ungefähr senkrecht zur Ebene des Blattes; Aufeinanderfolgende Aminosäurereste zeigen auf abwechselnden Flächen des Blattes nach außen.
Wasserstoffbrückenmuster[edit]
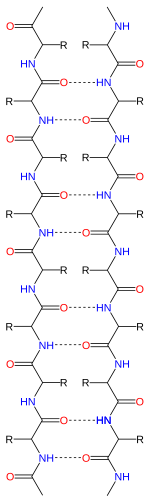

Da Peptidketten eine durch ihren N-Terminus und C-Terminus verliehene Richtwirkung haben, kann auch gesagt werden, dass β-Stränge gerichtet sind. Sie werden normalerweise in Proteintopologiediagrammen durch einen Pfeil dargestellt, der zum C-Terminus zeigt. Benachbarte β-Stränge können Wasserstoffbrücken in antiparalleler, paralleler oder gemischter Anordnung bilden.
In einer antiparallelen Anordnung wechseln die aufeinanderfolgenden β-Stränge die Richtungen, so dass der N-Terminus eines Strangs dem C-Terminus des nächsten benachbart ist. Dies ist die Anordnung, die die stärkste Stabilität zwischen den Strängen erzeugt, da dadurch die Wasserstoffbrücken zwischen den Strängen zwischen Carbonylen und Aminen planar sein können, was ihre bevorzugte Orientierung ist. Die Diederwinkel des Peptidrückgrats (φ, ψ) sind ungefähr (–140 °, 135 °) in antiparallelen Platten. In diesem Fall, wenn zwei Atome C.α
ich und Cα
j sind in zwei wasserstoffgebundenen β-Strängen benachbart, dann bilden sie zwei gegenseitige Wasserstoffbrückenbindungen im Grundgerüst zu den flankierenden Peptidgruppen des anderen; Dies ist bekannt als enges Paar von Wasserstoffbrückenbindungen.
In einer parallelen Anordnung sind alle N-Termini aufeinanderfolgender Stränge in die gleiche Richtung ausgerichtet; Diese Orientierung kann etwas weniger stabil sein, da sie eine Nichtplanarität in das Wasserstoffbrückenbindungsmuster zwischen den Strängen einführt. Die Diederwinkel (φ, ψ) sind ungefähr (–120 °, 115 °) in parallelen Blättern. Es ist selten, weniger als fünf wechselwirkende parallele Stränge in einem Motiv zu finden, was darauf hindeutet, dass eine geringere Anzahl von Strängen instabil sein kann. Es ist jedoch auch grundlegend schwieriger, parallele β-Faltblätter zu bilden, da Stränge mit N- und C-Termini unbedingt ausgerichtet sein müssen in der Reihenfolge sehr weit entfernt sein[citation needed]. Es gibt auch Hinweise darauf, dass paralleles β-Faltblatt stabiler sein kann, da kleine amyloidogene Sequenzen im Allgemeinen zu β-Faltblattfibrillen zu aggregieren scheinen, die hauptsächlich aus parallelen β-Faltblattsträngen bestehen, wobei man antiparallele Fibrillen erwarten würde, wenn antiparallel stabil wäre .
In paralleler β-Faltblattstruktur, wenn zwei Atome C.α
ich und Cα
j sind in zwei wasserstoffgebundenen β-Strängen benachbart, dann tun sie es nicht Wasserstoffbrücke untereinander; Vielmehr bildet ein Rest Wasserstoffbrücken zu den Resten, die den anderen flankieren (aber nicht umgekehrt). Zum Beispiel Rückstände ich kann Wasserstoffbrücken zu Rückständen bilden j – 1 und j + 1; Dies ist bekannt als breites Paar von Wasserstoffbrückenbindungen. Im Gegensatz dazu Rückstände j kann Wasserstoffbrücken an verschiedene Reste insgesamt oder an gar keine binden.
Die Wasserstoffbrückenanordnung im parallelen Beta-Faltblatt ähnelt der in einem Amidringmotiv mit 11 Atomen.
Schließlich kann ein einzelner Strang ein gemischtes Bindungsmuster aufweisen, mit einem parallelen Strang auf der einen Seite und einem antiparallelen Strang auf der anderen Seite. Solche Anordnungen sind weniger verbreitet, als eine zufällige Verteilung von Orientierungen vermuten lässt, was darauf hindeutet, dass dieses Muster weniger stabil ist als die antiparallele Anordnung. Die bioinformatische Analyse hat jedoch immer Probleme mit der Extraktion der strukturellen Thermodynamik, da in ganzen Proteinen immer zahlreiche andere strukturelle Merkmale vorhanden sind. Auch Proteine sind von Natur aus durch die Faltungskinetik sowie die Faltungsthermodynamik eingeschränkt, so dass man immer vorsichtig sein muss, um die Stabilität aus der bioinformatischen Analyse zu schließen.
Die Wasserstoffbindung von β-Strängen muss nicht perfekt sein, kann jedoch lokalisierte Störungen aufweisen, die als β-Ausbuchtungen bekannt sind.
Die Wasserstoffbrückenbindungen liegen ungefähr in der Ebene der Schicht, wobei die Peptidcarbonylgruppen mit aufeinanderfolgenden Resten in wechselnde Richtungen zeigen; Zum Vergleich weisen aufeinanderfolgende Carbonyle auf die gleich Richtung in der Alpha-Helix.
Aminosäure-Neigungen[edit]
Große aromatische Reste (Tyrosin, Phenylalanin, Tryptophan) und β-verzweigte Aminosäuren (Threonin, Valin, Isoleucin) sind bevorzugt in β-Strängen in der EU zu finden Mitte von β-Faltblättern. Verschiedene Arten von Rückständen (wie Prolin) sind wahrscheinlich in der zu finden Kante Stränge in β-Faltblättern, vermutlich um die “Rand-zu-Rand” -Verbindung zwischen Proteinen zu vermeiden, die zur Aggregation und Amyloidbildung führen könnte.[2]
Gemeinsame Strukturmotive[edit]


Ein sehr einfaches Strukturmotiv mit β-Faltblättern ist die β-Haarnadel, bei der zwei antiparallele Stränge durch eine kurze Schleife von zwei bis fünf Resten verbunden sind, von denen einer häufig ein Glycin oder ein Prolin ist, die beide die Dieder annehmen können Winkelkonformationen, die für eine enge Drehung oder eine β-Ausbuchtungsschleife erforderlich sind. Einzelne Stränge können auch auf aufwändigere Weise mit längeren Schleifen verknüpft werden, die α-Helices enthalten können.
Griechisches Schlüsselmotiv[edit]
Das griechische Schlüsselmotiv besteht aus vier benachbarten antiparallelen Strängen und ihren Verbindungsschleifen. Es besteht aus drei antiparallelen Strängen, die durch Haarnadeln verbunden sind, während der vierte an den ersten angrenzt und durch eine längere Schleife mit dem dritten verbunden ist. Diese Art von Struktur bildet sich leicht während des Proteinfaltungsprozesses.[3][4] Es wurde nach einem Muster benannt, das griechischen Zierkunstwerken gemeinsam ist (siehe Mäander).
β-α-β-Motiv[edit]
Aufgrund der Chiralität ihrer Aminosäurekomponenten weisen alle Stränge eine rechtshändige Verdrehung auf, die in den meisten β-Faltblattstrukturen höherer Ordnung erkennbar ist. Insbesondere die Verbindungsschleife zwischen zwei parallelen Strängen weist fast immer eine rechtshändige Crossover-Chiralität auf, die durch die inhärente Verdrehung des Blattes stark begünstigt wird.[5] Diese Verknüpfungsschleife enthält häufig eine helikale Region. In diesem Fall wird sie als β-α-β-Motiv bezeichnet. Ein eng verwandtes Motiv, das als β-α-β-α-Motiv bezeichnet wird, bildet die Grundkomponente der am häufigsten beobachteten Protein-Tertiärstruktur, des TIM-Zylinders.

β-Mäander-Motiv[edit]
Eine einfache supersekundäre Proteintopologie, die aus zwei oder mehr aufeinanderfolgenden antiparallelen β-Strängen besteht, die durch Haarnadelschleifen miteinander verbunden sind.[6][7] Dieses Motiv ist in β-Faltblättern üblich und kann in verschiedenen Strukturarchitekturen gefunden werden, einschließlich β-Fässern und β-Propellern.

Psi-Loop-Motiv[edit]
Das Psi-Loop-Motiv (Ψ-Loop) besteht aus zwei antiparallelen Strängen mit einem dazwischen liegenden Strang, der durch Wasserstoffbrücken mit beiden verbunden ist.[8] Es gibt vier mögliche Strang-Topologien für einzelne Ψ-Schleifen.[9] Dieses Motiv ist selten, da es unwahrscheinlich ist, dass der Prozess, der zu seiner Bildung führt, während der Proteinfaltung auftritt. Die Ψ-Schleife wurde erstmals in der Aspartat-Protease-Familie identifiziert.[9]
Strukturarchitekturen von Proteinen mit β-Faltblättern[edit]
β-Faltblätter sind in allen β-, α + β- und α / β-Domänen sowie in vielen Peptiden oder kleinen Proteinen mit schlecht definierter Gesamtarchitektur vorhanden.[10][11]All-β-Domänen können β-Fässer, β-Sandwiches, β-Prismen, β-Propeller und β-Helices bilden.
Strukturelle Topologie[edit]
Das Topologie eines β-Faltblatts beschreibt die Reihenfolge der wasserstoffgebundenen β-Stränge entlang des Grundgerüsts. Zum Beispiel hat die Flavodoxinfalte ein fünfsträngiges, paralleles β-Faltblatt mit der Topologie 21345; Somit sind die Randstränge β-Strang 2 und β-Strang 5 entlang des Rückgrats. Ausdrücklich ausgedrückt ist β-Strang 2 H-gebunden an β-Strang 1, der an β-Strang 3 H-gebunden ist, der an β-Strang 4 H-gebunden ist, der an β-Strang 5 H-gebunden ist der andere Randstrang. Im gleichen System hat das oben beschriebene griechische Schlüsselmotiv eine 4123-Topologie. Die Sekundärstruktur eines β-Faltblatts kann grob beschrieben werden, indem die Anzahl der Stränge, ihre Topologie und ob ihre Wasserstoffbrückenbindungen parallel oder antiparallel sind, angegeben werden.
β-Faltblätter können sein öffnen, was bedeutet, dass sie zwei Randstränge haben (wie in der Flavodoxin-Falte oder der Immunglobulin-Falte) oder sie können sein geschlossene β-Fässer (wie das TIM-Fass). β-Fässer werden oft durch ihre beschrieben staffeln oder scheren. Einige offene β-Faltblätter sind sehr gekrümmt und falten sich auf sich selbst (wie in der SH3-Domäne) oder bilden Hufeisenformen (wie im Ribonuklease-Inhibitor). Offene β-Faltblätter können sich von Angesicht zu Angesicht (wie die β-Propeller-Domäne oder die Immunglobulinfalte) oder von Kante zu Kante zusammensetzen und ein großes β-Faltblatt bilden.
Dynamische Funktionen[edit]
β-Faltblattstrukturen bestehen aus verlängerten β-Strang-Polypeptidketten, wobei die Stränge durch Wasserstoffbrücken mit ihren Nachbarn verbunden sind. Aufgrund dieser erweiterten Rückgratkonformation widerstehen β-Faltblätter einer Dehnung. β-Faltblätter in Proteinen können niederfrequente akkordeonartige Bewegungen ausführen, wie sie durch die Raman-Spektroskopie beobachtet werden[12] und mit dem Quasi-Kontinuum-Modell analysiert.[13]
Parallele β-Helices[edit]

Eine β-Helix wird aus sich wiederholenden Struktureinheiten gebildet, die aus zwei oder drei kurzen β-Strängen bestehen, die durch kurze Schleifen verbunden sind. Diese Einheiten “stapeln” sich helixartig übereinander, so dass aufeinanderfolgende Wiederholungen derselben Wasserstoffbrücke in paralleler Ausrichtung miteinander verbunden sind. Weitere Informationen finden Sie im Artikel zur β-Helix.
In linkshändigen β-Helices sind die Stränge selbst ziemlich gerade und ungedreht; Die resultierenden helikalen Oberflächen sind nahezu flach und bilden eine regelmäßige dreieckige Prismenform, wie für die archaeale Carboanhydrase 1QRE rechts gezeigt. Andere Beispiele sind das Lipid A-Syntheseenzym LpxA und Insekten-Frostschutzproteine mit einer regelmäßigen Anordnung von Thr-Seitenketten auf einer Seite, die die Struktur von Eis nachahmen.[14]
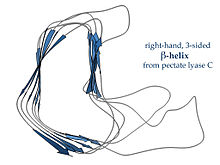
Rechtshändige β-Helices, die durch das links gezeigte Pektat-Lyase-Enzym oder das P22-Phagen-Tailspike-Protein charakterisiert sind, haben einen weniger regelmäßigen Querschnitt, sind länger und auf einer der Seiten eingerückt. Von den drei Linkerschleifen ist eine konsistent nur zwei Reste lang und die anderen sind variabel, oft ausgearbeitet, um eine Bindungs- oder aktive Stelle zu bilden.[15]
In einigen bakteriellen Metalloproteasen findet sich eine zweiseitige β-Helix (rechtshändig); Seine zwei Schleifen sind jeweils sechs Reste lang und binden stabilisierende Calciumionen, um die Integrität der Struktur aufrechtzuerhalten, wobei das Sauerstoffgerüst des Rückgrats und der Asp-Seitenkette eines GGXGXD-Sequenzmotivs verwendet wird.[16] Diese Falte wird in der SCOP-Klassifikation als β-Rolle bezeichnet.
In der Pathologie[edit]
Einige Proteine, die als Monomere ungeordnet oder helikal sind, wie Amyloid β (siehe Amyloidplaque), können β-Faltblatt-reiche oligomere Strukturen bilden, die mit pathologischen Zuständen assoziiert sind. Die oligomere Form des Amyloid-β-Proteins ist eine Ursache für Alzheimer. Seine Struktur muss noch vollständig bestimmt werden, aber neuere Daten legen nahe, dass es einer ungewöhnlichen zweisträngigen β-Helix ähneln könnte.[17]
Die Seitenketten aus den Aminosäureresten, die in einer β-Faltblattstruktur gefunden werden, können auch so angeordnet sein, dass viele der benachbarten Seitenketten auf einer Seite der Folie hydrophob sind, während viele der benachbarten auf der alternativen Seite der Folie nebeneinander liegen sind polar oder geladen (hydrophil),[18] Dies kann nützlich sein, wenn das Blatt eine Grenze zwischen polaren / wässrigen und unpolaren / fettigen Umgebungen bilden soll.
Siehe auch[edit]
Verweise[edit]
- ^ Voet D, Voet JG (2004). Biochemie (3. Aufl.). Hoboken, NJ: Wiley. pp. 227–231. ISBN 0-471-19350-X.
- ^ Richardson JS, Richardson DC (März 2002). “Natürliche Beta-Faltblatt-Proteine verwenden ein negatives Design, um eine Aggregation von Kante zu Kante zu vermeiden.”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 99 (5): 2754–9. doi:10.1073 / pnas.052706099. PMC 122420. PMID 11880627.
- ^ Struktur und Falten des tertiären Proteins: Abschnitt 4.3.2.1. Von Prinzipien der Proteinstruktur, vergleichende Proteinmodellierung und Visualisierung
- ^ Hutchinson EG, Thornton JM (April 1993). “Das griechische Schlüsselmotiv: Extraktion, Klassifizierung und Analyse”. Protein Engineering. 6 (3): 233–45. doi:10.1093 / Protein / 6.3.233. PMID 8506258.
- ^ Siehe Abschnitte II B und III C, D in Richardson JS (1981). Anatomie und Taxonomie von Proteinstrukturen. Fortschritte in der Proteinchemie. 34. S. 167–339. doi:10.1016 / s0065-3233 (08) 60520-3. ISBN 0-12-034234-0.
- ^ “SCOP: Fold: WW Domain-like”. Archiviert von das Original am 04.02.2012. Abgerufen 2007-06-01.
- ^ PPS ’96 – Super-Sekundärstruktur
- ^ Hutchinson EG, Thornton JM (Februar 1996). “PROMOTIF – ein Programm zur Identifizierung und Analyse von Strukturmotiven in Proteinen”. Proteinwissenschaft. 5 (2): 212–20. doi:10.1002 / pro.5560050204. PMC 2143354. PMID 8745398.
- ^ ein b Hutchinson EG, Thornton JM (1990). “HERA – ein Programm zum Zeichnen schematischer Diagramme von Proteinsekundärstrukturen”. Proteine. 8 (3): 203–12. doi:10.1002 / prot.340080303. PMID 2281084.
- ^ Hubbard TJ, Murzin AG, Brenner SE, Chothia C (Januar 1997). “SCOP: eine strukturelle Klassifikation der Proteindatenbank”. Nukleinsäureforschung. 25 (1): 236–9. doi:10.1093 / nar / 25.1.236. PMC 146380. PMID 9016544.
- ^ Fox NK, Brenner SE, Chandonia JM (Januar 2014). “SCOPe: Strukturelle Klassifizierung von Proteinen – erweitert, Integration von SCOP- und ASTRAL-Daten und Klassifizierung neuer Strukturen”. Nukleinsäureforschung. 42 (Datenbankproblem): D304-9. doi:10.1093 / nar / gkt1240. PMC 3965108. PMID 24304899.
- ^ Maler PC, Mosher LE, Rhoads C (Juli 1982). “Niederfrequenzmoden in den Raman-Spektren von Proteinen”. Biopolymere. 21 (7): 1469–72. doi:10.1002 / bip.360210715. PMID 7115900.
- ^ Chou KC (August 1985). “Niederfrequenzbewegungen in Proteinmolekülen. Beta-Sheet und Beta-Barrel”. Biophysical Journal. 48 (2): 289–97. doi:10.1016 / S0006-3495 (85) 83782-6. PMC 1329320. PMID 4052563.
- ^ Liou YC, Tocilj A., Davies PL, Jia Z. (Juli 2000). “Nachahmung der Eisstruktur durch Oberflächenhydroxylgruppen und Wasser eines Beta-Helix-Frostschutzproteins”. Natur. 406 (6793): 322–4. doi:10.1038 / 35018604. PMID 10917536.
- ^ Branden C, Tooze J (1999). Einführung in die Proteinstruktur. New York: Girlande. S. 20–32. ISBN 0-8153-2305-0.
- ^ Baumann U., Wu S., Flaherty KM, McKay DB (September 1993). “Dreidimensionale Struktur der alkalischen Protease von Pseudomonas aeruginosa: ein Zwei-Domänen-Protein mit einem Calcium-bindenden parallelen Beta-Roll-Motiv”. Das EMBO Journal. 12 (9): 3357–64. PMC 413609. PMID 8253063.
- ^ Nelson R., Sawaya MR, Balbirnie M., Madsen AØ, Riekel C., Grothe R., Eisenberg D. (Juni 2005). “Struktur der Cross-Beta-Kiefer von Amyloid-ähnlichen Fibrillen”. Natur. 435 (7043): 773–8. doi:10.1038 / nature03680. PMC 1479801. PMID 15944695.
- ^ Zhang S., Holmes T., Lockshin C., Rich A. (April 1993). “Spontane Assemblierung eines selbstkomplementären Oligopeptids zur Bildung einer stabilen makroskopischen Membran”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 90 (8): 3334–8. doi:10.1073 / pnas.90.8.3334. PMC 46294. PMID 7682699.
Weiterführende Literatur[edit]
Externe Links[edit]
Recent Comments