ATP5F1A – Wikipedia
| ATP5F1A | |||
|---|---|---|---|
 |
|||
| Bezeichner | |||
| Aliase | ATP5F1A, ATP5A, ATP5AL2, ATPM, HEL-S-123m, MC5DN4, MOM2, OMR, ORM, hATP1, COXPD22, ATP-Synthase, H+-Transport, mitochondrialer F1-Komplex, alpha 1, ATP-Synthase, H+-Transport, mitochondrialer F1-Komplex, alpha-Untereinheit 1, Herzmuskel, ATP5A1, ATP-Synthase F1-Untereinheit alpha | ||
| Externe IDs | OMIM: 164360 MGI: 88115 Homologen: 2985 GenCards: ATP5F1A | ||
| Orthologe | |||
| Spezies | Menschlich | Maus | |
| Entrez | |||
| Ensemble | |||
| UniProt | |||
| RefSeq (mRNA) | |||
| RefSeq (Protein) | |||
| Standort (UCSC) | Chr 18: 46,08 – 46,1 Mb | Chr 18: 77,77 – 77,78 Mb | |
| PubMed-Suche | [3] | [4] | |
| Wikidata | |||
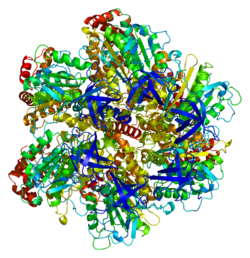
ATP-Synthase F1-Untereinheit alpha, mitochondrial ist ein Enzym, das beim Menschen durch die ATP5F1A Gen.[5][6]
Funktion[edit]
Dieses Gen kodiert für eine Untereinheit der mitochondrialen ATP-Synthase. Die mitochondriale ATP-Synthase katalysiert die ATP-Synthese unter Verwendung eines elektrochemischen Gradienten von Protonen durch die innere Membran während der oxidativen Phosphorylierung. Die ATP-Synthase besteht aus zwei verbundenen Komplexen aus mehreren Untereinheiten: dem löslichen katalytischen Kern, F1, und die membranaufspannende Komponente FÖ, umfassend den Protonenkanal. Der katalytische Teil der mitochondrialen ATP-Synthase besteht aus 5 verschiedenen Untereinheiten (Alpha, Beta, Gamma, Delta und Epsilon), die mit einer Stöchiometrie von 3 Alpha, 3 Beta und einem einzigen Vertreter der anderen 3 zusammengesetzt sind. Der Protonenkanal besteht aus drei Hauptuntereinheiten (a, b, c). Dieses Gen kodiert die Alpha-Untereinheit des katalytischen Kerns. Alternativ wurden gespleißte Transkriptvarianten identifiziert, die das gleiche Protein kodieren. Pseudogene dieses Gens befinden sich auf den Chromosomen 9, 2 und 16.[6]
Struktur[edit]
Die ATP5F1A Gen, das sich auf dem q-Arm von Chromosom 18 in Position 21 befindet, besteht aus 13 Exons und ist 20.090 Basenpaare lang.[6] Das Protein ATP5F1A wiegt 59,7 kDa und besteht aus 553 Aminosäuren.[7][8] Das Protein ist eine Untereinheit des katalytischen Teils des F1FÖ ATPase, auch als Komplex V bekannt, besteht aus 14 nuklearen und 2 mitochondrial kodierten Untereinheiten. Als Alpha-Untereinheit ist ATP5F1A im katalytischen F . enthalten1 Teil des Komplexes.[6] Die Nomenklatur des Enzyms hat eine lange Geschichte. Die F1 Bruch leitet seinen Namen vom Begriff “Fraktion 1” und F . abÖ (geschrieben als tiefgestellter Buchstabe “o”, nicht “Null”) leitet seinen Namen von der Bindungsfraktion für Oligomycin ab, eine Art natürlich gewonnenes Antibiotikum, das in der Lage ist, das F . zu hemmenÖ Einheit der ATP-Synthase.[9][10] Die F1 Partikel ist groß und kann im Transmissionselektronenmikroskop durch negative Färbung gesehen werden.[11] Dabei handelt es sich um Partikel mit einem Durchmesser von 9 nm, die die innere Mitochondrienmembran pfeffern. Sie wurden ursprünglich Elementarteilchen genannt und man dachte, dass sie den gesamten Atmungsapparat des Mitochondriums enthalten, aber durch eine lange Reihe von Experimenten haben Efraim Racker und seine Kollegen (die zuerst die F . isolierten)1 Partikel im Jahr 1961) konnten zeigen, dass dieses Partikel mit der ATPase-Aktivität in ungekoppelten Mitochondrien und mit der ATPase-Aktivität in subchochondrialen Partikeln korreliert, die durch Ultraschalleinwirkung von Mitochondrien erzeugt wurden. Diese ATPase-Aktivität wurde außerdem durch eine lange Reihe von Experimenten in vielen Laboratorien mit der Bildung von ATP in Verbindung gebracht.
Funktion[edit]
ATP-Synthase der Mitochondrienmembran (F1FÖ ATP-Synthase oder Komplex V) produziert ATP aus ADP in Gegenwart eines Protonengradienten durch die Membran, der durch Elektronentransportkomplexe der Atmungskette erzeugt wird. F-Typ-ATPasen bestehen aus zwei strukturellen Domänen, F1 – enthält den extramembranösen katalytischen Kern und FÖ – den Membran-Protonenkanal enthaltend, miteinander verbunden durch einen zentralen Stiel und einen peripheren Stiel. Während der Katalyse wird die ATP-Synthese in der katalytischen Domäne von F1 ist über einen Drehmechanismus der zentralen Stiel-Untereinheiten an die Protonen-Translokation gekoppelt. Untereinheiten alpha und beta bilden den katalytischen Kern in F1. Die Rotation des zentralen Stiels gegen die umgebenden alpha(3)beta(3)-Untereinheiten führt zur Hydrolyse von ATP an drei separaten katalytischen Zentren auf den Beta-Untereinheiten. Die Untereinheit alpha trägt nicht die katalytisch hochaffinen ATP-Bindungsstellen.[12]
Klinische Bedeutung[edit]
Mutationen, die das ATP5F1A-Gen betreffen, verursachen einen kombinierten oxidativen Phosphorylierungsmangel 22 (COXPD22), eine mitochondriale Erkrankung, die durch intrauterine Wachstumsverzögerung, Mikrozephalie, Hypotonie, pulmonale Hypertonie, Gedeihstörung, Enzephalopathie und Herzinsuffizienz gekennzeichnet ist. Mutationen im ATP5F1A-Gen verursachen auch mitochondrialen Komplex V-Mangel, Nuclear 4 (MC5DN4), eine mitochondriale Störung mit heterogenen klinischen Manifestationen, einschließlich dysmorphen Merkmalen, psychomotorischer Retardierung, Hypotonie, Wachstumsverzögerung, Kardiomyopathie, vergrößerter Leber, hypoplastischen Nieren und erhöhten Laktatwerten im Urin , Plasma und Liquor.[13]
Die Resveratrol-Hemmung des katalytischen F1-Kerns erhöht die Adenosinmonophosphat-(AMP)-Spiegel, wodurch das AMP-aktivierte Proteinkinase-Enzym aktiviert wird.[14]
Modellorganismen[edit]
Modellorganismen wurden bei der Untersuchung der ATP5F1A-Funktion verwendet. Eine bedingte Knockout-Mauslinie, genannt Atp5a1tm1a(EUCOMM)Wtsi[21][22] wurde im Rahmen des International Knockout Mouse Consortium-Programms erstellt – einem Hochdurchsatz-Mutagenese-Projekt zur Generierung und Verteilung von Tiermodellen von Krankheiten an interessierte Wissenschaftler.[23][24][25]
Männliche und weibliche Tiere wurden einem standardisierten phänotypischen Screening unterzogen, um die Auswirkungen der Deletion zu bestimmen.[19][26] An mutierten Mäusen wurden 22 Tests durchgeführt und es wurden 5 signifikante Anomalien beobachtet.[19] Während der Trächtigkeit wurden keine homozygoten mutierten Embryonen identifiziert, und daher überlebte keiner bis zum Absetzen. Die restlichen Tests wurden an heterozygoten mutierten adulten Mäusen durchgeführt und bei weiblichen Tieren wurde ein verringertes Körpergewicht, eine fettfreie Körpermasse und eine Hypoproteinämie beobachtet.[19]
Verweise[edit]
- ^ ein B C GRCh38: Ensemble-Release 89: ENSG00000152234 – Ensemble, Mai 2017
- ^ ein B C GRCm38: Ensemble-Release 89: ENSMUSG00000025428 – Ensemble, Mai 2017
- ^ “Menschliche PubMed-Referenz:”. National Center for Biotechnology Information, US National Library of Medicine.
- ^ “Maus PubMed-Referenz:”. National Center for Biotechnology Information, US National Library of Medicine.
- ^ Kataoka H, Biswas C (Juli 1991). „Nukleotidsequenz einer cDNA für die Alpha-Untereinheit der menschlichen mitochondrialen ATP-Synthase“. Biochimica et Biophysica Acta (BBA) – Genstruktur und Expression. 1089 (3): 393–5. mach:10.1016/0167-4781(91)90183-m. PMID 1830491.
- ^ ein B C D “Entrez-Gen: ATP5F1A ATP-Synthase F1-Untereinheit alpha”.
- ^ Zong NC, Li H, Li H, Lam MP, Jimenez RC, Kim CS, Deng N, Kim AK, Choi JH, Zelaya I, Liem D, Meyer D, Odeberg J, Fang C, Lu HJ, Xu T, Weiss J , Duan H, Uhlen M, Yates JR, Apweiler R, Ge J, Hermjakob H, Ping P (Oktober 2013). “Integration von kardialer Proteombiologie und Medizin durch eine spezialisierte Wissensdatenbank”. Auflagenforschung. 113 (9): 1043–53. mach:10.1161/CIRCRESAHA.113.301151. PMC 4076475. PMID 23965338.
- ^ “ATP-Synthase-Untereinheit alpha, mitochondrial”. Wissensdatenbank zu kardialen Organellen Proteinatlas (COPaKB). Archiviert von das Original am 2018-07-20. Abgerufen 2018-07-18.
- ^ Kagawa Y, Racker E (Mai 1966). “Partielle Auflösung der Enzyme, die die oxidative Phosphorylierung katalysieren. 8. Eigenschaften eines Faktors, der der mitochondrialen Adenosintriphosphatase eine Oligomycin-Empfindlichkeit verleiht”. Die Zeitschrift für biologische Chemie. 241 (10): 2461–6. mach:10.1016/S0021-9258(18)96640-8. PMID 4223640.
- ^ McCarty RE (November 1992). “H+-ATPasen UND ATP-SYNTHASEN AUS DER SICHT EINES PFLANZENBIOCHEMISTEN”. Die Zeitschrift für experimentelle Biologie. 172 (Teil 1): 431–441. mach:10.1242/jeb.172.1.431. PMID 9874753.
- ^ Fernandez Moran H, Oda T, Blair PV, Green DE (Juli 1964). “Eine makromolekulare sich wiederholende Einheit der mitochondrialen Struktur und Funktion. Korrelierte elektronenmikroskopische und biochemische Studien isolierter Mitochondrien und submitochondrialer Partikel des Rinderherzmuskels”. Die Zeitschrift für Zellbiologie. 22 (1): 63–100. mach:10.1083/jcb.22.1.63. PMC 2106494. PMID 14195622.
- ^ “ATP-Synthase-Untereinheit alpha, mitochondrial”. UniProt. Das UniProt-Konsortium.
- ^ “ATP5F1A”. NCBI Genetics Home-Ressource.
- ^ Joshi T, Singh AK, Haratipour P, Farzaei MH (2019). „Targeting AMPK-Signalweg durch Naturstoffe zur Behandlung von Diabetes mellitus und seinen Komplikationen“. Zeitschrift für Zellphysiologie. 234 (10): 17212–17231. mach:10.1002/jcp.28528. PMID 30916407. S2CID 85533334.
- ^ “Körpergewichtsdaten für Atp5a1”. Wellcome Trust Sanger Institute.
- ^ “DEXA-Daten für Atp5a1”. Wellcome Trust Sanger Institute.
- ^ “Klinische Chemiedaten für Atp5a1”. Wellcome Trust Sanger Institute.
- ^ “Citrobacter Infektionsdaten für Atp5a1″. Wellcome Trust Sanger Institute.
- ^ ein B C D Gerdin AK (2010). „Das Sanger Mouse Genetics Program: High-Throughput-Charakterisierung von Knockout-Mäusen“. Acta Ophthalmologica. 88: 925–7. mach:10.1111/j.1755-3768.2010.4142.x. S2CID 85911512.
- ^ Maus-Ressourcenportal, Wellcome Trust Sanger Institute.
- ^ “Internationales Knockout-Maus-Konsortium”.
- ^ “Mausgenom-Informatik”.
- ^ Skarnes WC, Rosen B, West AP, Koutsourakis M, Bushell W, Iyer V, Mujica AO, Thomas M, Harrow J, Cox T, Jackson D, Severin J, Biggs P, Fu J, Nefedov M, de Jong PJ, Stewart AF, Bradley A (Juni 2011). „Eine bedingte Knockout-Ressource für die genomweite Untersuchung der Genfunktion von Mäusen“. Natur. 474 (7351): 337–42. mach:10.1038/natur10163. PMC 3572410. PMID 21677750.
- ^ Dolgin E (Juni 2011). “Mausbibliothek soll KO sein”. Natur. 474 (7351): 262–3. mach:10.1038/474262a. PMID 21677718.
- ^ Collins FS, Rossant J, Wurst W (Januar 2007). “Eine Maus aus allen Gründen”. Zelle. 128 (1): 9–13. mach:10.1016/j.cell.2006.12.018. PMID 17218247.
- ^ van der Weyden L, White JK, Adams DJ, Logan DW (Juni 2011). “Das Mausgenetik-Toolkit: Funktion und Mechanismus aufdecken”. Genombiologie. 12 (6): 224. doi:10.1186/de-2011-12-6-224. PMC 3218837. PMID 21722353.
Weiterlesen[edit]
- Dawson SJ, White LA (Mai 1992). „Behandlung von Haemophilus aphrophilus Endokarditis mit Ciprofloxacin“. Das Journal der Infektion. 24 (3): 317–20. mach:10.1016/S0163-4453(05)80037-4. PMID 1602151.
- Kovalyov LI, Shishkin SS, Efimochkin AS, Kovalyova MA, Ershova ES, Egorov TA, Musalyamov AK (Juli 1995). „Das Hauptproteinexpressionsprofil und die zweidimensionale Proteindatenbank des menschlichen Herzens“. Elektrophorese. 16 (7): 1160–9. mach:10.1002/elps.11501601192. PMID 7498159. S2CID 32209361.
- Abrahams JP, Leslie AG, Lutter R, Walker JE (August 1994). „Struktur bei 2.8 Eine Auflösung von F1-ATPase aus Rinderherzmitochondrien“. Natur. 370 (6491): 621–8. mach:10.1038/370621a0. PMID 8065448. S2CID 4275221.
- Akiyama S, Endo H, Inohara N, Ohta S, Kagawa Y (September 1994). „Genstruktur und Zelltyp-spezifische Expression der menschlichen ATP-Synthase-Alpha-Untereinheit“. Biochimica et Biophysica Acta (BBA) – Genstruktur und Expression. 1219 (1): 129–40. mach:10.1016/0167-4781(94)90255-0. PMID 8086450.
- Jabs EW, Thomas PJ, Bernstein M, Coss C, Ferreira GC, Pedersen PL (Mai 1994). „Chromosomale Lokalisierung von Genen, die für die Endschritte des oxidativen Stoffwechsels erforderlich sind: Alpha- und Gamma-Untereinheiten der ATP-Synthase und der Phosphatträger“. Humangenetik. 93 (5): 600-2. mach:10.1007/bf00202832. PMID 8168843. S2CID 39597611.
- Godbout R, Bisgrove DA, Honoré LH, Day RS (Januar 1993). „Amplifikation des Gens, das die Alpha-Untereinheit des mitochondrialen ATP-Synthase-Komplexes in einer menschlichen Retinoblastom-Zelllinie kodiert“. Gen. 123 (2): 195–201. mach:10.1016/0378-1119(93)90124-L. PMID 8428659.
- Godbout R, Pandita A, Beatty B, Bie W, Squire JA (1997). „Eine vergleichende genomische Hybridisierungsanalyse von Y79 und FISH-Kartierung zeigt, dass das amplifizierte menschliche mitochondriale ATP-Synthase-Alpha-Untereinheitsgen (ATP5A) auf Chromosom 18q12 -> q21 kartiert“. Zytogenetik und Zellgenetik. 77 (3–4): 253–6. mach:10.1159/000134588. PMID 9284928.
- Elston T, Wang H, Oster G (Januar 1998). „Energietransduktion in ATP-Synthase“. Natur. 391 (6666): 510–3. Bibcode:1998Natur.391..510E. mach:10.1038/35185. PMID 9461222. S2CID 4406161.
- Wang H, Oster G (November 1998). „Energietransduktion im F1-Motor der ATP-Synthase“. Natur. 396 (6708): 279–82. Bibcode:1998Natur.396..279W. mach:10.1038/24409. PMID 9834036. S2CID 4424498.
- Moser TL, Stack MS, Asplin I, Enghild JJ, Højrup P, Everitt L, Hubchak S, Schnaper HW, Pizzo SV (März 1999). „Angiostatin bindet ATP-Synthase auf der Oberfläche menschlicher Endothelzellen“. Proceedings of the National Academy of Sciences of the United States of America. 96 (6): 2811–6. Bibcode:1999PNAS…96.2811M. mach:10.1073/pnas.96.6.2811. PMC 15851. PMID 10077593.
- Moser TL, Kenan DJ, Ashley TA, Roy JA, Goodman MD, Misra UK, Cheek DJ, Pizzo SV (Juni 2001). “Endotheliale Zelloberfläche F1-F0 ATP-Synthase ist bei der ATP-Synthese aktiv und wird durch Angiostatin gehemmt”. Proceedings of the National Academy of Sciences of the United States of America. 98 (12): 6656–61. Bibcode:2001PNAS…98.6656M. mach:10.1073/pnas.131067798. PMC 34409. PMID 11381144.
- Wang ZG, White PS, Ackerman SH (August 2001). „Atp11p und Atp12p sind Assemblierungsfaktoren für die F(1)-ATPase in menschlichen Mitochondrien“. Die Zeitschrift für biologische Chemie. 276 (33): 30773–8. mach:10.1074/jbc.M104133200. PMID 11410595.
- SY Chang, SG Park, S. Kim, CY Kang (März 2002). “Interaktion der C-terminalen Domäne von p43 und der Alpha-Untereinheit der ATP-Synthase. Seine funktionelle Bedeutung für die Endothelzellproliferation”. Die Zeitschrift für biologische Chemie. 277 (10): 8388–94. mach:10.1074/jbc.M108792200. PMID 11741979.
- Sergeant N, Wattez A, Galván-valencia M, Ghestem A, David JP, Lemoine J, Sautiére PE, Dachary J, Mazat JP, Michalski JC, Velours J, Mena-López R, Delacourte A (2003). „Assoziation der ATP-Synthase-Alpha-Kette mit neurofibrillärer Degeneration bei der Alzheimer-Krankheit“. Neurowissenschaften. 117 (2): 293–303. mach:10.1016/S0306-4522(02)00747-9. PMID 12614671. S2CID 43991411.
- Bouwmeester T, Bauch A, Ruffner H, Angrand PO, Bergamini G, Croughton K, Cruciat C, Eberhard D, Gagneur J, Ghidelli S, Hopf C, Huhse B, Mangano R, Michon AM, Schirle M, Schlegl J, Schwab M , Stein MA, Bauer A, Casari G, Drewes G, Gavin AC, Jackson DB, Joberty G, Neubauer G, Rick J, Kuster B, Superti-Furga G (Februar 2004). „Eine physikalische und funktionelle Karte des menschlichen TNF-alpha/NF-kappa B-Signaltransduktionsweges“. Natur Zellbiologie. 6 (2): 97–105. mach:10.1038/ncb1086. PMID 14743216. S2CID 11683986.
- Kreuz RL (Januar 2004). “Molekulare Motoren: Drehen des ATP-Motors”. Natur. 427 (6973): 407–8. Bibcode:2004Natur.427..407C. mach:10.1038/427407b. PMID 14749816. S2CID 52819856.
- Jin J, Smith FD, Stark C, Wells CD, Fawcett JP, Kulkarni S, Metalnikov P, O’Donnell P, Taylor P, Taylor L, Zougman A, Woodgett JR, Langeberg LK, Scott JD, Pawson T (August 2004) . “Proteomische, funktionelle und domänenbasierte Analyse von in vivo 14-3-3-bindenden Proteinen, die an der Regulation des Zytoskeletts und der zellulären Organisation beteiligt sind”. Aktuelle Biologie. 14 (16): 1436–50. mach:10.1016/j.cub.2004.07.051. PMID 15324660.
Externe Links[edit]
Dieser Artikel enthält Text aus der National Library of Medicine der Vereinigten Staaten, der gemeinfrei ist.















Recent Comments