Arabinogalactan-Protein – Wikipedia
Arabinogalactan-Proteine (AGPs) sind hochglykosylierte Proteine (Glykoproteine), die in den Zellwänden von Pflanzen vorkommen. Jedes besteht aus einem Protein mit daran befestigten Zuckermolekülen (die mehr als 90% der Gesamtmasse ausmachen können). Sie sind Mitglieder der breiteren Klasse von Hydroxyprolin (Hyp)-reichen Zellwandglykoproteinen, einer großen und vielfältigen Gruppe glykosylierter Zellwandproteine.
AGPs wurden in einer Vielzahl von höheren Pflanzen in Samen, Wurzeln, Stängeln, Blättern und Blütenständen berichtet. AGPs machen nur einen kleinen Teil der Zellwand aus, normalerweise nicht mehr als 1 % der Trockenmasse der Primärwand. Sie wurden auch in Sekreten von Zellkulturmedien von Wurzel-, Blatt-, Endosperm- und Embryogeweben berichtet, und einige exsudatproduzierende Zelltypen, wie beispielsweise Griffelkanalzellen, sind in der Lage, große Mengen an AGPs zu produzieren. Sie sind an verschiedenen Aspekten des Pflanzenwachstums und der Pflanzenentwicklung beteiligt, darunter Wurzelverlängerung, somatische Embryogenese, Hormonreaktionen, Xylemdifferenzierung, Pollenschlauchwachstum und -lenkung, programmierter Zelltod, Zellexpansion, Salztoleranz, Wirt-Pathogen-Interaktionen und zelluläre Signalübertragung.
AGPs haben aufgrund ihrer hochkomplexen Strukturen und potentiellen Rollen bei der Signalisierung große Aufmerksamkeit auf sich gezogen. Darüber hinaus haben sie aufgrund ihrer chemisch-physikalischen Eigenschaften (Wasserrückhaltevermögen, Haftung und Emulgierung) industrielle und gesundheitliche Anwendungen.
Reihenfolge und Klassifizierung[edit]

Die Proteinkomponente von AGPs ist reich an den Aminosäuren Prolin (P), Alanin (A), Serin (S) und Threonin (T), auch bekannt als “PAST”. identifizieren sie.[1][2][3][4] AGPs sind intrinsisch ungeordnete Proteine, da sie einen hohen Anteil an ungeordneten Aminosäuren wie Prolin enthalten, die die Bildung stabiler gefalteter Strukturen stören. Charakteristisch für intrinsisch ungeordnete Proteine enthalten AGPs auch Wiederholungsmotive und posttranslationale Modifikationen.[2][5] Prolinreste im Proteinrückgrat können abhängig von den umgebenden Aminosäuren zu Hydroxyprolin (O) hydroxyliert werden. Die „Hyp-Kontiguitätshypothese“ [6][2][3] sagt voraus, dass wenn O nicht zusammenhängend auftritt, zum Beispiel die Sequenz ‘SOTO’, wie sie in AGPs vorkommt, dies als Signal für Ö-verknüpfte Glykosylierung von großen verzweigten Typ II Arabinogalactan (AG) Polysacchariden.[7] Sequenzen, die die AG-Glykosylierung (SO, TO, AO, VO) steuern, werden AGP-Glykomotive genannt.
Alle AGP-Proteinrückgrate enthalten mindestens 3 gruppierte AGP-Glykomotive und ein N-terminales Signalpeptid, das das Protein in das endoplasmatische Retikulum (ER) leitet, wo posttranslationale Modifikationen beginnen.[8] Die Prolyl-Hydroxylierung von P zu O wird von Prolyl-4-Hydroxylasen (P4Hs) erfüllt, die zur Familie der 2-Oxoglutarat-abhängigen Dioxygenasen gehören.[9]P4H wurde sowohl im ER- als auch im Golgi-Apparat identifiziert.[10] Die Zugabe des Glycosylphosphatidylinositol (GPI)-Ankers erfolgt in den meisten, aber nicht allen AGPs.[3][4]
Familien[edit]
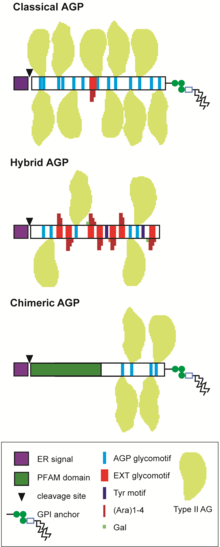
AGPs gehören zu großen Multigenfamilien und werden je nach vorhergesagter Proteinsequenz in mehrere Untergruppen unterteilt.[11][4][12][13][2][14] “Klassische” AGPs umfassen die GPI-AGPs, die aus einem Signalpeptid am N-Terminus, einer PAST-reichen Sequenz von 100-150 AS und einer hydrophoben Region am C-Terminus bestehen, die die Addition eines GPI-Ankers steuert; Nicht-GPI-AGPs, denen die C-terminale GPI-Signalsequenz fehlt, Lysin(K)-reiche AGPs, die eine K-reiche Region innerhalb des PAST-reichen Rückgrats enthalten und AG-Peptide, die ein kurzes PAST-reiches Rückgrat von 10-15 . aufweisen aa (Figur 2). Chimäre AGPs bestehen aus Proteinen, die eine AGP-Region und eine zusätzliche Region mit einer anerkannten Domäne der Proteinfamilie (Pfam) aufweisen. Zu den chimären AGPs gehören Fasziklin-ähnliche AGPs (FLAs), Phytocyanin-ähnliche AGPs (PAGs/PLAs, auch bekannt als Early-Nodulin-ähnliche Proteine, ENODLs) und Xylogen-ähnliche AGPs (XYLPs), die Lipidtransfer-ähnliche Domänen enthalten.[1] Mehrere andere mutmaßliche chimäre AGP-Klassen wurden identifiziert, die AG-Glykomotive umfassen, die mit Proteinkinase, leucinreicher Wiederholung, X8, FH2 und anderen Domänen der Proteinfamilie assoziiert sind.[2][15][16] Es gibt auch andere nicht-klassische AGPs, wie solche, die eine Cystein(C)-reiche Domäne, auch PAC-Domänen genannt, und/oder eine Histidin(H)-reiche Domäne enthalten.[17][18] sowie viele Hybrid-HRGPs, die für AGPs und andere HRGP-Mitglieder charakteristische Motive aufweisen, üblicherweise Extensin- und Tyr-Motive.[17][19][1][2][14] AGPs sind evolutionär alt und wurden in Grünalgen sowie Chromista und Glaucophyta identifiziert.[2][14][20] Es wird vermutet, dass Landpflanzen, die in der gesamten Pflanzenlinie vorkommen, die in Algen vorhandenen AGP-Proteinrückgrat-Gene geerbt und diversifiziert haben, um eine enorme Anzahl von AGP-Glykoformen zu erzeugen.
Struktur[edit]
Die Kohlenhydratanteile von AGPs sind reich an Arabinose und Galactan, aber auch andere Zucker wie L-Rhamnopyranose (L-RhaP), D-Mannopyranose (ManP), D-Xylopyranose (XylP), L-Fucose (Fuc), D-Glucopyranose (GlcP), D-Glucuronsäure (GlcA) und ihr 4-O-Methyl-Derivat und D-Galakturonsäure (GalA) und ihr 4-O-Methyl-Derivat.[21][22] Das in AGPs gefundene AG ist vom Typ II (Typ II AGs) – das heißt, ein Galactan-Rückgrat von (1-3)-verknüpfter β-D-Galactopyranose (GalP) Reste, mit Verzweigungen (zwischen einem und drei Resten lang) von (1,6)-verknüpftem β-D–GalP. In den meisten Fällen enden die Gal-Reste mit α-L-Arabinofuranose (AraF) Rückstände. Einige AGPs sind reich an Uronsäuren (GlcA), was zu einer geladenen Polysaccharideinheit führt, und andere haben kurze Oligosaccharide von AraF.[23] Spezifische Sätze von Hydroxyprolin-O-β-Galactosyltransferasen, β-1,3-Galactosyltransferasen, β-1,6-Galactosyltransferasen, α-Arabinosyltransferasen, β-Glucuronosyltransferasen, α-Rhamnosyltransferasen und α-Fucosyltransferasen sind für die Synthese dieser Komplexe verantwortlich Strukturen.[24]
Eines der Merkmale von Typ-II-AGs, insbesondere der (1,3)-verknüpften β-D-GalP Reste, ist ihre Fähigkeit, an die Yariv-Phenylglykoside zu binden. Yariv-Phenylglycoside werden häufig als zytochemische Reagenzien zur Störung der molekularen Funktionen von AGPs sowie zum Nachweis, zur Quantifizierung, Reinigung und Färbung von AGPs verwendet.[21] Kürzlich wurde berichtet, dass keine Interaktion mit Yariv für β-1,6-Galacto-Oligosaccharide jeglicher Länge nachgewiesen wurde.[25] Es wurde gefolgert, dass Yariv-Phenylglycoside spezifische Bindungsreagenzien für β-1,3-Galaktanketten sind, die länger als fünf Reste sind. Sieben Reste und länger reichen für die Vernetzung aus, was zur Ausfällung der Glykane mit den Yariv-Phenylglykosiden führt, die bei klassischen AGPs beobachtet werden, die an β-Yariv-Farbstoffe binden. Die gleichen Ergebnisse wurden beobachtet, wenn in AGPs mindestens 5–7 β-1,3-verknüpfte Gal-Einheiten zu benötigen scheinen, um mit dem Yariv-Reagens Aggregate zu bilden.[26]
Biosynthese[edit]
Nach der Translation sind die AGP-Proteinrückgrate hochgradig mit komplexen Kohlenhydraten dekoriert, hauptsächlich mit Typ-II-AG-Polysacchariden.[27] Die Biosynthese des reifen AGP beinhaltet die Spaltung des Signalpeptids am N-Terminus, die Hydroxylierung an den P-Resten und die anschließende Glykosylierung und in vielen Fällen die Zugabe eines GPI-Ankers.
Verarbeitung und Transport[edit]
Es wird vorgeschlagen, dass die Glykosylierung des AGP-Rückgrats im ER mit der Zugabe von erster Gal durch . beginnt Ö-Galactosyltransferase, die überwiegend in ER-Fraktionen lokalisiert ist.[28] Die Kettenverlängerung erfolgt dann hauptsächlich im GA.[29] Bei den AGPs, die einen GPI-Anker enthalten, erfolgt die Addition während der cotranslationalen Migration in das ER.
Arabinogalactan-Seitenketten[edit]
Die Struktur der AG-Glykane besteht aus einem Rückgrat von β-1,3 verknüpfte Galactose (Gal), mit Seitenketten von β-1,6 verknüpfte Gal und haben endständige Reste von Arabinose (Ara), Rhamnose (Rha), Gal, Fucose (Fuc) und Glucuronsäure (GlcA). Diese AG-Glycan-Einheiten werden durch Glycosyltransferasen (GTs) aufgebaut.[30]Ö-Glykosylierung von AGPs wird durch die Wirkung von Hyp-Ö-Galactosyltransferasen (Hyp-Ö-GalTs), die dem Protein die erste Gal hinzufügen. Die komplexen Glykanstrukturen werden dann durch eine Reihe von Glykosyltransferasen ausgearbeitet, von denen die meisten biochemisch nicht charakterisiert sind. Die GT31-Familie ist eine der Familien, die an der Biosynthese des AGP-Glykanrückgrats beteiligt sind.[31][32] Zahlreiche Mitglieder der GT31-Familie wurden mit Hyp-Ö-GALT-Aktivität[33][34] und der Kern β-(1,3)-Galactan-Rückgrat wird wahrscheinlich auch von der GT31-Familie synthetisiert.[32] Mitglieder der GT14-Familie sind am Hinzufügen beteiligt β-(1,6)- und β-(1,3)-Galaktane zu AGPs.[35][36] Bei Arabidopsis wird vorgeschlagen, dass terminale Zucker wie Fucose durch AtFUT4 (eine Fucosyltransferase) und AtFUT6 in der GT37-Familie hinzugefügt werden [37][38] und der terminale GlcA-Einbau kann durch die GT14-Familie katalysiert werden.[35][39] Es müssen noch eine Reihe von GTs identifiziert werden, beispielsweise diejenigen, die für das Terminal Rha verantwortlich sind.
GPI-Anker[edit]
Die bioinformatische Analyse sagt das Hinzufügen eines GPI-Ankers auf vielen AGPs voraus.[4] Die frühe Synthese der GPI-Einheit erfolgt auf der zytoplasmatischen Oberfläche des ER und der anschließende Zusammenbau findet im Lumen des ER statt. Dazu gehört der Zusammenbau von Trimannose (Man), Galactose, nicht-N-acetyliertem Glucosamin (GlcN) und Ethanolaminphosphat, um die reife GPI-Einheit zu bilden.[40][41] AGPs durchlaufen eine GPI-Ankeraddition, während sie cotranslational in das ER wandern, und diese beiden Prozesse konvergieren schließlich. Anschließend spaltet ein Transamidase-Komplex gleichzeitig das Core-Protein am C-Terminus, wenn er die ω-Schnittstelle erkennt und überträgt den vollständig assemblierten GPI-Anker auf den Aminosäurerest am C-Terminus des Proteins. Diese Ereignisse treten vor der Prolylhydroxylierung und Glykosylierung auf.[42][10] Die Kernglykanstruktur von GPI-Ankern ist Man-α-1,2-Mann-α-1,6-Mann-α-1,4-GlcN-Inositol (Mann: Mannose, GlcN: Glucosaminyl), das in vielen Eukaryoten konserviert ist.[41][43][44][40][10][45] Die einzige bisher charakterisierte pflanzliche GPI-Ankerstruktur ist die GPI-verankerte AGP von Pyrus Communis Suspensions-kultivierte Zellen.[40] Dies zeigte eine teilweise modifizierte Glykaneinheit im Vergleich zu zuvor charakterisierten GPI-Ankern, da sie enthalten β-1,4-Gal. Es wird vorgeschlagen, dass der GPI-Ankersynthese- und Proteinassemblierungsweg in Säugetieren und Pflanzen konserviert ist.[10] Die Integration eines GPI-Ankers ermöglicht die Anheftung des Proteins an die Membran des ER, das zum GA übergeht, was zur Sekretion an das äußere Segel der wandzugewandten Plasmamembran führt.[46] Wie von Oxley und Bacic vorgeschlagen,[40] die GPI-verankerten AGPs werden wahrscheinlich durch Spaltung durch einige Phospholipasen (PLs) (C oder D) freigesetzt und in das extrazelluläre Kompartiment sezerniert.
Funktionell charakterisierte Gene, die an der AGP-Glykosylierung beteiligt sind[edit]
Bioinformatische Analysen unter Verwendung von Säugetier-β-1,3-Galactosyltransferase (GalT)-Sequenzen als Matrizen legten eine Beteiligung der kohlenhydrat-aktiven Enzyme (CAZy) Glycosyltransferase (GT) 31-Familie an der Synthese der Galactanketten des AG-Rückgrats nahe.[47] Mitglieder der GT31-Familie wurden in 11 Kladen gruppiert, wobei vier Kladen pflanzenspezifisch sind: Kladen 1, 7, 10 und 11. Die Domänen und Motive der Kladen 1 und 11 sind nicht genau definiert; während die Kladen 7 und 10 Domänenähnlichkeiten mit Proteinen bekannter GalT-Funktion in Säugetiersystemen aufweisen.[47] Clade-7-Proteine enthalten sowohl GalT- als auch Galectin-Domänen, während Clade-10-Proteine eine GalT-spezifische Domäne enthalten.[48] Es wird vorgeschlagen, dass die Galectin-Domäne dem GalT ermöglicht, an den ersten Gal-Rest auf dem Polypeptid-Rückgrat von AGPs zu binden; so wird die Position nachfolgender Gal-Reste auf dem Proteinrückgrat bestimmt, ähnlich der Aktivität von Proteinen, die eine menschliche Galectindomäne enthalten.[47]
Acht Enzyme der GT31-Familie zeigten die Fähigkeit, den ersten Gal-Rest auf Hyp-Resten in AGP-Kernproteinen zu platzieren. Diese Enzyme heißen GALT2, GALT3, GALT4, GALT5, GALT6,[49] die Mitglieder von Clade 7 sind, und HPGT1, HPGT2 und HPGT3,[50] die Mitglieder von Clade 10 sind. Vorläufige Studien zur Enzymsubstratspezifität zeigten, dass ein anderes GT31-Clade-10-Enzym, At1g77810, β-1,3-GalT-Aktivität aufwies.[47] Ein GT31 Clade 10-Gen, KNS4/UPEX1, kodiert für ein β-1,3-GalT, das in der Lage ist, β-1,3-Gal-Bindungen zu synthetisieren, die in Typ-II-AGs gefunden werden, die in AGPs und/oder pektischem Rhamnogalacturonan I (RG-I) vorhanden sind.[51] Ein weiteres Mitglied der GT31 Clade 10 mit dem Namen GALT31A, kodiert ein β-1,6-GalT, wenn heterolog exprimiert in E coli und Nicotiana Benthamiana und verlängerte β-1,6-Galactan-Seitenketten von AGP-Glykanen.[52] Es wurde identifiziert, dass GALT29A, ein Mitglied der GT29-Familie, mit GALT31A koexprimiert wird und kooperativ wirkt und Komplexe bildet.[53]
Von drei Mitgliedern von GT14 namens GlcAT14A, GlcAT14B und GlcAT14C wurde berichtet, dass sie GlcA sowohl an β-1,6- als auch an β-1,3-Gal-Ketten in an . hinzufügen in vitro Enzymassay nach heterologer Expression in Pichia pastoris.[54] Zwei α-Fucosyltransferase-Gene, FUT4 und FUT6, die beide zur GT37-Familie gehören, kodieren für Enzyme, die α-1,2-Fucose-Reste an AGPs anfügen.[55][56] Sie scheinen teilweise redundant zu sein, da sie etwas andere AGP-Substratspezifitäten aufweisen.[55] Ein Mitglied der GT77-Familie, REDUCED ARABINOSE YARIV (RAY1), ist eine β-Arabinosyltransferase, die ein β-Ara . hinzufügtF zu Methyl-β-Gal eines Yariv-präzipitierbaren Wandpolymers.[57] Es wird erwartet, dass weitere Forschungen andere Gene, die an der AGP-Glykosylierung beteiligt sind, und ihre Interaktionen mit anderen Pflanzenzellwandkomponenten funktionell identifizieren.
Biologische Rollen von AGP[edit]
Zu den menschlichen Anwendungen von AGPs gehört die Verwendung von Gummi arabicum in der Lebensmittel- und Pharmaindustrie aufgrund der natürlichen Eigenschaften beim Verdicken und Emulgieren.[58][59] AGPs in Getreidekörnern haben potenzielle Anwendungen in der Biofortifikation,[60] als Ballaststoffquelle zur Unterstützung von Darmbakterien[61] und Schutzmittel gegen Ethanoltoxizität.[62]
AGPs werden in einer Vielzahl von Pflanzengeweben gefunden, in Sekreten von Zellkulturmedien von Wurzel-, Blatt-, Endosperm- und Embryogeweben und einigen exsudatproduzierenden Zelltypen, wie beispielsweise Griffelkanalzellen.[19][63] Es wurde gezeigt, dass AGPs viele Aspekte des Pflanzenwachstums und der Pflanzenentwicklung regulieren, einschließlich der männlich-weiblichen Erkennung in Fortpflanzungsorganen, der Zellteilung und -differenzierung in der Embryonal- und Postembryoentwicklung, der Entwicklung der Samenschleimzellwand, der Wurzelsalztoleranz und der Wurzel-Mikroben-Interaktion.[64][10][65] Diese Studien legen nahe, dass sie multifunktional sind, ähnlich dem, was in Säugetier-Proteoglykanen/Glykoproteinen gefunden wird.[66][67][68] Herkömmliche Methoden zum Studium der Funktionen von AGPs umfassen die Verwendung von β-Glycosyl (normalerweise Glucosyl) Yariv-Reagenzien und monoklonale Antikörper (mAbs). β-Glycosyl Yariv-Reagenzien sind synthetische Phenylazoglycosid-Sonden, die spezifisch, aber nicht kovalent an AGPs binden und zur Ausfällung von AGPs aus Lösung verwendet werden können.[69] Sie werden auch häufig als histochemische Färbemittel verwendet, um die Lage und Verteilung von AGPs zu untersuchen.[70][71] Eine Reihe von Studien hat gezeigt, dass die Zugabe von β-Yariv-Reagenzien für das Pflanzenwachstumsmedium können das Keimlingswachstum, die Zellverlängerung, die somatische Embryogenese und die Ansammlung frischer Zellwandmassen hemmen.[72][73][74] Die Verwendung von mAbs, die spezifisch an Kohlenhydrat-Epitope von AGPs binden, wurde auch verwendet, um Funktionen basierend auf der Lage und dem Muster der AGP-Epitope abzuleiten.[75] Üblicherweise verwendete mAb gegen AGPs umfassen CCRC-M7, LM2, JIM8, JIM13 und JIM14.[76]
Die Funktion einzelner AGPs wurde weitgehend durch Studien an Mutanten abgeleitet. Zum Beispiel die Arabidopsis wurzelspezifisch Bei AGP30 erwies sich als erforderlich für in vitro Wurzelregeneration, was auf eine Funktion bei der Regeneration der Wurzel durch Modulation der Phytohormonaktivität hindeutet.[77] Studium von agp6 und agp11 Mutanten in Arabidopsis haben die Bedeutung dieser AGPs zur Verhinderung der unkontrollierten Bildung der Pollenkörner und für das normale Wachstum des Pollenschlauchs gezeigt.[78][79] Die funktionellen Mechanismen von AGPs bei der Zellsignalisierung sind nicht gut verstanden. Ein vorgeschlagenes Modell legt nahe, dass AGPs interagieren und die Freisetzung von Calcium aus AG-Glykan (über GlcA-Reste) kontrollieren können, um nachgeschaltete Signalwege auszulösen, die durch Calcium vermittelt werden.[80][81][82] Ein weiterer möglicher Mechanismus, der weitgehend auf der Untersuchung von FLAs basiert, legt nahe, dass die Kombination von Fascicilin-Domäne und AG-Glykanen die Zell-Zell-Adhäsion vermitteln kann.[83][84]
| Biologische Rolle | AGP [a][b] | Standorte) | Funktion(en) | Verweise |
|---|---|---|---|---|
| Embryogenese | GhPLA1 | Somatische Embryonen | Förderung der somatischen Embryogenese | [85] |
| DcAGPs | Somatische Embryonen | Förderung der somatischen Embryogenese | [86] | |
| AtAGPs | Embryonen | Embryonenentwicklung und -differenzierung | [87] | |
| NtAGPs | Embryonen | Embryonenentwicklung | [88] | |
| BgAGPs | Somatische Embryonen | Entwicklungsrate und Morphologie des somatischen Embryos | [89] | |
| BnAGPs | Embryonen | Embryonenentwicklung | [21] | |
| MaAGPs | Somatische Embryonen | Förderung der somatischen Embryogenese | [90] | |
| PSAGPs | Förderung der somatischen Embryogenese | [91] | ||
| FsAGPs | Embryonen | Aufbau und Stabilität der Zellwand | [20] | |
| VcALGAL-CAM | Embryonen | Embryozelladhäsion | [83] | |
| VcISG | Embryonen | Inversion des Embryos | [92] | |
| Reproduktion | AtAGP4 (JAGGER) | Stempel | Verstopfung des Pollenschlauchs | [93] |
| Bei AGP6, bei AGP11 | Staubblatt, Pollenkorn und Pollenschlauch | Pollenkornentwicklung und Pollenschlauchwachstum | [94][95] | |
| Bei AGP18 | Samenanlage | Megasporenauswahl | [96][97] | |
| AtFLA3 | Pollenkorn und Pollenschlauch | Entwicklung von Mikrosporen | [98] | |
| BeiENODL11-15 | Mikropylar | Pollenschlauchempfang | [99][100] | |
| BcmMF8 | Pollenkorn und Pollenschlauch | Pollenwandentwicklung und Pollenschlauchwachstum | [101] | |
| BcmMF18 | Pollenkorn | Pollenkornentwicklung, Intinenbildung | [102] | |
| NtTTS | Stempel | Pollenschlauchwachstum und -führung | [103] | |
| Np/Na120kD | Stempel | S-spezifische Pollenabstoßung (Selbstunverträglichkeit) | [104] | |
| OsMTR1 | Männliche Fortpflanzungszellen | Staubbeutelentwicklung und Pollenfruchtbarkeit | [105] | |
| Pflanzenentwicklung | Bei AGP19 | Stängel, Blüte, Wurzel und Blatt | Zellteilung und -expansion, Blattentwicklung und -vermehrung | [106] |
| Bei AGP57C | Rosettenblatt, Silique, Samen, Blüte und Triebspitze des Blütenstandsstiels | Pflege der Zellwandstruktur | [107] | |
| AtFLA1 | Spaltöffnungen, Trichom, Blattgefäße, Primärwurzelspitze und Seitenwurzel | Seitliche Wurzelentwicklung und Sprossregeneration | [108] | |
| AtFLA4 (SOS5) | Blume, Blatt, Stängel, Wurzel, Silique | Wurzelsalz-Stresstoleranz; Haftung von Samenschleim | [109][110][111][112][113] | |
| PpAGP1 | Apikale Zellen | Apikale Zellexpansion | [114] | |
| Bei AGP30 | Wurzel | Wurzelregeneration und Samenkeimung | [47] | |
| BcrFLA1 | Wurzel | Verlängerung der Wurzelhaare | [115] | |
| Sekundärwandentwicklung | BeiFLA11, beiFLA12 | Stamm und Zweig | Sekundäre Zellwandsynthese/-strukturierung | [116] |
| AtXYP1, AtXYP2 | Zellwände differenzierender Trachealelemente | Entwicklung und Musterung von Gefäßgewebe | [117] | |
| GhAGP4 | Baumwollfaser | Baumwollfaserinitiierung und -dehnung | [98] | |
| GhFLA1 | Baumwollfaser | Faserinitiierung und -dehnung | [52] | |
| PtFLA6 | Stamm-Xylem-Faser | Sekundäre Zellwandsynthese/-strukturierung | [118] | |
| Verteidigung | SlattAGP | Ort des Parasitenbefalls | Fördert das Anhaften von Parasiten | [119] |
| Pflanzen-Mikroben-Interaktion | Bei AGP17 | Wurzel | Agrobacterium tumefaciens Wurzeltransformation | [120] |
- ^ Gh: Gossypium hirsutum, Dc: Daucus carota, Bei: Arabidopsis thaliana, Nt: Nicotiana tabacum, Bg: Bactris gasipaes, Mrd.: Brassica napus, Ma: Musa spp. AAA, PS: Pelargonium sidoides, Fs: Fucus serratus, Vc: Volvox-Kartei, Bcm: Brassica campestris, Np: Nicotiana plumbaginifolia, N / A: Nicotiana alata, Os: Oryza sativa, pp: Physcomitrella patens, Bkr: Brassica carinata, Punkt: Populus trichocarpa, Sl: Solanum lycopersicum.
- ^ PLA: Phytocyanin-ähnliches AGP. ALGAL-CAM: Algenzelladhäsionsmolekül. ISG: Inversionsspezifisches Glykoprotein. FLA: fasziklin wie AGP. ENODL: wie ein uraltes Knötchen. MF: männliche Fruchtbarkeit. TTS: gewebespezifisch übertragen. MTR: Mikrosporen- und Tapetumregulator. SOS: Salz überempfindlich. XYP: Xylogenprotein. attAGP: Anhang AGP.
Die Funktionen von AGPs in Pflanzenwachstums- und Entwicklungsprozessen hängen stark von der unglaublichen Vielfalt ihrer Glykan- und Proteinrückgrat-Einheiten ab.[121] Insbesondere die AG-Polysaccharide sind am wahrscheinlichsten an der Entwicklung beteiligt.[122] Die meisten biologischen Rollen von AGPs wurden durch T-DNA-Insertionsmutanten-Charakterisierung von Genen oder Enzymen identifiziert, die an der AGP-Glykosylierung beteiligt sind, hauptsächlich in Arabidopsis thaliana. Die galt2-6 einzelne Mutanten zeigten unter normalen Wachstumsbedingungen einige physiologische Phänotypen, einschließlich reduzierter Wurzelhaarlänge und -dichte, reduzierter Samenansatz, reduzierter anhaftender Samenhüllenschleim und vorzeitiger Alterung.[123] Jedoch, galt2galt5 Doppelmutanten zeigten schwerere und pleiotropere physiologische Phänotypen als die Einzelmutanten in Bezug auf Wurzelhaarlänge und -dichte und Samenhüllenschleim.[123] Ähnlich, hpgt1hpgt2hpgt3 Dreifachmutanten zeigten mehrere pleiotrope Phänotypen, darunter längere Seitenwurzeln, erhöhte Wurzelhaarlänge und -dichte, dickere Wurzeln, kleinere Rosettenblätter, kürzere Blattstiele, kürzere Blütenstandsstiele, reduzierte Fruchtbarkeit und kürzere Schoten.[50] Im Falle des GALT31AEs wurde festgestellt, dass es für die Entwicklung des Embryos unerlässlich ist Arabidopsis. Eine T-DNA-Insertion im 9. Exon von GALT31A führte zu Embryoletalität dieser mutierten Linie.[52] Inzwischen Knockout-Mutanten für KNS4/UPEX1 haben kollabierte Pollenkörner und eine abnorme Pollen-Exine-Struktur und -Morphologie.[124] Zusätzlich, kns4 einzelne Mutanten zeigten eine reduzierte Fertilität, was bestätigt, dass KNS4/UPEX1 ist entscheidend für die Lebensfähigkeit und Entwicklung von Pollen.[51] Knockout-Mutanten für FUT4 und FUT6 zeigte unter Salzbedingungen eine starke Hemmung des Wurzelwachstums[56] während Knockout-Mutanten für GlcAT14A, GlcAT14B, und GlcAT14C zeigten erhöhte Zellverlängerungsraten in dunkel gewachsenen Hypokotylen und hell gewachsenen Wurzeln während des Sämlingswachstums.[125] Im Falle des ray1 mutierte Sämlinge, die auf vertikalen Platten gewachsen sind, die Länge der Primärwurzel wurde beeinflusst durch RAY1 Mutation. Darüber hinaus ist die Primärwurzel von ray1 Mutanten wuchsen langsamer als Wildtyp Arabidopsis.[57] Zusammengenommen liefern diese Studien den Beweis, dass die richtige Glykosylierung von AGPs für die AGP-Funktion beim Pflanzenwachstum und bei der Entwicklung wichtig ist.
Verwendung durch den Menschen[edit]
Zu den menschlichen Anwendungen von AGPs gehört die Verwendung von Gummi arabicum in der Lebensmittel- und Pharmaindustrie aufgrund der natürlichen Eigenschaften beim Verdicken und Emulgieren.[58][59] AGPs in Getreidekörnern haben potenzielle Anwendungen in der Biofortifikation,[60] als Ballaststoffquelle zur Unterstützung von Darmbakterien[61] und Schutzmittel gegen Ethanoltoxizität.[62]
Siehe auch[edit]
Verweise[edit]
![]() Dieser Artikel wurde aus der folgenden Quelle unter a . angepasst CC BY 4.0 Lizenz (2021) (Gutachterberichte):
Dieser Artikel wurde aus der folgenden Quelle unter a . angepasst CC BY 4.0 Lizenz (2021) (Gutachterberichte):
Yingxuan-Ma; Kim Johnson (15. Januar 2021). “Arabinogalactan-Proteine” (PDF). WikiJournal of Science. 4 (1): 2. doi:10.15347/WJS/2021.002. ISSN 2470-6345. Wikidata Q99557488.
- ^ ein B C Showalter, Allan M.; Keppler, Brian; Lichtenberg, Jens; Gu, Dazhang; Welch, Lonnie R. (2010-04-15). “Ein bioinformatischer Ansatz zur Identifizierung, Klassifizierung und Analyse von Hydroxyprolin-reichen Glykoproteinen”. Pflanzenphysiologie. 153 (2): 485–513. mach:10.1104/pp.110.156554. ISSN 0032-0889. PMC 2879790. PMID 20395450.
- ^ ein B C D e F g Johnson, Kim L.; Cassin, Andrew M.; Lonsdale, Andrew; Bacic, Antony; Döblin, Monika S.; Schultz, Carolyn J. (2017-04-26). “Pipeline zur Identifizierung von Hydroxyprolin-reichen Glykoproteinen”. Pflanzenphysiologie. 174 (2): 886–903. mach:10.1104/S.17.00294. ISSN 0032-0889. PMC 5462032. PMID 28446635.
- ^ ein B C Johnson, Kim L.; Cassin, Andrew M.; Lonsdale, Andrew; Wong, Gane Ka-Shu; Soltis, Douglas E.; Miles, Nicholas W.; Melkonian, Michael; Melkonian, Barbara; Deyholos, Michael K. (2017-04-26). “Einblicke in die Evolution von Hydroxyprolin-reichen Glykoproteinen aus 1000 Pflanzentranskriptomen”. Pflanzenphysiologie. 174 (2): 904–921. mach:10.1104/S.17.00295. ISSN 0032-0889. PMC 5462033. PMID 28446636.
- ^ ein B C D Schultz, Carolyn J.; Rumsewicz, Michael P.; Johnson, Kim L.; Jones, Brian J.; Gaspar, Yolanda M.; Bacic, Antony (2002-08-01). „Mit genomischen Ressourcen die Forschungsrichtungen lenken. Die Arabinogalactan-Protein-Genfamilie als Testfall“. Pflanzenphysiologie. 129 (4): 1448–1463. mach:10.1104/pp.003459. ISSN 0032-0889. PMC 166734. PMID 12177459.
- ^ Shafee, Thomas; Bacic, Antony; Johnson, Kim (2020-08-01). Wilke, Claus (Hrsg.). “Evolution sequenz-diverser ungeordneter Regionen in einer Proteinfamilie: Ordnung im Chaos”. Molekularbiologie und Evolution. 37 (8): 2155–2172. mach:10.1093/molbev/msaa096. ISSN 0737-4038. PMID 32359163.
- ^ Kieliszewski, Marcia J.; Lamport, Derek TA (Februar 1994). “Extensin: repetitive Motive, funktionelle Stellen, posttranslationale Codes und Phylogenie”. Das Pflanzenjournal. 5 (2): 157–172. mach:10.1046/j.1365-313x.1994.05020157.x. ISSN 0960-7412. PMID 8148875.
- ^ Tan, Li; Leykam, Joseph F.; Kieliszewski, Marcia J. (2003-06-12). „Glykosylierungsmotive, die die Addition von Arabinogalactan an Arabinogalactan-Proteine steuern“. Pflanzenphysiologie. 132 (3): 1362–1369. mach:10.1104/pp.103.021766. ISSN 0032-0889. PMC 167076. PMID 12857818.
- ^ Schultz, Carolyn; Gilson, Paul; Oxley, David; Du, Joelian; Bacic, Antony (November 1998). “GPI-Anker auf Arabinogalactan-Proteinen: Implikationen für die Signalübertragung in Pflanzen”. Trends in der Pflanzenwissenschaft. 3 (11): 426–431. mach:10.1016/s1360-1385(98)01328-4. ISSN 1360-1385.
- ^ Koski, M. Kristian; Hieta, Reija; Böllner, Claudia; Kivirikko, Kari I.; Myllyharju, Johanna; Wierenga, Rik K. (2007-12-21). “Das aktive Zentrum einer Algen-Prolyl-4-Hydroxylase hat eine große strukturelle Plastizität”. Die Zeitschrift für biologische Chemie. 282 (51): 37112–37123. mach:10.1074/jbc.M706554200. ISSN 0021-9258. PMID 17940281.
- ^ ein B C D e Ellis, Miriam; Egelund, Jack; Schultz, Carolyn J.; Bacic, Antony (2010-04-13). “Arabinogalactan-Proteine: Schlüsselregulatoren an der Zelloberfläche?”. Pflanzenphysiologie. 153 (2): 403–419. mach:10.1104/pp.110.156000. ISSN 0032-0889. PMC 2879789. PMID 20388666.
- ^ Showalter, AM (September 2001). “Arabinogalactan-Proteine: Struktur, Expression und Funktion”. Zelluläre und molekulare Biowissenschaften. 58 (10): 1399–1417. mach:10.1007/pl00000784. ISSN 1420-682X. PMID 11693522. S2CID 206858189.
- ^ Gaspar, Yolanda-Maria; Nam, Jaesung; Schultz, Carolyn Jane; Lee, Lan-Ying; Gilson, Paul R.; Gelvin, Stanton B.; Bacic, Antony (2004-07-30). “Charakterisierung der Arabidopsis-Lysin-reichen Arabinogalactan-Protein-AtAGP17-Mutante (rat1), die zu einer verringerten Effizienz der Agrobakterium-Transformation führt”. Pflanzenphysiologie. 135 (4): 2162–2171. mach:10.1104/pp.104.045542. ISSN 0032-0889. PMC 520787. PMID 15286287.
- ^ Huang, Geng-Qing; Gong, Si-Ying; Xu, Wen-Liang; Li, Wen; Li, Peng; Zhang, Chao-Jun; Li, Deng-Di; Zheng, Yong; Li, Fu-Guang (2013-01-24). “Ein fasziklinähnliches Arabinogalactan-Protein, GhFLA1, ist an der Faserinitiierung und -dehnung von Baumwolle beteiligt”. Pflanzenphysiologie. 161 (3): 1278-1290. mach:10.1104/pp.112.203760. ISSN 0032-0889. PMC 3585596. PMID 23349362.
- ^ ein B C Johnson, Kim L.; Cassin, Andrew M.; Lonsdale, Andrew; Wong, Gane Ka-Shu; Soltis, Douglas E.; Miles, Nicholas W.; Melkonian, Michael; Melkonian, Barbara; Deyholos, Michael K. (2017-04-26). “Einblicke in die Evolution von Hydroxyprolin-reichen Glykoproteinen aus 1000 Pflanzentranskriptomen”. Pflanzenphysiologie. 174 (2): 904–921. mach:10.1104/S.17.00295. ISSN 0032-0889. PMC 5462033. PMID 28446636.
- ^ Dragićević, Mailand B; Paunović, Danijela M; Bogdanović, Milica D; Todorović, Sladjana I; Simonović, Ana D (01.01.2020). “ragp: Pipeline zum Abbau pflanzlicher Hydroxyprolin-reicher Glykoproteine mit Umsetzung in R”. Glykobiologie. 30 (1): 19–35. mach:10.1093/glycob/cwz072. ISSN 1460-2423. PMID 31508799.
- ^ Pfeifer, Lukas; Shafee, Thomas; Johnson, Kim L.; Bacic, Antony; Classen, Birgit (Dezember 2020). “Arabinogalactan-Proteine von Zostera marina L. enthalten einzigartige Glykanstrukturen und geben Einblick in Anpassungsprozesse an salzhaltige Umgebungen”. Wissenschaftliche Berichte. 10 (1): 8232. Bibcode:2020NatSR..10.8232P. mach:10.1038/s41598-020-65135-5. ISSN 2045-2322. PMC 7237498. PMID 32427862.
- ^ ein B Baldwin, Timothy C.; Domingo, Concha; Schindler, Thomas; Seetharaman, Gouri; Stacey, Nicola; Roberts, Keith (2001). „DcAGP1, ein sezerniertes Arabinogalactan-Protein, ist mit einer Familie basischer prolinreicher Proteine verwandt“. Molekularbiologie der Pflanzen. 45 (4): 421–435. mach:10.1023/A:1010637426934. PMID 11352461. S2CID 8322072.
- ^ Nguyen-Kim, Huan; San Clemente, Helene; Laimer, Josef; Lackner, Peter; Gadermaier, Gabriele; Dunand, Christophe; Jamet, Elisabeth (2020-04-03). “Die Zellwand PAC (Prolin-Rich, Arabinogalactan Proteins, Conserved Cysteines) Domänenproteine sind in der grünen Linie konserviert”. Internationale Zeitschrift für Molekularwissenschaften. 21 (7): 2488. doi:10.3390/ijms21072488. ISSN 1422-0067. PMC 7177597. PMID 32260156.
- ^ ein B Gaspar, Y.; Johnson, KL; McKenna, JA; Bacic, A.; Schultz, CJ (September 2001). “Die komplexen Strukturen von Arabinogalactan-Proteinen und der Weg zum Verständnis der Funktion”. Molekularbiologie der Pflanzen. 47 (1–2): 161–176. mach:10.1023/A:1010683432529. ISSN 0167-4412. PMID 11554470. S2CID 19541545.
- ^ ein B Hervé, Cécile; Siméon, Amandine; Marmelade, Murielle; Cassin, Andrew; Johnson, Kim L.; Salmeán, Armando A.; Willats, William GT; Döblin, Monika S.; Bacic, Antony (2015-12-15). “Arabinogalactan-Proteine haben tiefe Wurzeln in Eukaryoten: Identifizierung von Genen und Epitopen in Braunalgen und ihre Rolle bei der Entwicklung von Fucus serratusembryos”. Neuer Phytologe. 209 (4): 1428–1441. mach:10.1111/nph.13786. ISSN 0028-646X. PMID 26667994.
- ^ ein B C Fincher, GB; Stein, BA; Clarke, AE (1983). „Arabinogalactan-Proteine: Struktur, Biosynthese und Funktion“. Jährliche Überprüfung der Pflanzenphysiologie. 34 (1): 47–70. mach:10.1146/annurev.pp.34.060183.000403.
- ^ Inaba, Miho; Maruyama, Takuma; Yoshimi, Yoshihisa; Kotake, Toshihisa; Matsuoka, Koji; Koyama, Tetsuo; Tryfona, Theodora; Dupree, Paul; Tsumuraya, Yoichi (2015-10-13). “l-Fucose-haltiges Arabinogalactan-Protein in Rettichblättern”. Kohlenhydratforschung. 415: 1–11. mach:10.1016/j.carres.2015.07.002. ISSN 0008-6215. PMC 4610949. PMID 26267887.
- ^ Du, er; Clarke, Adrienne E.; Bacic, Antony (1996-11-01). „Arabinogalactan-Proteine: eine Klasse von extrazellulären Matrix-Proteoglykanen, die an Pflanzenwachstum und -entwicklung beteiligt sind“. Trends in der Zellbiologie. 6 (11): 411–414. mach:10.1016/S0962-8924(96)20036-4. ISSN 0962-8924. PMID 15157509.
- ^ Showalter, Allan M.; Basu, Debarati (2016). “Extensin und Arabinogalactan-Protein-Biosynthese: Glykosyltransferasen, Forschungsherausforderungen und Biosensoren”. Grenzen in der Pflanzenwissenschaft. 7: 814. doi:10.3389/fpls.2016.00814. ISSN 1664-462X. PMC 4908140. PMID 27379116.
- ^ Kitazawa, Kiminari; Tryfona, Theodora; Yoshimi, Yoshihisa; Hayashi, Yoshihiro; Kawauchi, Susumu; Antonow, Lüdmil; Tanaka, Hiroshi; Takahashi, Takashi; Kaneko, Satoshi (2013-03-01). “β-Galactosyl Yariv Reagent bindet an das β-1,3-Galaktan von Arabinogalactan-Proteinen”. Pflanzenphysiologie. 161 (3): 1117–1126. mach:10.1104/S.112.211722. ISSN 0032-0889. PMC 3585584. PMID 23296690.
- ^ Paulsen, BS; Craik, DJ; Dunstan, DE; Stein, BA; Bacic, A. (2014-06-15). „Das Yariv-Reagenz: Verhalten in verschiedenen Lösungsmitteln und Wechselwirkung mit einem Gummiarabikum-Arabinogalactanprotein“. Kohlenhydrat-Polymere. 106: 460–468. mach:10.1016/j.carbpol.2014.01.009. ISSN 0144-8617. PMID 24721102.
- ^ Liang, Yan; Basu, Debarati; Pattathil, Sivakumar; Xu, Wen-Liang; Venetien, Alexandra; Martin, Stanton L.; Faik, Ahmed; Hahn, Michael G.; Showalter, Allan M. (13.10.2013). “Biochemische und physiologische Charakterisierung von fut4- und fut6-Mutanten, die bei der Arabinogalactan-Protein-Fucosylierung in Arabidopsis defekt sind”. Zeitschrift für experimentelle Botanik. 64 (18): 5537–5551. mach:10.1093/jxb/ert321. ISSN 1460-2431. PMC 3871811. PMID 24127514.
- ^ Okay, Takuji; Saito, Fumie; Shimma, Yoh-ichi; Yoko-o, Takehiko; Nomura, Yoshiyuki; Matsuoka, Ken; Jigami, Yoshifumi (2009-11-18). “Charakterisierung von endoplasmatischer Retikulum-lokalisierter UDP-d-Galactose: Hydroxyprolin-O-Galactosyltransferase mit synthetischen Peptidsubstraten in Arabidopsis”. Pflanzenphysiologie. 152 (1): 332–340. mach:10.1104/pp.109.146266. ISSN 0032-0889. PMC 2799367. PMID 19923238.
- ^ Kato, Hideaki; Takeuchi, Yoshimi; Tsumuraya, Yoichi; Hashimoto, Yohichi; Nakano, Hirofumi; Kováč, Pavol (2003-02-11). “In-vitro-Biosynthese von Galactanen durch membrangebundene Galactosyltransferase aus Rettich-Keimlingen (Raphanus sativus L.)”. Planta. 217 (2): 271–282. mach:10.1007/s00425-003-0978-7. ISSN 0032-0935. PMID 12783335. S2CID 5783849.
- ^ Showalter, Allan M.; Basu, Debarati (2016-06-15). “Extensin und Arabinogalactan-Protein-Biosynthese: Glykosyltransferasen, Forschungsherausforderungen und Biosensoren”. Grenzen in der Pflanzenwissenschaft. 7: 814. doi:10.3389/fpls.2016.00814. ISSN 1664-462X. PMC 4908140. PMID 27379116.
- ^ Egelund, Jack; Obel, Nicolai; Ulvskov, Peter; Geshi, Noomi; Pauly, Markus; Bacic, Antony; Petersen, Bent Larsen (2007-03-31). “Molekulare Charakterisierung von zwei Arabidopsis thaliana Glycosyltransferase-Mutanten, rra1 und rra2, die einen reduzierten Restarabinosegehalt in einem Polymer aufweisen, das eng mit dem Zellulosewandrest verbunden ist”. Molekularbiologie der Pflanzen. 64 (4): 439–451. mach:10.1007/s11103-007-9162-y. ISSN 0167-4412. PMID 17401635. S2CID 11643754.
- ^ ein B Qu, Yongmei; Egelund, Jack; Gilson, Paul R.; Houghton, Fiona; Gleeson, Paul A.; Schultz, Carolyn J.; Bacic, Antony (2008-06-12). “Identifizierung einer neuen Gruppe von mutmaßlichen Arabidopsis thaliana β-(1,3)-Galactosyltransferasen”. Molekularbiologie der Pflanzen. 68 (1–2): 43–59. mach:10.1007/s11103-008-9351-3. ISSN 0167-4412. PMID 18548197. S2CID 25896609.
- ^ Basu, Debarati; Tian, Lu; Wang, Wuda; Bobs, Shauni; Herock, Hayley; Travers, Andrew; Showalter, Allan M. (Dezember 2015). “Eine kleine multigene Hydroxyprolin-O-Galactosyltransferase-Familie funktioniert bei der Arabinogalactan-Protein-Glykosylierung, dem Wachstum und der Entwicklung bei Arabidopsis”. BMC Pflanzenbiologie. fünfzehn (1): 295. doi:10.1186/s12870-015-0670-7. ISSN 1471-2229. PMC 4687291. PMID 26690932.
- ^ Ogawa-Ohnishi, Mari; Matsubayashi, Yoshikatsu (2015-02-24). “Identifizierung von drei potenten HydroxyprolinO-galactosyltransferasen in Arabidopsis”. Das Pflanzenjournal. 81 (5): 736–746. mach:10.1111/tpj.12764. ISSN 0960-7412. PMID 25600942.
- ^ ein B Knoch, Eva; Dilokpimol, Adiphol; Tryfona, Theodora; Poulsen, Christian P.; Xiong, Guangyan; Harholt, Jesper; Petersen, Bent L.; Ulvskov, Peter; Hadi, Masood Z. (2013-11-29). “Eine β-Glucuronosyltransferase aus Arabidopsis thaliana, die an der Biosynthese von Typ-II-Arabinogalactan beteiligt ist, spielt eine Rolle bei der Zellverlängerung während des Keimlingswachstums”. Das Pflanzenjournal. 76 (6): 1016–1029. mach:10.1111/tpj.12353. ISSN 0960-7412. PMID 24128328.
- ^ Dilokpimol, Adiphol; Geshi, Naomi (2014-04-16). “Arabidopsis thalianaglucuronosyltransferase in der Familie GT14”. Signalgebung und Verhalten von Pflanzen. 9 (6): e28891. mach:10.4161/psb.28891. ISSN 1559-2324. PMC 4091549. PMID 24739253.
- ^ Wu, Yinging; Williams, Matthew; Bernhard, Sophie; Driouich, Azeddin; Showalter, Allan M.; Faik, Ahmed (2010-04-30). “Funktionale Identifizierung von zwei nicht redundanten Arabidopsis alpha(1,2)Fucosyltransferasen, die spezifisch für Arabinogalactan-Proteine sind”. Die Zeitschrift für biologische Chemie. 285 (18): 13638–13645. mach:10.1074/jbc.M110.102715. ISSN 1083-351X. PMC 2859526. PMID 20194500.
- ^ Tryfona, Theodora; Sie, Tina E.; Wagner, Tanja; Stott, Katherine; Keegstra, Kenneth; Dupree, Paul (2014-03-25). “Charakterisierung von FUT4- und FUT6-α-(1→2)-Fucosyltransferasen zeigt, dass das Fehlen der Wurzel-Arabinogalactan-Fucosylierung die Empfindlichkeit gegenüber Arabidopsis-Wurzelwachstumssalzen erhöht”. PLUS EINS. 9 (3): e93291. Bibcode:2014PLoSO…993291T. mach:10.1371/journal.pone.0093291. ISSN 1932-6203. PMC 3965541. PMID 24667545.
- ^ Dilokpimol, Adiphol; Poulsen, Christian; Vereb, György; Kaneko, Satoshi; Schulz, Alexander; Geshi, Naomi (2014). “Galactosyltransferasen aus Arabidopsis thaliana in der Biosynthese von Arabinogalactan Typ II: Molekulare Interaktion verstärkt Enzymaktivität”. BMC Pflanzenbiologie. 14 (1): 90. doi:10.1186/1471-2229-14-90. ISSN 1471-2229. PMC 4234293. PMID 24693939.
- ^ ein B C D Oxley, D.; Bacic, A. (1999-12-07). “Struktur des Glycosylphosphatidylinositol-Ankers eines Arabinogalactan-Proteins aus Suspensionskulturzellen von Pyrus communis”. Proceedings of the National Academy of Sciences of the United States of America. 96 (25): 14246-14251. Bibcode:1999PNAS…9614246O. mach:10.1073/pnas.96.25.14246. ISSN 0027-8424. PMC 24422. PMID 10588691.
- ^ ein B Yeats, Trevor H.; Bacic, Antony; Johnson, Kim L. (August 2018). “Pflanzenglykosylphosphatidylinositol verankerte Proteine am Plasmamembran-Zellwand-Nexus: Pflanzen GPI-verankerte Proteine”. Zeitschrift für integrative Pflanzenbiologie. 60 (8): 649–669. mach:10.1111/jipb.12659. hdl:11343/283902. ISSN 1744-7909. PMID 29667761.
- ^ Imhof, Isabella; Flury, Isabelle; Vionnet, Christine; Roubaty, Carole; Egger, Diane; Conzelmann, Andreas (2004-05-07). “Glycosylphosphatidylinositol (GPI) Proteine von Saccharomyces cerevisiae enthalten Ethanolaminphosphatgruppen an der alpha1,4-verknüpften Mannose des GPI-Ankers”. Die Zeitschrift für biologische Chemie. 279 (19): 19614–19627. mach:10.1074/jbc.M401873200. ISSN 0021-9258. PMID 14985347.
- ^ Ferguson, M.; Homans, S.; Dwek, R.; Rademacher, T. (1988-02-12). “Glycosyl-Phosphatidylinositol-Einheit, die das Oberflächen-Glykoprotein der Trypanosoma brucei-Variante an der Membran verankert”. Wissenschaft. 239 (4841): 753–759. Bibcode:1988Sc…239..753F. mach:10.1126/science.3340856. ISSN 0036-8075. PMID 3340856.
- ^ Ferguson, MA (September 1999). “Die Struktur, Biosynthese und Funktionen von Glycosylphosphatidylinositol-Ankern und die Beiträge der Trypanosomenforschung”. Zeitschrift für Zellwissenschaft. 112 (17): 2799–2809. mach:10.1242/jcs.112.17.2799. ISSN 0021-9533. PMID 10444375.
- ^ Strasser, Richard (2016-02-23). “Pflanzenproteinglykosylierung”. Glykobiologie. 26 (9): 926–939. mach:10.1093/glycob/cww023. ISSN 0959-6658. PMC 5045529. PMID 26911286.
- ^ Muniz, M.; Zurzolo, C. (2014-06-06). “Sortierung von GPI-verankerten Proteinen von Hefe zu Säugetieren – gemeinsame Wege an verschiedenen Standorten?”. Zeitschrift für Zellwissenschaft. 127 (13): 2793–2801. mach:10.1242/jcs.148056. ISSN 0021-9533. PMID 24906797.
- ^ ein B C D e Qu, Yongmei; Egelund, Jack; Gilson, Paul R.; Houghton, Fiona; Gleeson, Paul A.; Schultz, Carolyn J.; Bacic, Antony (2008-09-01). „Identifizierung einer neuen Gruppe von mutmaßlichen Arabidopsis thaliana β-(1,3)-Galactosyltransferasen“. Molekularbiologie der Pflanzen. 68 (1–2): 43–59. mach:10.1007/s11103-008-9351-3. ISSN 0167-4412. PMID 18548197. S2CID 25896609.
- ^ Egelund, Jack; Ellis, Miriam; Döblin, Monika; Qu, Yongmei; Bacic, Antony (2010). Ulvskov, Peter (Hrsg.). Jährliche Anlagenbewertungen. Wiley-Blackwell. S. 213–234. mach:10.1002/9781444391015.ch7. ISBN 9781444391015.
- ^ Basu, Debarati; Liang, Yan; Liu, Xiao; Himmeldirk, Klaus; Faik, Ahmed; Kieliszewski, Marcia; Held, Michael; Showalter, Allan M. (2013-04-05). “Funktionale Identifizierung einer Hydroxyprolin-O-galactosyltransferase, die für die Arabinogalactan-Proteinbiosynthese in Arabidopsis spezifisch ist”. Zeitschrift für biologische Chemie. 288 (14): 10132–10143. mach:10.1074/jbc.m112.432609. ISSN 0021-9258. PMC 3617256. PMID 23430255.
- ^ ein B Ogawa-Ohnishi, Mari; Matsubayashi, Yoshikatsu (2015-03-01). „Identifizierung von drei potenten Hydroxyprolin O-Galactosyltransferasen in Arabidopsis“. Das Pflanzenjournal. 81 (5): 736–746. mach:10.1111/tpj.12764. ISSN 1365-313X. PMID 25600942.
- ^ ein B Suzuki, Toshiya; Narciso, Joan Oñate; Zeng, Wei; Meene, Allison van de; Yasutomi, Masayuki; Takemura, Shunsuke; Lampugnani, Edwin R.; Döblin, Monika S.; Bacic, Antony (2017-01-01). “KNS4/UPEX1: Eine Typ-II-Arabinogalactan-β-(1,3)-Galactosyltransferase, die für die Pollenexine-Entwicklung erforderlich ist”. Pflanzenphysiologie. 173 (1): 183–205. mach:10.1104/S.16.01385. ISSN 0032-0889. PMC 5210738. PMID 27837085.
- ^ ein B C Geshi, Noomi; Johansen, Jorunn N.; Dilokpimol, Adiphol; Rolland, Aurélia; Belcram, Katia; Verger, Stephane; Kotake, Toshihisa; Tsumuraya, Yoichi; Kaneko, Satoshi (2013-10-01). “Eine Galactosyltransferase, die auf Arabinogalactan-Proteinglykane wirkt, ist für die Embryonalentwicklung bei Arabidopsis essentiell”. Das Pflanzenjournal. 76 (1): 128–137. mach:10.1111/tpj.12281. ISSN 1365-313X. PMID 23837821.
- ^ Dilokpimol, Adiphol; Poulsen, Christian Peter; Vereb, György; Kaneko, Satoshi; Schulz, Alexander; Geshi, Naomi (2014-04-03). “Galactosyltransferasen aus Arabidopsis thaliana in der Biosynthese von Arabinogalactan Typ II: Molekulare Interaktion verstärkt Enzymaktivität”. BMC Pflanzenbiologie. 14: 90. doi:10.1186/1471-2229-14-90. ISSN 1471-2229. PMC 4234293. PMID 24693939.
- ^ Dilokpimol, Adiphol; Geshi, Naomi (2014-06-01). “Arabidopsis thaliana Glucuronosyltransferase in der Familie GT14”. Signalgebung und Verhalten von Pflanzen. 9 (6): e28891. mach:10.4161/psb.28891. PMC 4091549. PMID 24739253.
- ^ ein B Wu, Yinging; Williams, Matthew; Bernhard, Sophie; Driouich, Azeddin; Showalter, Allan M.; Faik, Ahmed (2010-04-30). “Funktionale Identifizierung von zwei nicht redundanten Arabidopsis α(1,2)Fucosyltransferasen spezifisch für Arabinogalactan Proteine”. Zeitschrift für biologische Chemie. 285 (18): 13638–13645. mach:10.1074/jbc.m110.102715. ISSN 0021-9258. PMC 2859526. PMID 20194500.
- ^ ein B Liang, Yan; Basu, Debarati; Pattathil, Sivakumar; Xu, Wen-Liang; Venetien, Alexandra; Martin, Stanton L.; Faik, Ahmed; Hahn, Michael G.; Showalter, Allan M. (2013-12-01). “Biochemische und physiologische Charakterisierung von fut4- und fut6-Mutanten, die bei der Arabinogalactan-Protein-Fucosylierung in Arabidopsis defekt sind”. Zeitschrift für experimentelle Botanik. 64 (18): 5537–5551. mach:10.1093/jxb/ert321. ISSN 0022-0957. PMC 3871811. PMID 24127514.
- ^ ein B Gille, Sascha; Sharma, Vaishali; Baidoo, Edward EK; Keasling, Jay D.; Scheller, Henrik Vibe; Pauly, Markus (2013-07-01). “Arabinosylierung eines mit Yariv präzipitierbaren Zellwandpolymers beeinflusst das Pflanzenwachstum, wie am Beispiel der Arabidopsis-Glycosyltransferase-Mutante ray1 gezeigt”. Molekulare Pflanze. 6 (4): 1369–1372. mach:10.1093/mp/sst029. ISSN 1674-2052. PMID 23396039.
- ^ ein B Saha, Dipjyoti; Bhattacharya, Suvendu (Dezember 2010). “Hydrokolloide als Verdickungs- und Geliermittel in Lebensmitteln: eine kritische Überprüfung”. Zeitschrift für Lebensmittelwissenschaft und -technologie. 47 (6): 587–597. mach:10.1007/s13197-010-0162-6. ISSN 0022-1155. PMC 3551143. PMID 23572691.
- ^ ein B Barclay, Thomas G.; Tag, Candace Minhthu; Petrowski, Nikolai; Garg, Sanjay (Oktober 2019). “Überblick über die funktionelle Wirkstoffabgabe auf Polysaccharid-Partikelbasis”. Kohlenhydrat-Polymere. 221: 94–112. mach:10.1016/j.carbpol.2019.05.067. PMC 6626612. PMID 31227171.
- ^ ein B Aizat, WanM.; Preuß, James M.; Johnson, Alexander AT; Tester, Mark A.; Schultz, Carolyn J. (November 2011). “Untersuchung eines His-reichen Arabinogalactan-Proteins zur Mikronährstoff-Biofortifikation von Getreidekorn”. Physiologia Plantarum. 143 (3): 271–286. mach:10.1111/j.1399-3054.2011.01499.x. PMID 21707638.
- ^ ein B Fujita, Kiyotaka; Sasaki, Yuki; Kitahara, Kanefumi (September 2019). “Abbau pflanzlicher Arabinogalactan-Proteine durch Darmbakterien: Eigenschaften und Funktionen der beteiligten Enzyme”. Angewandte Mikrobiologie und Biotechnologie. 103 (18): 7451-7457. mach:10.1007/s00253-019-10049-0. ISSN 0175-7598. PMID 31384991. S2CID 199451378.
- ^ ein B Singha, Prajjal K.; Roy, Somenath; Dey, Satyahari (April 2007). “Schutzwirkung von Andrographolid- und Arabinogalactan-Proteinen aus Andrographis paniculata Nees. gegen ethanolinduzierte Toxizität bei Mäusen”. Zeitschrift für Ethnopharmakologie. 111 (1): 13–21. mach:10.1016/j.jep.2006.10.026. PMID 17127022.
- ^ Fincher, GB; Stein, BA; Clarke, AE (1983-06-01). “Arabinogalactan-Proteine: Struktur, Biosynthese und Funktion”. Jährliche Überprüfung der Pflanzenphysiologie. 34 (1): 47–70. mach:10.1146/annurev.pp.34.060183.000403. ISSN 0066-4294.
- ^ Ma, Yingxuan; Zeng, Wei; Bacic, Antony; Johnson, Kim (2018). „AGPs durch Zeit und Raum“. Jährliche Pflanzenbewertungen online. 3. S. 767–804. mach:10.1002/9781119312994.apr0608. ISBN 9781119312994. ISSN 2639-3832.
- ^ Nguema-Ona, Eric; Vicré-Gibouin, Maïté; Cannesan, Marc-Antoine; Driouich, Azeddine (August 2013). “Arabinogalactan-Proteine in Wurzel-Mikroben-Interaktionen”. Trends in der Pflanzenwissenschaft. 18 (8): 440–449. mach:10.1016/j.tplants.2013.03.006. ISSN 1878-4372. PMID 23623239.
- ^ Filmus, Jorge; Capurro, Mariana; Rast, Jonathan (2008). “Glypicaner”. Genombiologie. 9 (5): 224. doi:10.1186/de-2008-9-5-224. ISSN 1465-6906. PMC 2441458. PMID 18505598.
- ^ Schäfer, Liliana; Schäfer, Roland M. (2009-06-10). “Proteoglykane: Von Strukturverbindungen zu Signalmolekülen”. Zell- und Gewebeforschung. 339 (1): 237–246. mach:10.1007/s00441-009-0821-y. ISSN 0302-766X. PMID 19513755. S2CID 20358779.
- ^ Tan, Li; Showalter, Allan M.; Egelund, Jack; Hernandez-Sanchez, Arianna; Döblin, Monika S.; Bacic, Antony (2012). “Arabinogalactan-Proteine und die Forschungsherausforderungen für diese rätselhaften Proteoglykane auf der Pflanzenzelloberfläche”. Grenzen in der Pflanzenwissenschaft. 3: 140. doi:10.3389/fpls.2012.00140. ISSN 1664-462X. PMC 3384089. PMID 22754559.
- ^ Kitazawa, Kiminari; Tryfona, Theodora; Yoshimi, Yoshihisa; Hayashi, Yoshihiro; Kawauchi, Susumu; Antonow, Lüdmil; Tanaka, Hiroshi; Takahashi, Takashi; Kaneko, Satoshi (März 2013). “β-Galactosyl Yariv Reagent bindet an das β-1,3-Galaktan von Arabinogalactan-Proteinen”. Pflanzenphysiologie. 161 (3): 1117–1126. mach:10.1104/pp.112.211722. ISSN 0032-0889. PMC 3585584. PMID 23296690.
- ^ Yariv, J; Bericht, MM; Graf, L (1962-11-01). “Die Wechselwirkung von Glykosiden und Sacchariden mit Antikörpern zu den entsprechenden Phenylazoglykosiden”. Biochemisches Journal. 85 (2): 383–388. mach:10.1042/bj0850383. ISSN 0006-2936. PMC 1243744. PMID 14002491.
- ^ Tang, X.-C. (2006-07-07). “Die Rolle von Arabinogalactan-Proteinen, die an Yariv-Reagenzien binden, bei der Initiation, dem Schicksal der Zellentwicklung und der Aufrechterhaltung der Mikrosporenembryogenese in Brassica napus L. cv. Topas”. Zeitschrift für experimentelle Botanik. 57 (11): 2639–2650. mach:10.1093/jxb/erl027. ISSN 0022-0957. PMID 16829548.
- ^ Willats, William GT; Knox, J. Paul (Juni 1996). “Eine Rolle von Arabinogalactan-Proteinen bei der Pflanzenzellexpansion: Beweise aus Studien zur Interaktion von Beta-Glucosyl Yariv-Reagenz mit Sämlingen von Arabidopsis thaliana”. Das Pflanzenjournal. 9 (6): 919–925. mach:10.1046/j.1365-313x.1996.9060919.x. ISSN 0960-7412. PMID 8696368.
- ^ Chapman, Audrey; Blervacq, Anne-Sophie; Vasseur, Jacques; Hilbert, Jean-Louis (2000-08-10). “Arabinogalactan-Proteine in der somatischen Embryogenese von Cichorium: Wirkung von β-Glucosyl Yariv-Reagenz und Epitop-Lokalisierung während der Embryonalentwicklung”. Planta. 211 (3): 305–314. mach:10.1007/s004250000299. ISSN 0032-0935. PMID 10987548. S2CID 23116408.
- ^ Zagorchev, L; Stoineva, R; Odjakova, M (2013). “Änderungen der Arabinogalactan-Proteine während der somatischen Embryogenese In Suspension In-vitro-Kulturen von Dactylis glomerata L.” (PDF). Bulgarische Zeitschrift für Agrarwissenschaften. 17 (2): 35–38. ISSN 1310-0351.
- ^ Ruprecht, Colin; Bartetzko, MaxP.; Senf, Deborah; Dallabernadina, Pietro; Boos, Irene; Andersen, Mathias CF; Kotake, Toshihisa; Knox, J.Paul; Hahn, Michael G. (November 2017). „Ein synthetischer Glykan-Mikroarray ermöglicht die Epitop-Kartierung von Glykan-gerichteten Antikörpern der Pflanzenzellwand“. Pflanzenphysiologie. 175 (3): 1094-1104. mach:10.1104/S.17.00737. ISSN 0032-0889. PMC 5664464. PMID 28924016.
- ^ Seifert, Georg J.; Roberts, Keith (Juni 2007). “Die Biologie der Arabinogalactan-Proteine”. Jährliche Überprüfung der Pflanzenbiologie. 58 (1): 137–161. mach:10.1146/annurev.arplant.58.032806.103801. ISSN 1543-5008. PMID 17201686.
- ^ van Hengel, Arjon J.; Roberts, Keith (Oktober 2003). „AtAGP30, ein Arabinogalactan-Protein in den Zellwänden der Primärwurzel, spielt eine Rolle bei der Wurzelregeneration und Samenkeimung“. Das Pflanzenjournal. 36 (2): 256–270. mach:10.1046/j.1365-313x.2003.01874.x. ISSN 0960-7412. PMID 14535889.
- ^ Coimbra, Silvia; Costa, Mario; Mendes, Marta Adelina; Pereira, Ana Marta; Pinto, João; Pereira, Luis Gustavo (2010-02-17). “Frühe Keimung von Arabidopsis-Pollen in einer Doppelnullmutante für die Arabinogalactan-Proteingene AGP6 und AGP11”. Sexuelle Pflanzenreproduktion. 23 (3): 199–205. mach:10.1007/s00497-010-0136-x. ISSN 0934-0882. PMID 20162305. S2CID 32823162.
- ^ Suzuki, Toshiya; Narciso, Joan Oñate; Zeng, Wei; van de Meene, Allison; Yasutomi, Masayuki; Takemura, Shunsuke; Lampugnani, Edwin R.; Döblin, Monika S.; Bacic, Antony (2016-11-09). “KNS4/UPEX1: Eine Typ-II-Arabinogalactan-β-(1,3)-Galactosyltransferase, die für die Pollenexine-Entwicklung erforderlich ist”. Pflanzenphysiologie. 173 (1): 183–205. mach:10.1104/S.16.01385. ISSN 0032-0889. PMC 5210738. PMID 27837085.
- ^ Lamport, Derek TA; Várnai, Péter (Januar 2013). “Periplasmatische Arabinogalactan-Glykoproteine wirken als Kalziumkondensator, der das Pflanzenwachstum und die Entwicklung reguliert”. Neuer Phytologe. 197 (1): 58–64. mach:10.1111/nph.12005. ISSN 0028-646X. PMID 23106282.
- ^ Lamport, Derek TA; Tan, Li; Held, Michael; Kieliszewski, Marcia J. (2020-02-09). „Phyllotaxis schlägt ein neues Blatt auf – eine neue Hypothese“. Internationale Zeitschrift für Molekularwissenschaften. 21 (3): 1145. doi:10.3390/ijms21031145. ISSN 1422-0067. PMC 7037126. PMID 32050457.
- ^ Lopez-Hernandez, Federico; Tryfona, Theodora; Rizza, Annalisa; Yu, Xiaolan L.; Harris, Matthew OB; Webb, Alex AR; Kotake, Toshihisa; Dupree, Paul (Oktober 2020). “Calciumbindung durch Arabinogalactan-Polysaccharide ist wichtig für die normale Pflanzenentwicklung”. Die Pflanzenzelle. 32 (10): 3346–3369. mach:10.1105/tSt.20.00027. ISSN 1040-4651. PMC 7534474. PMID 32769130.
- ^ ein B Huber, O.; Sumper, M. (1994-09-15). “Algen-CAMs: Isoformen eines Zelladhäsionsmoleküls in Embryonen der Alge Volvox mit Homologie zu Drosophila fasciclin I”. Das EMBO-Journal. 13 (18): 4212–4222. mach:10.1002/j.1460-2075.1994.tb06741.x. ISSN 0261-4189. PMC 395348. PMID 7925267.
- ^ Seifert, Georg J. (2018-05-31). “Faszinierende Fasziline: Eine überraschend weit verbreitete Familie von Proteinen, die Wechselwirkungen zwischen dem Zelläußeren und der Zelloberfläche vermitteln”. Internationale Zeitschrift für Molekularwissenschaften. 19 (6): 1628. doi:10.3390/ijms19061628. ISSN 1422-0067. PMC 6032426. PMID 29857505.
- ^ Poon, Simon; Heide, Robyn Louise; Clarke, Adrienne Elizabeth (2012-08-02). “Ein chimäres Arabinogalactan-Protein fördert die somatische Embryogenese in Baumwollzellkulturen”. Pflanzenphysiologie. 160 (2): 684–695. mach:10.1104/pp.112.203075. ISSN 0032-0889. PMC 3461548. PMID 22858635.
- ^ Toonen, Marcel AJ; Schmidt, Ed DL; van Kammen, Ab; de Vries, Sacco C. (1997-09-26). “Fördernde und hemmende Wirkungen verschiedener Arabinogalactan-Proteine auf die somatische Embryogenese von Daucus carota L.”. Planta. 203 (2): 188-195. mach:10.1007/s004250050181. ISSN 0032-0935. S2CID 35053257.
- ^ Hu, Ying; Qin, Yuan; Zhao, Jie (2006-10-06). “Lokalisierung eines Arabinogalactan-Proteinepitops und die Wirkung von Yariv-Phenylglycosid während der zygotischen Embryoentwicklung von Arabidopsis thaliana”. Protoplasma. 229 (1): 21–31. mach:10.1007/s00709-006-0185-z. ISSN 0033-183X. PMID 17019527. S2CID 9707077.
- ^ Qin, Y. (2006-01-31). “Lokalisierung von Arabinogalactan-Proteinen in Eizellen, Zygoten und zweizelligen Proembryonen und Auswirkungen von -D-Glucosyl Yariv-Reagenz auf die Eizellbefruchtung und Zygotenteilung in Nicotiana tabacum L.” Zeitschrift für experimentelle Botanik. 57 (9): 2061-2074. mach:10.1093/jxb/erj159. ISSN 0022-0957. PMID 16720612.
- ^ Steinmacher, Douglas A.; Saare-Surminski, Katja; Lieberei, Reinhard (2012-06-19). “Arabinogalactan-Proteine und das Oberflächennetzwerk der extrazellulären Matrix während der somatischen Embryogenese der Pfirsichpalme”. Physiologia Plantarum. 146 (3): 336–349. mach:10.1111/j.1399-3054.2012.01642.x. ISSN 0031-9317. PMID 22574975.
- ^ Pan, Xiao; Yang, Xiao; Lin, Guimei; Zou, Ru; Chen, Houbin; amaj, Jozef; Xu, Chunxiang (2011-05-24). “Ultrastrukturelle Veränderungen und die Verteilung von Arabinogalactan-Proteinen während der somatischen Embryogenese von Bananen (Musa spp. AAA cv. ‘Yueyoukang 1’)”. Physiologia Plantarum. 142 (4): 372–389. mach:10.1111/j.1399-3054.2011.01478.x. ISSN 0031-9317. PMID 21496030.
- ^ Duchow, Stefanie; Dahlke, Renate I.; Geske, Thomas; Blaschek, Wolfgang; Classen, Birgit (November 2016). “Arabinogalactan-Proteine stimulieren die somatische Embryogenese und Pflanzenvermehrung von Pelargonium sidoides”. Kohlenhydrat-Polymere. 152: 149–155. mach:10.1016/j.carbpol.2016.07.015. ISSN 0144-8617. PMID 27516259.
- ^ Hallmann, A.; Kirk, DL (Dezember 2000). „Das entwicklungsregulierte ECM-Glykoprotein ISG spielt eine wesentliche Rolle bei der Organisation der ECM und der Orientierung der Zellen von Volvox“. Zeitschrift für Zellwissenschaft. 113 (24): 4605–4617. mach:10.1242/jcs.113.24.4605. ISSN 0021-9533. PMID 11082052.
- ^ Pereira, Ana Marta; Lopes, Ana Lucia; Coimbra, Silvia (2016-07-14). “JAGGER, ein AGP, das für die anhaltende Synergid-Degeneration und die Polytubey-Blockade bei Arabidopsis essentiell ist”. Signalgebung und Verhalten von Pflanzen. 11 (8): e1209616. mach:10.1080/15592324.2016.1209616. ISSN 1559-2324. PMC 5022411. PMID 27413888.
- ^ Levitin, Bella; Richter, Dganit; Markovich, Inbal; Zik, Moriyah (November 2008). “Arabinogalactan Proteine 6 und 11 werden für die Staubblatt- und Pollenfunktion bei Arabidopsis benötigt”. Das Pflanzenjournal. 56 (3): 351–363. mach:10.1111/j.1365-313x.2008.03607.x. ISSN 0960-7412. PMID 18644001.
- ^ Coimbra, S.; Costa, M.; Jones, B.; Mendes, MA; Pereira, LG (2009-05-11). “Pollenkornentwicklung ist in Arabidopsis agp6 agp11 Nullmutanten beeinträchtigt”. Zeitschrift für experimentelle Botanik. 60 (11): 3133–3142. mach:10.1093/jxb/erp148. ISSN 0022-0957. PMC 2718217. PMID 19433479.
- ^ Acosta-García, Gerardo; Vielle-Calzada, Jean-Philippe (2004-09-17). „Ein klassisches Arabinogalactan-Protein ist essentiell für die Initiierung der weiblichen Gametogenese bei Arabidopsis“. Die Pflanzenzelle. 16 (10): 2614–2628. mach:10.1105/tSt.104.024588. ISSN 1040-4651. PMC 520959. PMID 15377758.
- ^ Demesa-Arévalo, Edgar; Vielle-Calzada, Jean-Philippe (April 2013). “Das klassische Arabinogalactan-Protein AGP18 vermittelt die Megasporenselektion bei Arabidopsis”. Die Pflanzenzelle. 25 (4): 1274–1287. mach:10.1105/tSt.112.106237. ISSN 1040-4651. PMC 3663267. PMID 23572547.
- ^ ein B Li, Yunjing; Liu, Diqiu; Tu, Lili; Zhang, Xianlong; Wang, Li; Zhu, Longfu; Tan, Jiafu; Deng, Fenglin (2009-12-30). “Die Unterdrückung der GhAGP4-Genexpression unterdrückte die Initiierung und Verlängerung von Baumwollfasern”. Pflanzenzellenberichte. 29 (2): 193–202. mach:10.1007/s00299-009-0812-1. ISSN 0721-7714. PMID 20041253. S2CID 1341378.
- ^ Mashiguchi, Kiyoshi; Asami, Tadao; Suzuki, Yoshihito (2009-11-23). “Genomweite Identifizierung, Struktur- und Expressionsstudien und Mutantensammlung von 22 frühen Nodulin-ähnlichen Proteingenen in Arabidopsis”. Biowissenschaften, Biotechnologie und Biochemie. 73 (11): 2452–2459. mach:10.1271/bbb.90407. ISSN 0916-8451. PMID 19897921. S2CID 27449840.
- ^ Hou, Yingnan; Guo, Xinyang; Cypris, Philipp; Zhang, Ying; Bleckmann, Andrea; Cai, Le; Huang, Qingpei; Luo, Yu; Gu, Hongya (September 2016). “Mütterliche ENODLs sind für den Pollenschlauchempfang bei Arabidopsis erforderlich”. Aktuelle Biologie. 26 (17): 2343–2350. mach:10.1016/j.cub.2016.06.053. ISSN 0960-9822. PMC 5522746. PMID 27524487.
- ^ Lin, Sue; Dong, Heng; Zhang, Fang; Qiu, Lin; Wang, Fangzhan; Cao, Jiashu; Huang, Li (2014-01-31). „BcMF8, ein mutmaßliches für das Arabinogalactan-Protein kodierendes Gen, trägt zur Entwicklung der Pollenwand, der Bildung von Öffnungen und des Pollenschlauchwachstums bei Brassica campestris bei“. Annalen der Botanik. 113 (5): 777-788. mach:10.1093/aob/mct315. ISSN 1095-8290. PMC 3962243. PMID 24489019.
- ^ Lin, Sue; Yue, Xiaoyan; Miao, Yingjing; Yu, Youjian; Dong, Heng; Huang, Li; Cao, Jiashu (2018-03-09). “Die unterschiedlichen Funktionen zweier klassischer Arabinogalactan-Proteine BcMF8 und BcMF18 während der Pollenwandentwicklung in Brassica campestris”. Das Pflanzenjournal. 94 (1): 60–76. mach:10.1111/tpj.13842. ISSN 0960-7412. PMID 29385650.
- ^ Cheung, Alice Y; Wang, Hong; Wu, Hen-ming (August 1995). “Ein floral übertragendes gewebespezifisches Glykoprotein zieht Pollenschläuche an und stimuliert deren Wachstum”. Zelle. 82 (3): 383–393. mach:10.1016/0092-8674(95)90427-1. ISSN 0092-8674. PMID 7634328. S2CID 17604437.
- ^ Nathan Hancock, C.; Kent, Lia; McClure, Bruce A. (2005-08-08). “Das stylare 120 kDa Glykoprotein wird für die S-spezifische Pollenabstoßung in Nicotiana benötigt”. Das Pflanzenjournal. 43 (5): 716–723. mach:10.1111/j.1365-313x.2005.02490.x. ISSN 0960-7412. PMID 16115068.
- ^ Tan, Hexen; Liang, Wanqi; Hu, Jianping; Zhang, Dabing (Juni 2012). “MTR1 kodiert ein sekretorisches Fasziklin-Glykoprotein, das für die männliche Fortpflanzungsentwicklung in Reis benötigt wird”. Entwicklungszelle. 22 (6): 1127-1137. mach:10.1016/j.devcel.2012.04.011. ISSN 1534-5807. PMID 22698279.
- ^ Yang, Jie; Sardar, Harjinder S.; McGovern, Kathleen R.; Zhang, Yizhu; Showalter, Allan M. (2007-01-08). “Ein lysinreiches Arabinogalactan-Protein in Arabidopsis ist für das Pflanzenwachstum und die Entwicklung, einschließlich der Zellteilung und -expansion, unerlässlich.”. Das Pflanzenjournal. 49 (4): 629–640. mach:10.1111/j.1365-313x.2006.02985.x. ISSN 0960-7412. PMID 17217456.
- ^ Tan, Li; Eberhard, Stefan; Pattathil, Sivakumar; Wärter, Clayton; Glushka, John; Yuan, Chunhua; Hao, Zhangying; Zhu, Xiang; Avci, Utku (Januar 2013). “Ein Arabidopsis-Zellwand-Proteoglykan besteht aus Pektin und Arabinoxylan, die kovalent an ein Arabinogalactan-Protein gebunden sind”. Die Pflanzenzelle. 25 (1): 270–287. mach:10.1105/tSt.112.107334. ISSN 1040-4651. PMC 3584541. PMID 23371948.
- ^ Johnson, Kim L.; Kibble, Natalie AJ; Bacic, Antony; Schultz, Carolyn J. (2011-09-22). “Eine Fasciclin-ähnliche Arabinogalactan-Protein (FLA)-Mutante von Arabidopsis thaliana, fla1, zeigt Defekte in der Sprossregeneration”. PLUS EINS. 6 (9): e25154. Bibcode:2011PLoSO…625154J. mach:10.1371/journal.pone.0025154. ISSN 1932-6203. PMC 3178619. PMID 21966441.
- ^ Shi, Huazhong; Kim, YongSig; Guo, Yan; Stevenson, Becky; Zhu, Jian-Kang (2002-12-13). “Der SOS5-Locus von Arabidopsis kodiert für ein mutmaßliches Zelloberflächen-Adhäsionsprotein und ist für die normale Zellexpansion erforderlich”. Die Pflanzenzelle. fünfzehn (1): 19–32. mach:10.1105/tpc.007872. ISSN 1040-4651. PMC 143448. PMID 12509519.
- ^ Harpaz-Saad, Smadar; McFarlane, Heather E.; Xu, Schulter; Divi, Uday K.; Vorwärts, Bronwen; Westlich, Tamara L.; Kieber, Joseph J. (2011-10-10). “Die Zellulose-Synthese über den FEI2 RLK/SOS5-Weg und die CELLULOSE-SYNTHASE 5 ist für die Struktur des Samenhüllenschleims in Arabidopsis erforderlich”. Das Pflanzenjournal. 68 (6): 941–953. mach:10.1111/j.1365-313x.2011.04760.x. ISSN 0960-7412. PMID 21883548.
- ^ Griffiths, Jonathan S.; Tsai, Allen Yi-Lun; Xue, Hui; Voiniciuc, Cătălin; ola, Krešimir; Seifert, Georg J.; Mansfield, Shawn D.; Haughn, George W. (2014-05-07). “SALZ-ÜBERLÄSSIG SENSITIVE5 vermittelt die Adhärenz und Organisation von Arabidopsis-Samenhüllenschleim durch Pektine”. Pflanzenphysiologie. 165 (3): 991–1004. mach:10.1104/pp.114.239400. ISSN 0032-0889. PMC 4081351. PMID 2408103.
- ^ Griffiths, Jonathan S.; Crepeau, Marie-Jeanne; Ralet, Marie-Christine; Seifert, Georg J.; Norden, Helen M. (2016-07-29). “Sezieren von Samenschleimadhärenz, vermittelt durch FEI2 und SOS5”. Grenzen in der Pflanzenwissenschaft. 7: 1073. doi:10.3389/fpls.2016.01073. ISSN 1664-462X. PMC 4965450. PMID 27524986.
- ^ Xue, Hui; Veit, Christiane; Abas, Lindy; Tryfona, Theodora; Maresch, Daniel; Ricardi, Martiniano M.; Estevez, José Manuel; Strasser, Richard; Seifert, Georg J. (2017-06-13). “Arabidopsis thaliana FLA4 fungiert als Glykan-stabilisierter löslicher Faktor über seine Carboxy-proximale Fasciclin-1-Domäne”. Das Pflanzenjournal. 91 (4): 613–630. mach:10.1111/tpj.13591. ISSN 0960-7412. PMC 5575511. PMID 28482115.
- ^ Lee, Kieran JD; Sakata, Yoichi; Mau, Shaio-Lim; Pettolino, Filomena; Bacic, Antony; Quatrano, Ralph S.; Ritter, Celia D.; Knox, J. Paul (2005-09-30). “Arabinogalactan-Proteine sind für die apikale Zellverlängerung im Moos Physcomitrella patens erforderlich”. Die Pflanzenzelle. 17 (11): 3051-3065. mach:10.1105/tSt.105.034413. ISSN 1040-4651. PMC 1276029. PMID 16199618.
- ^ Kirchner, Thomas W.; Niehaus, Markus; Debener, Thomas; Schenk, Manfred K.; Herde, Marco (2017-09-22). “Effiziente Generierung von durch CRISPR/Cas9 vermittelten Mutationen im Haarwurzel-Transformationssystem von Brassica carinata”. PLUS EINS. 12 (9): e0185429. Bibcode:2017PLoSO..1285429K. mach:10.1371/journal.pone.0185429. ISSN 1932-6203. PMC 5609758. PMID 28937992.
- ^ MacMillan, Colleen P.; Mansfield, Shawn D.; Stachurski, Zbigniew H.; Evans, Rob; Southerton, Simon G. (2010-02-24). “Fasciclin-like Arabinogalactan Proteins: Spezialisierung für Stammbiomechanik und Zellwandarchitektur bei Arabidopsis und Eukalyptus”. Das Pflanzenjournal. 62 (4): 689–703. mach:10.1111/j.1365-313x.2010.04181.x. ISSN 0960-7412. PMID 20202165.
- ^ Motose, Hiroyasu; Sugiyama, Munetaka; Fukuda, Hiroo (Juni 2004). “Ein Proteoglycan vermittelt induktive Interaktion während der Entwicklung von Pflanzengefäßen”. Natur. 429 (6994): 873–878. Bibcode:2004Natur.429..873M. mach:10.1038/natur02613. ISSN 0028-0836. PMID 15215864. S2CID 4393158.
- ^ Wang, Haihai; Jiang, Chunmei; Wang, Schneiden; Yang, Yang; Yang, Lei; Gao, Xiaoyan; Zhang, Hongxia (2014-11-26). “Die Antisense-Expression des fasziklinähnlichen Arabinogalactan-Proteins FLA6-Gen in Populus hemmt die Expression seiner homologen Gene und verändert die Stammbiomechanik und Zellwandzusammensetzung in transgenen Bäumen”. Zeitschrift für experimentelle Botanik. 66 (5): 1291–1302. mach:10.1093/jxb/eru479. ISSN 1460-2431. PMC 4339592. PMID 25428999.
- ^ Albert, Markus; Belastegui-Macadam, Xana; Kaldenhoff, Ralf (November 2006). „Ein Angriff des Pflanzenparasiten Cuscuta reflexa induziert die Expression von attAGP, einem Bindungsprotein der Wirtstomate“. Das Pflanzenjournal. 48 (4): 548–556. mach:10.1111/j.1365-313x.2006.02897.x. ISSN 0960-7412. PMID 17076801.
- ^ Gaspar, Yolanda-Maria; Nam, Jaesung; Schultz, Carolyn Jane; Lee, Lan-Ying; Gilson, Paul R.; Gelvin, Stanton B.; Bacic, Antony (August 2004). “Charakterisierung der Arabidopsis-Lysin-reichen Arabinogalactan-Protein-AtAGP17-Mutante (rat1), die zu einer verringerten Effizienz der Agrobakterium-Transformation führt”. Pflanzenphysiologie. 135 (4): 2162–2171. mach:10.1104/pp.104.045542. ISSN 0032-0889. PMC 520787. PMID 15286287.
- ^ Tan, Li; Showalter, Allan M.; Egelund, Jack; Hernandez-Sanchez, Arianna; Döblin, Monika S.; Bacic, Antony (2012). “Arabinogalactan-Proteine und die Forschungsherausforderungen für diese rätselhaften Proteoglykane auf der Pflanzenzelloberfläche”. Grenzen in der Pflanzenwissenschaft. 3: 140. doi:10.3389/fpls.2012.00140. ISSN 1664-462X. PMC 3384089. PMID 22754559.
- ^ Johnson, Kim L.; Jones, Brian J.; Bacic, Antony; Schultz, Carolyn J. (2003-12-01). “Die fasziklinähnlichen Arabinogalactan-Proteine von Arabidopsis. Eine multigene Familie von mutmaßlichen Zelladhäsionsmolekülen”. Pflanzenphysiologie. 133 (4): 1911–1925. mach:10.1104/pp.103.031237. ISSN 0032-0889. PMC 300743. PMID 14645732.
- ^ ein B Basu, Debarati; Tian, Lu; Wang, Wuda; Bobs, Shauni; Herock, Hayley; Travers, Andrew; Showalter, Allan M. (2015-12-21). “Eine kleine multigene Hydroxyprolin-O-Galactosyltransferase-Familie funktioniert bei der Arabinogalactan-Protein-Glykosylierung, dem Wachstum und der Entwicklung bei Arabidopsis”. BMC Pflanzenbiologie. fünfzehn: 295. doi:10.1186/s12870-015-0670-7. ISSN 1471-2229. PMC 4687291. PMID 26690932.
- ^ Li, Wenhua L.; Liu, Yuanyuan; Douglas, Carl J. (2017-01-01). “Rolle von Glycosyltransferasen bei der Bildung von Pollenwand-Primexinen und der Exine-Musterung”. Pflanzenphysiologie. 173 (1): 167–182. mach:10.1104/S.16.00471. ISSN 0032-0889. PMC 5210704. PMID 27495941.
- ^ Knoch, Eva; Dilokpimol, Adiphol; Geshi, Naomi (2014). “Arabinogalactan-Proteine: Fokus auf kohlenhydrataktiven Enzymen”. Grenzen in der Pflanzenwissenschaft. 5: 198. doi:10.3389/fpls.2014.00198. ISSN 1664-462X. PMC 4052742. PMID 24966860.
Recent Comments