Exon – Wikipedia
Genanteil, der beim RNA-Spleißen nicht entfernt wird und Teil der reifen mRNA wird

Ein Exon ist ein Teil eines Gens, der einen Teil der endgültigen reifen RNA codiert, die von diesem Gen produziert wird, nachdem Introns durch RNA-Spleißen entfernt wurden. Der Begriff Exon bezieht sich sowohl auf die DNA-Sequenz innerhalb eines Gens als auch auf die entsprechende Sequenz in RNA-Transkripten. Beim RNA-Spleißen werden Introns entfernt und Exons als Teil der Erzeugung der reifen Messenger-RNA kovalent miteinander verbunden. So wie der gesamte Satz von Genen für eine Spezies das Genom bildet, bildet der gesamte Satz von Exons das Exom.
Geschichte[edit]
Der Begriff Exon stammt aus der exprimierten Region und wurde 1978 vom amerikanischen Biochemiker Walter Gilbert geprägt: “Der Begriff des Cistrons … muss durch den einer Transkriptionseinheit ersetzt werden, die Regionen enthält, die vom reifen Botenstoff verloren gehen – was wir Introns nennen ( für intragene Regionen) – abwechselnd mit Regionen, die exprimiert werden – Exons. “[1]
Diese Definition wurde ursprünglich für Protein-kodierende Transkripte gemacht, die vor der Übersetzung gespleißt werden. Der Begriff umfasste später Sequenzen, die aus der rRNA entfernt wurden[2] und tRNA,[3] und es wurde später auch für RNA-Moleküle verwendet, die aus verschiedenen Teilen des Genoms stammen und dann durch Transspleißen ligiert werden.[4]
Beitrag zum Genom und zur Größenverteilung[edit]
Obwohl einzellige Eukaryoten wie Hefe entweder keine oder nur sehr wenige Introns aufweisen, weisen Metazoen und insbesondere Wirbeltiergenome einen großen Anteil an nichtkodierender DNA auf. Beispielsweise werden im menschlichen Genom nur 1,1% des Genoms von Exons überspannt, während 24% in Introns liegen, wobei 75% des Genoms intergene DNA sind.[5] Dies kann einen praktischen Vorteil in der omikgestützten Gesundheitsversorgung (wie der Präzisionsmedizin) bieten, da die kommerzialisierte Sequenzierung des gesamten Exoms eine kleinere und kostengünstigere Herausforderung darstellt als die kommerzialisierte Sequenzierung des gesamten Genoms. Die große Variation der Genomgröße und des C-Werts über die Lebensformen hinweg stellte eine interessante Herausforderung dar, die als C-Wert-Rätsel bezeichnet wird.
Über alle eukaryotischen Gene in der GenBank gab es (im Jahr 2002) durchschnittlich 5,48 Exons pro Gen. Das durchschnittliche Exon codierte 30-36 Aminosäuren.[6] Während das längste Exon im menschlichen Genom 11555 bp lang ist, wurde festgestellt, dass einige Exons nur 2 bp lang sind.[7] Ein Einzelnukleotid-Exon wurde von der Arabidopsis Genom.[8]
Struktur und Funktion[edit]
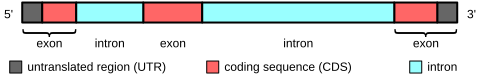
Exons in einem Messenger-RNA-Vorläufer (Prä-mRNA). Exons können sowohl Sequenzen enthalten, die für Aminosäuren (rot) kodieren, als auch nicht translatierte Sequenzen (grau). Introns – die Teile der Prä-mRNA, die sich nicht in der mRNA befinden – (blau) werden entfernt und die Exons werden zusammengefügt (gespleißt), um die endgültige funktionelle mRNA zu bilden. Die 5′- und 3′-Enden der mRNA sind markiert, um die beiden nicht translatierten Regionen zu unterscheiden (grau).
In Protein-kodierenden Genen umfassen die Exons sowohl die Protein-kodierende Sequenz als auch die 5′- und 3′-untranslatierten Regionen (UTR). Oft enthält das erste Exon sowohl die 5′-UTR als auch den ersten Teil der codierenden Sequenz, aber Exons, die nur Regionen von 5′-UTR oder (seltener) 3′-UTR enthalten, treten in einigen Genen auf, dh die UTRs können Introns enthalten .[9] Einige nicht-kodierende RNA-Transkripte haben auch Exons und Introns.
Reife mRNAs, die aus demselben Gen stammen, müssen nicht dieselben Exons enthalten, da verschiedene Introns in der Prä-mRNA durch den Prozess des alternativen Spleißens entfernt werden können.
Exonisierung ist die Erzeugung eines neuen Exons als Ergebnis von Mutationen in Introns.[10]
Experimentelle Ansätze mit Exons[edit]
Exon-Trapping oder “Gen-Trapping” ist eine molekularbiologische Technik, die die Existenz des Intron-Exon-Spleißens ausnutzt, um neue Gene zu finden.[11] Das erste Exon eines “gefangenen” Gens spleißt in das Exon, das in der Insertions-DNA enthalten ist. Dieses neue Exon enthält den ORF für ein Reportergen, das nun unter Verwendung der Enhancer, die das Zielgen steuern, exprimiert werden kann. Ein Wissenschaftler weiß, dass ein neues Gen eingefangen wurde, als das Reportergen exprimiert wurde.
Das Spleißen kann experimentell modifiziert werden, so dass gezielte Exons von reifen mRNA-Transkripten ausgeschlossen werden, indem der Zugang von spleißdirigierenden kleinen nuklearen Ribonukleoproteinpartikeln (snRNPs) zu Prä-mRNA unter Verwendung von Morpholino-Antisense-Oligos blockiert wird.[12] Dies ist zu einer Standardtechnik in der Entwicklungsbiologie geworden. Morpholino-Oligos können auch gezielt eingesetzt werden, um zu verhindern, dass Moleküle, die das Spleißen regulieren (z. B. Spleißverstärker, Spleißunterdrücker), an Prä-mRNA binden und so die Spleißmuster verändern.
Siehe auch[edit]
Verweise[edit]
- ^ Gilbert W (Februar 1978). “Warum Gene in Stücken?” Natur. 271 (5645): 501. doi:10.1038 / 271501a0. PMID 622185.
- ^ Kister KP, Eckert WA (März 1987). “Charakterisierung eines authentischen Intermediats im Selbstspleißprozess von ribosomaler Vorläufer-RNA in Makronuklei von Tetrahymena thermophila”. Nukleinsäureforschung. 15 (5): 1905–20. doi:10.1093 / nar / 15.5.1905. PMC 340607. PMID 3645543.
- ^ Valenzuela P., Venegas A., Weinberg F., Bischof R., Rutter WJ (Januar 1978). “Struktur von Hefe-Phenylalanin-tRNA-Genen: ein intervenierendes DNA-Segment innerhalb der Region, die für die tRNA kodiert”. Verfahren der National Academy of Sciences der Vereinigten Staaten von Amerika. 75 (1): 190–4. doi:10.1073 / pnas.75.1.190. PMC 411211. PMID 343104.
- ^ Liu AY, Van der Ploeg LH, FA Rijsewijk, P Borst (Juni 1983). “Die Transpositionseinheit des varianten Oberflächenglykoprotein-Gens 118 von Trypanosoma brucei. Vorhandensein wiederholter Elemente an seiner Grenze und Abwesenheit von Promotor-assoziierten Sequenzen”. Journal of Molecular Biology. 167 (1): 57–75. doi:10.1016 / S0022-2836 (83) 80034-5. PMID 6306255.
- ^ Venter JC; et al. (2000). “Die Sequenz des menschlichen Genoms”. Wissenschaft. 291 (5507): 1304–51. doi:10.1126 / science.1058040. PMID 11181995.
- ^ Sakharkar M, Passetti F, de Souza JE, Long M, de Souza SJ (2002). “ExInt: eine Exon-Intron-Datenbank”. Nucleic Acids Res. 30 (1): 191–4. doi:10.1093 / nar / 30.1.191. PMC 99089. PMID 11752290.
- ^ Sakharkar MK; Chow VT; Kangueane P. (2004). “Verteilung von Exons und Introns im menschlichen Genom”. In Silico Biol. 4 (4): 387–93. PMID 15217358.
- ^ Guo Lei, Liu Chun-Ming (2015). “”Ein Einzelnukleotid-Exon gefunden in Arabidopsis “. Wissenschaftliche Berichte. 5: 18087. doi:10.1038 / srep18087. PMC 4674806. PMID 26657562.
- ^ Bicknell, AA (Dezember 2012). “Introns in UTRs: Warum wir aufhören sollten, sie zu ignorieren”. BioEssays. 34 (12): 1025–1034. doi:10.1002 / bies.201200073. PMID 23108796. S2CID 5808466.
- ^ Sorek R (Oktober 2007). “Die Geburt neuer Exons: Mechanismen und evolutionäre Konsequenzen”. RNA. 13 (10): 1603–8. doi:10.1261 / rna.682507. PMC 1986822. PMID 17709368.
- ^ Duyk G. M; Kim SW; Myers R. M; Cox D. R (1990). “Exon-Trapping: ein genetischer Screen zur Identifizierung von transkribierten Kandidatensequenzen in geklonter genomischer Säugetier-DNA”. Verfahren der Nationalen Akademie der Wissenschaften. 87 (22): 8995–8999. doi:10.1073 / pnas.87.22.8995. PMC 55087. PMID 2247475.
- ^ Morcos PA (Juni 2007). “Erreichen einer gezielten und quantifizierbaren Veränderung des mRNA-Spleißens mit Morpholino-Oligos”. Biochemische und biophysikalische Forschungskommunikation. 358 (2): 521–7. doi:10.1016 / j.bbrc.2007.04.172. PMID 17493584.
Allgemeine Hinweise[edit]
Externe Links[edit]
| Nachsehen Exon in Wiktionary, dem kostenlosen Wörterbuch. |
Recent Comments