Stop Codon – Wikipedia
Ein Codon, das das Ende einer Protein-codierenden Sequenz markiert

In der Molekularbiologie (insbesondere Proteinbiosynthese) a Codon stoppen (oder Terminationscodon) ist ein Codon (Nukleotidtriplett innerhalb der Messenger-RNA), das die Beendigung des Translationsprozesses des aktuellen Proteins signalisiert.[1] Die meisten Codons in Messenger-RNA entsprechen der Addition einer Aminosäure an eine wachsende Polypeptidkette, die letztendlich zu einem Protein werden kann; Stopcodons signalisieren die Beendigung dieses Prozesses durch Bindung von Freisetzungsfaktoren, die dazu führen, dass sich die ribosomalen Untereinheiten trennen und die Aminosäurekette freisetzen.
Während Startcodons nahegelegene Sequenzen oder Initiationsfaktoren benötigen, um die Translation zu starten, reicht ein Stoppcodon allein aus, um die Terminierung zu initiieren.
Eigenschaften[edit]
Standardcodons[edit]
Im genetischen Standardcode gibt es drei verschiedene Terminationscodons:
| Codon | Standardcode (Übersetzungstabelle 1) |
Name | ||
|---|---|---|---|---|
| DNA | RNA | |||
| ETIKETT | UAG | STOP = Ter | ||
| "Bernstein" | TAA | UAA STOP = Ter | ||
| "Ocker" | TGA UGA | STOP = Ter | ||
[edit]
[2]“opal” (oder “umber”)Alternative Stoppcodons[3] Es gibt Variationen des genetischen Standardcodes, und alternative Stoppcodons wurden im mitochondrialen Genom von Wirbeltieren gefunden. Scenedesmus obliquus,[4]
| und | Thraustochytrium | . | Biochemische Eigenschaften von Aminosäuren | Unpolar | Polar |
Basic[edit]
Säure[5]
| Neu zugewiesene Stoppcodons | Der nukleare genetische Code ist flexibel, wie durch variante genetische Codes veranschaulicht, die Standard-Stopcodons Aminosäuren neu zuweisen. Tabelle der bedingten Stoppcodons und Vergleich mit dem genetischen Standardcode |
Genetischer Code | Übersetzung Tabelle |
Codon | ||||
|---|---|---|---|---|---|---|---|---|
| Bedingt | Übersetzung | |||||||
| Standardübersetzung | DNA | RNA | Karyorelict Atom | 27 TGA | UGA | Ter | oder | |
| Trp (W) | Ter | Kondylostoma nuklear | 28 | TAA UAA | Ter | |||
| oder | Gln (Q) | Ter | ETIKETT UAG | |||||
| Ter | oder | Gln | (Q) Ter | |||||
| TGA UGA | Ter | oder Trp | (W) | Ter | Blastocrithidia | |||
| nuklear | 31 | TAA UAA | Ter | oder Glu | ||||
(E)[edit]
Ter[6]
[edit]
ETIKETT[7][8] UAG Ter [9] [10] oder[11]
Glu[edit]
(E)[12] Ter[13]
[edit]
Übersetzung2007 wurde das UGA-Codon als das für Selenocystein (Sec) kodierende Codon identifiziert und in 25 Selenoproteinen gefunden, die sich im aktiven Zentrum des Proteins befinden. Die Translation dieses Codons wird durch die Nähe des SECIS-Elements (SElenoCysteine Incorporation Sequence) ermöglicht. Das UAG-Codon kann auf ähnliche Weise in Pyrrolysin (Pyl) übersetzt werden.Genomverteilung
Die Verteilung der Stopcodons im Genom eines Organismus ist nicht zufällig und kann mit dem GC-Gehalt korrelieren. Zum Beispiel dieE coliDas K-12-Genom enthält 2705 TAA (63%), 1257 TGA (29%) und 326 TAG (8%) Stopcodons (GC-Gehalt 50,8%).[edit]
Auch die Substrate für die Stoppcodons Freisetzungsfaktor 1 oder Freisetzungsfaktor 2 korrelieren stark mit der Häufigkeit von Stoppcodons. Eine groß angelegte Untersuchung von Bakterien mit einem breiten Spektrum an GC-Gehalten zeigt, dass die Häufigkeit des Auftretens von TAA zwar negativ mit dem GC-Gehalt korreliert und die Häufigkeit des Auftretens von TGA positiv mit dem GC-Gehalt korreliert, die Häufigkeit des Auftretens jedoch des TAG-Stopcodons, das häufig das minimal verwendete Stopcodon in einem Genom ist, wird nicht durch den GC-Gehalt beeinflusst. AnerkennungDie Erkennung von Stopcodons in Bakterien wurde mit dem sogenannten “Tripeptid-Anticodon” in Verbindung gebracht. ein hochkonserviertes Aminosäuremotiv in RF1 (PxT) und RF2 (SPF). Obwohl dies durch Strukturstudien gestützt wird, wurde gezeigt, dass die Tripeptid-Anticodon-Hypothese eine übermäßige Vereinfachung darstellt.Nomenklatur[14][15]
Stopcodons wurden historisch viele verschiedene Namen gegeben, da sie jeweils einer bestimmten Klasse von Mutanten entsprachen, die sich alle auf ähnliche Weise verhielten. Diese Mutanten wurden zuerst in Bakteriophagen (T4 und Lambda) isoliert, Viren, die die Bakterien infizieren[16]
Escherichia coli
. Mutationen in viralen Genen schwächten ihre Infektionsfähigkeit und erzeugten manchmal Viren, die nur innerhalb bestimmter Sorten von infizieren und wachsen konnten E coli.Bernstein[edit]
Mutationen ( UAG)[15]
Sie waren die ersten entdeckten Unsinnmutationen, die von Richard H. Epstein und Charles Steinberg isoliert und nach ihrem Freund und Caltech-Doktoranden Harris Bernstein benannt wurden, dessen Nachname bedeutet[17]
“Bernstein” auf Deutsch ( vgl. Bernstein).Viren mit Bernsteinmutationen zeichnen sich durch ihre Fähigkeit aus, nur bestimmte Bakterienstämme zu infizieren, die als Bernsteinunterdrücker bekannt sind. Diese Bakterien tragen ihre eigene Mutation, die eine Wiederherstellung der Funktion in den mutierten Viren ermöglicht. Beispielsweise ermöglicht eine Mutation in der tRNA, die das Amber-Stop-Codon erkennt, dass die Translation das Codon “durchliest” und ein Protein voller Länge erzeugt, wodurch die normale Form des Proteins wiederhergestellt und die Amber-Mutation “unterdrückt” wird.[edit]
Somit sind Bernsteinmutanten eine ganze Klasse von Virusmutanten, die in Bakterien wachsen können, die Bernstein-Suppressormutationen enthalten. Ähnliche Suppressoren sind auch für Ocker- und Opal-Stop-Codons bekannt.[18]
Ocker Mutationen (UAA[15] )
Es war die zweite entdeckte Stop-Codon-Mutation. Dieses zweite Stoppcodon erinnert an die übliche gelb-orange-braune Farbe von Bernstein und erhielt den Namen[edit]
“Ocker”[edit]
, ein orange-rotbraunes Mineralpigment. Ocker-Mutantenviren hatten eine ähnliche Eigenschaft wie Bernsteinmutanten, da sie die Infektionsfähigkeit innerhalb bestimmter Suppressorstämme von Bakterien wiedererlangten. Der Satz von Ocker-Suppressoren unterschied sich von Bernstein-Suppressoren, so dass gefolgert wurde, dass Ocker-Mutanten einem anderen Nukleotid-Triplett entsprechen. Durch eine Reihe von Mutationsexperimenten, bei denen diese Mutanten miteinander und mit anderen bekannten Aminosäurecodons verglichen wurden, gelangte Sydney Brenner zu dem Schluss, dass die Bernstein- und Ocker-Mutationen den Nukleotidtripletts “UAG” und “UAA” entsprachen.Opal
oder[edit]
Umber Mutationen ( UGA
)
Das dritte und letzte Stoppcodon im genetischen Standardcode wurde bald danach entdeckt und entspricht dem Nukleotidtriplett “UGA”.[19]Um weiterhin mit dem Thema der farbigen Mineralien übereinzustimmen, wurde das dritte Unsinn-Codon bekannt als[20] “Opal”[21] Dies ist eine Art Kieselsäure, die eine Vielzahl von Farben zeigt.[22]
Nonsense-Mutationen, die dieses vorzeitige Stoppcodon erzeugten, wurden später als Opalmutationen oder Umbermutationen bezeichnet.[edit]
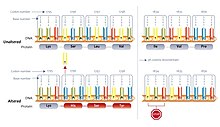
Unsinn Nonsense-Mutationen sind Änderungen in der DNA-Sequenz, die ein vorzeitiges Stoppcodon einführen und dazu führen, dass das resultierende Protein abnormal verkürzt wird. Dies führt häufig zu einem Funktionsverlust des Proteins, da kritische Teile der Aminosäurekette nicht mehr entstehen. Aufgrund dieser Terminologie wurden Stoppcodons auch als bezeichnet Unsinn Codons.[23]
Nonstop[24]
EIN[25]
Nonstop-Mutation[edit]
ist eine Punktmutation, die innerhalb eines Stoppcodons auftritt. Nonstop-Mutationen bewirken die fortgesetzte Translation eines mRNA-Strangs in eine nicht translatierte Region. Die meisten Polypeptide, die aus einem Gen mit einer Nonstop-Mutation resultieren, sind aufgrund ihrer extremen Länge nicht funktionsfähig. Nonstop-Mutationen unterscheiden sich von Nonsense-Mutationen darin, dass sie kein Stop-Codon erzeugen, sondern eines löschen. Nonstop-Mutationen unterscheiden sich auch von Missense-Mutationen, bei denen es sich um Punktmutationen handelt, bei denen ein einzelnes Nukleotid geändert wird, um einen Ersatz durch eine andere Aminosäure zu bewirken. Nonstop-Mutationen wurden mit mehreren angeborenen Erkrankungen in Verbindung gebracht, einschließlich angeborener Nebennierenhyperplasie, variable Dysgenese des vorderen Segments,[26][27] Mukoviszidose[28] und mitochondriale neurogastrointestinale Enzephalomyopathie.[29]
Versteckte Haltestellen[edit]
Ein Beispiel für eine einzelne Basendeletion, die ein Stoppcodon bildet.[30]
Versteckte Haltestellen[edit]
sind Non-Stop-Codons, die als Stop-Codons gelesen würden, wenn sie um +1 oder -1 verschoben wären. Diese beenden die Translation vorzeitig, wenn die entsprechende Frame-Verschiebung (z. B. aufgrund eines ribosomalen RNA-Schlupfes) vor dem versteckten Stopp auftritt. Es wird angenommen, dass dies die Ressourcenverschwendung bei nicht funktionierenden Proteinen und die Produktion potenzieller Zytotoxine verringert. Forscher der Louisiana State University schlagen die[edit]
- Hinterhalt Hypothese , für die versteckte Stopps ausgewählt sind. Codons, die versteckte Stopps bilden können, werden in Genomen häufiger verwendet als synonyme Codons, die andernfalls für dieselbe Aminosäure codieren würden. Instabile rRNA in einem Organismus korreliert mit einer höheren Häufigkeit versteckter Stopps. Diese Hypothese konnte jedoch mit einem größeren Datensatz nicht validiert werden.Stoppcodons und versteckte Stopps zusammen werden zusammen als Stoppsignale bezeichnet. Forscher an der Universität von Memphis fanden heraus, dass die Verhältnisse der Stoppsignale auf den drei Leserahmen eines Genoms (als Translationsstoppsignalverhältnis oder TSSR bezeichnet) genetisch verwandter Bakterien trotz ihrer großen Unterschiede im Gengehalt sehr ähnlich sind . Dieser nahezu identische Genom-TSSR-Wert genetisch verwandter Bakterien könnte darauf hindeuten, dass die Expansion des Bakteriengenoms durch ihre einzigartige Stoppsignal-Verzerrung dieser Bakterienspezies begrenzt ist. Translationales DurchlesenStoppen Sie die Codon-Unterdrückung
- oder translationales Durchlesen tritt auf, wenn in der Translation ein Stoppcodon als Sensecodon interpretiert wird, dh wenn eine (Standard-) Aminosäure durch das Stoppcodon “codiert” wird. Mutierte tRNAs können die Ursache für das Durchlesen sein, aber auch bestimmte Nukleotidmotive in der Nähe des Stoppcodons. Das translatorische Durchlesen ist bei Viren und Bakterien sehr häufig und wurde auch als Genregulationsprinzip bei Menschen, Hefen, Bakterien und Drosophila gefunden.Diese Art des endogenen translationalen Durchlesens stellt eine Variation des genetischen Codes dar, da ein Stoppcodon für eine Aminosäure codiert. Im Fall von menschlicher Malatdehydrogenase wird das Stoppcodon mit einer Häufigkeit von etwa 4% durchgelesen. Die am Stoppcodon inserierte Aminosäure hängt von der Identität des Stoppcodons selbst ab: Gln, Tyr und Lys wurden für die UAA- und UAG-Codons gefunden, während Cys, Trp und Arg für das UGA-Codon nach Masse identifiziert wurden Spektrometrie.Als Wasserzeichen verwenden Als Craig Venter 2010 die erste voll funktionsfähige, reproduzierende Zelle vorstellte, die von synthetischer DNA kontrolliert wird, beschrieb er, wie sein Team häufige Stoppcodons verwendete, um Wasserzeichen in RNA und DNA zu erzeugen, um zu bestätigen, dass die Ergebnisse tatsächlich synthetisch (und nicht kontaminiert oder anderweitig) waren es, um die Namen und Adressen der Autoren zu verschlüsseln. Siehe auchVerweise^ Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM (2000).“Kapitel 10 (Molekularbiologie der Genfunktion): Genetischer Code: Stopcodons” .Eine Einführung in die genetische Analyse . WH Freeman und Company.^
- Barrell, BG; Bankier, AT; Drouin, J. (1979-11-08). “Ein anderer genetischer Code in menschlichen Mitochondrien” . Natur .282 (5735): 189–194. doi:10.1038 / 282189a0 . ISSN 0028-0836. PMID226894 . S2CID4335828 .^AM Nedelcu, RW Lee, G. Lemieux, MW Gray, G. Burger (Juni 2000).
- “Die vollständige mitochondriale DNA-Sequenz von Scenedesmus obliquus spiegelt ein Zwischenstadium in der Entwicklung des mitochondrialen Genoms der Grünalgen wider “. Genomforschung. 10 (6): 819–831. doi:10.1101 / gr.10.6.819. PMC310893. PMID 10854413. CS1-Wartung: mehrere Namen: Autorenliste (Link)^ Wideman, Jeremy G.; Monier, Adam; Rodríguez-Martínez, Raquel; Leonard, Guy; Koch, Emily; Poirier, Camille; Maguire, Finlay; Milner, David S.; Irwin, Nicholas AT; Moore, Karen; Santoro, Alyson E. (25.11.2019).“Unerwartete mitochondriale Genomdiversität durch gezielte Einzelzellgenomik heterotropher Flagellenprotisten”
- . Naturmikrobiologie .5 (1): 154–165. doi:10.1038 / s41564-019-0605-4 . hdl: 10871/39819. ISSN2058-5276 . PMID31768028 . S2CID208279678
- . ^ Swart, Estienne Carl; Serra, Valentina; Petroni, Giulio; Nowacki, Mariusz (2016).“Genetische Codes ohne dediziertes Stoppcodon: Kontextabhängige Beendigung der Übersetzung” .Zelle . 166(3): 691–702. doi:10.1016 / j.cell.2016.06.020 . PMC4967479 . PMID27426948
- . ^ Papp, Laura Vanda; Lu, Jun; Holmgren, Arne; Khanna, Kum Kum (2007).“Von Selen zu Selenoproteinen: Synthese, Identität und ihre Rolle für die menschliche Gesundheit” .Antioxidantien & Redox-Signalisierung .9(7): 775–806. doi:10.1089 / ars.2007.1528 . PMID17508906 . S2CID38176932
- . ^ Povolotskaya IS, Kondrashov FA, Ledda A, Vlasov PK (2012).“Stopcodons in Bakterien sind nicht selektiv äquivalent” .Biologie Direkt . 7: 30. doi:10.1186 / 1745-6150-7-30 . PMC3549826 . PMID22974057
- . ^Korkmaz, Gürkan; Holm, Mikael; Wiens, Tobias; Sanyal, Suparna (2014). “Umfassende Analyse der Verwendung von Stopcodons in Bakterien und ihrer Korrelation mit der Häufigkeit von Freisetzungsfaktoren” [Genbank Accession Number: U00096].Das Journal of Biological Chemistry .289(44): 775–806. doi: 10.1074 / jbc.M114.606632. PMC
- 4215218 . PMID 25217634. ^“” Escherichia coli str. K-12 substr. MG1655, vollständiges Genom“”. GenBank. NCBI . Abgerufen2013-01-27
- . ^ Korkmaz, Gürkan; Holm, Mikael; Wiens, Tobias; Sanyal, Suparna (2014).“Umfassende Analyse der Verwendung von Stopcodons in Bakterien und ihrer Korrelation mit der Häufigkeit von Freisetzungsfaktoren” .Das Journal of Biological Chemistry . 289(44): 775–806. doi:10.1074 / jbc.M114.606632 . PMC4215218 . PMID25217634
- . ^ Wong, Tit-Yee; Fernandes, Sanjit; Sankhon, Naby; Leong, Patrick P; Kuo, Jimmy; Liu, Jong-Kang (2008).“Rolle vorzeitiger Stoppcodons in der bakteriellen Evolution” . Journal of Bacteriology.190 (20): 6718–6725. doi:10.1128 / JB.00682-08 . PMC2566208
- . PMID 18708500 . ^Ito, Koichi; Uno, Makiko; Nakamura, Yoshikazu (1999). “Ein Tripeptid ‘Anticodon’ entschlüsselt Codons in Messenger-RNA”.Natur .403 (6770): 680–684. doi: 10.1038 / 35001115. PMID10688208 . S2CID4331695 .^
- Korkmaz, Gürkan; Sanyal, Suparna (2017). Die R213I-Mutation in Freisetzungsfaktor 2 (RF2) ist ein Fortschritt für die Entwicklung eines allmächtigen Freisetzungsfaktors in Bakterien Escherichia coli“” .Journal of Biological Chemistry . 292 (36): 15134–15142. doi:10.1074 / jbc.M117.785238 . PMC5592688
- . PMID 28743745 . ^ Stahl FW (1995). “Die Bernsteinmutanten des Phagen T4”. Genetik.
- 141 (2): 439–442. PMC 1206745. PMID 8647382.
- ^ ein bc Lewin, Benjamin; Krebs, Jocelyn E.; Goldstein, Elliott S.; Kilpatrick, Stephen T. (2011-04-18). Lewins wesentliche GENE. Jones & Bartlett Verlag. ISBN978-1-4496-4380-5.^ Robin Cook.“Zusammenfassung der Mutationen von Bernstein, Ocker und Opal” .Welt der Genetik
- . Sturm. ^ Brenner, S.; Stretton, AOW; Kaplan, S. (1965). “Genetischer Code: Die ‘Unsinn’-Drillinge zur Kettenbeendigung und ihrer Unterdrückung”.Natur . 206(4988): 994–8. Bibcode:1965Natur.206..994B. doi:10.1038 / 206994a0 . PMID5320272 . S2CID28502898
- . ^ Brenner, S.; Barnett, L.; Katz, ER; Crick, FHC (1967). “UGA: Ein drittes Unsinn-Triplett im genetischen Code”.Natur . 213(5075): 449–50. Bibcode:1967Natur.213..449B . doi:10.1038 / 213449a0
- . PMID 6032223 . S2CID 4211867 .^ Pang S.; Wang W.; et al. (2002). “Eine neuartige Nonstop-Mutation im Stopcodon und eine neuartige Missense-Mutation im 3beta-Hydroxysteroid-Dehydrogenase (3beta-HSD) -Gen vom Typ II, die eine nichtklassische bzw. klassische angeborene Nebennierenhyperplasie mit 3beta-HSD-Mangel verursacht.”J Clin Endocrinol Metab . 87(6): 2556–63. doi:10.1210 / jc.87.6.2556 . PMID12050213 .^
- Doucette, L.; et al. (2011). “Eine neuartige Non-Stop-Mutation in FOXE3verursacht eine autosomal dominante Form der variablen Dysgenese des vorderen Segments einschließlich Peters-Anomalie ” .Europäisches Journal für Humangenetik . 19(3): 293–299. doi:10.1038 / ejhg.2010.210 . PMC3062009 . PMID21150893 .^
- Guimbellot, Jennifer; Sharma, Jyoti; Rowe, Steven M. (November 2017). “Auf dem Weg zu einer integrativen Therapie mit CFTR-Modulatoren: Fortschritte und Herausforderungen” . Pädiatrische Pulmonologie. 52(S48): S4 – S14. doi: 10.1002 / S. 23773 . ISSN1099-0496. PMC 6208153. PMID
- 28881097 . ^Torres-Torronteras, J.; Rodriguez-Palmero, A.; et al. (2011). “Eine neuartige Nonstop-Mutation in TYMP induziert bei einem MNGIE-Patienten mit schwerer Neuropathie keinen Nonstop-mRNA-Zerfall.” (PDF).Summen. Mutat .32
- (4): E2061 – E2068. doi: 10.1002 / humu.21447 . PMID21412940 .^ Seligmann, Hervé; Pollock, David D. (2004). “Die Hinterhaltshypothese: Versteckte Stoppcodons verhindern das Lesen von Genen außerhalb des Rahmens”. DNA und Zellbiologie.23 (10): 701–5. doi:10.1089 / 1044549042476910 . PMID15585128
- . ^ Cavalcanti, Andre; Chang, Charlotte H.; Morgens, David W. (2013).“Hinterhalt der Hinterhaltshypothese: Vorhersage und Bewertung von Off-Frame-Codonfrequenzen in prokaryotischen Genomen” . BMC Genomics.14 (418): 1–8. doi:10.1186 / 1471-2164-14-418 . PMC3700767
- . PMID 23799949 .^ Wong, Tit-Yee; Schwartzbach, Steve (2015). “Eine Fehlbeendigung von Proteinen löst genetische Krankheiten und Krebs aus und schränkt die Expansion des bakteriellen Genoms ein.” Zeitschrift für Umweltwissenschaften und Gesundheit, Teil C..33 (3): 255–85. doi:10.1080 / 10590501.2015.1053461
- . PMID 26087060 . S2CID20380447 .^ Namy O., Rousset JP, Napthine S., Brierley I. (2004). “Reprogrammierte genetische Dekodierung bei der zellulären Genexpression”.Molekulare Zelle.13 (2): 157–68. doi:10.1016 / S1097-2765 (04) 00031-0 . PMID14759362
- . ^ Schueren F., Lingner T., George R., Hofhuis J., Gartner J., Thoms S. (2014).“Peroxisomale Lactatdehydrogenase wird durch translationales Durchlesen bei Säugetieren erzeugt.” .eLife . 3: e03640. doi:10.7554 / eLife.03640 . PMC4359377 . PMID25247702
- . ^ Hofhuis J., Schueren F., Nötzel C., Lingner T., Gärtner J., Jahn O., Thoms S. (2016). “Die funktionelle Durchleseverlängerung der Malatdehydrogenase zeigt eine Modifikation des genetischen Codes.”.Öffnen Sie Biol .6 (11): 160246. doi: 10.1098 / rsob.160246. PMC5133446 . PMID27881739 .^
- Blanchet S., Cornu D., Argentini M., Namy O. (2014). “Neue Einblicke in den Einbau natürlicher Suppressor-tRNAs an Stopcodons inSaccharomyces cerevisiae“”.
Nucleic Acids Res
.
42
(15): 10061–72. doi:
10.1093 / nar / gku663
. PMC
4150775
. PMID
25056309
.
^
“Schau mir zu, wie ich das synthetische Leben enthülle”
“”
“”
.
Recent Comments