DN Politrasa-ウィキペディア、無料百科事典
ポリメラーゼDNA それらは、DNA複製プロセスに関与する酵素(携帯電話)です。彼らは、デオキシリボヌクレリン酸(DNTP)と対応する相補性型型DNAに対応するデオキシリボヌクレオチドと一致する新しいDNA鎖の合成を実行します。 DNAの複製で使用されるDNTPには、デオキシリボースの5 ‘ヒドロキシル基に付着した3つのリン酸塩が含まれており、窒素塩基に依存します。根本的な反応は、3’-OH基が成長している鎖の3 ‘末端で求核試薬として機能するリン酸塩基の移動です。求核攻撃は、デオキシリボヌクレオシド5 ‘トリフォスリン酸のαリン酸(デオキシリボースに最も近い)に生成され、無機ピオリン酸を放出し、DNAを伸ばす(新しいホスホディエステル結合を形成するとき)に生成されます。 1つのリン酸基のみが分離されている細胞で発生するほとんどの生物学的プロセスとは異なり(P 私 )、複製中に、最後の2つのリン酸基が分離され、ピロリン基群(pp) 私 ))
このプロセスは、化学式で要約できます。
(DNA) n +dntp↔(DNA) n+1 + pp 私
DNAポリメラーゼには4つの異なるDNTPと一致するアクティブ部位のみがありますが、塩基ペアA:T、C:Gの正しい結合はその形状に基づいて可能です。結合が正しくない場合、リン酸αの変位が3′-OH端になり、したがって正しい塩基のリズムを遅くします。
DNAポリメラーゼは、1秒あたり最大1000ヌクレオチドを追加することができます。これは、その性質、つまり、コピーするDNA型に関連付けられるたびに追加できるヌクレオチドの数によるものです。ヌクレオチドの添加は数ミリ秒続くプロセスであるため、触媒速度はDNAポリメラーゼがDNA、つまりその加工性に関連する時間に依存します。

鎖の成長は5 ‘→3’方向に生成されます。なぜなら、これはDNTPのαリン酸塩に対する求核攻撃を実行するものであるため、合成のαリン酸塩を実行するため、合成のαリン酸塩を実行するため、合成のα-OHイニシエーターを必要とするため、合成の開始には5 ‘→3’方向に生成されます。プライマーの3 ‘端はプライマーと呼ばれます。
ポリメラーゼDNAは、複製プロセス中に他の機能も実行します。伸長に参加することに加えて、彼らは拡散活動3 ‘のおかげで修正および修復機能を実行します。そうでなければ、DNAのコピー中に生成されたエラーが突然変異をもたらすため、これらの補正メカニズムが存在することが重要です。
DNAポリメラーゼは、形成中のDNA鎖の3 ‘末端にのみ遊離ヌクレオチドを加えることができます。これにより、フォーメーション内のチェーンの延長が5’-3 ‘アドレスで実行されます。既知のポリメラーゼDNAは、DNA鎖を再び開始することはできません。つまり、2番目のヌクレオチドを鎖に、次に3番目のヌクレオチドを結合した後、鎖鎖に配置することはできません。彼らにできることは、3’-OH端の既存のチェーンを長くすることです。そのため、常に最初のフラグメントが形成されている必要があります。これが、DNAポリメラーゼがヌクレオチドを追加するために事前に形成されたプライマーを必要とする理由です。プライマーは、RNAまたはDNAによって形成できます。 DNA複製では、最初の2つの塩基は常にARNであり、PRIMAと呼ばれる別の酵素によって合成されます。ヘリカーゼと呼ばれる酵素は、DNAを解き、その領域の二重鎖構造を元に戻し、2つの単純な分岐鎖(この領域は複製フォーク)の構造を形成するために必要です。
一部のポリメラーゼDNAが持っている別の特性は、すべてではありませんが、エラーの修正です。このプロセスは、ネオシンテット化されたDNAの形成中に生成されたエラーを修正します。誤って配置されたベースペアが認識されると、DNAポリメラーゼは、一対の塩基を遅らせることにより、動きの方向を投資します。酵素のこのエキソヌクレアーゼ活性3′-5 ‘は、誤った塩基のペアを排除し、複製を継続するポリメラーゼ自体に正しいものに置き換えることができます。このアクティビティはテスト補正と呼ばれます。 DNAポリメラーゼは、分子生物学実験で広く使用されています。
DNAポリメラーゼには非常に保存された構造があります。つまり、触媒サブユニットは種ごとにほとんど変化しません。保存された構造は一般に、セル内で重要でかけがえのない機能を実行し、そのメンテナンスは一連の利点を生み出します。
構造 [ 編集します ]
既知のポリメラーゼDNAには非常に保存された構造があります。つまり、ドメイン構造に関係なく、それらの一般的な触媒サブユニットは種ごとにほとんど変化しません。保存された構造は通常、細胞の重要でかけがえのない機能を示しており、そのメンテナンスは進化的な利点を提供します。形状は、親指、指、手のひらのドメインを備えた右手として説明できます。手のひらの領域は、ホスホリル移動反応におけるホスホリル基の移動を触媒するように機能しているようです。 DNAは、酵素が活性であるときに手のひらに結合します。この反応は2つの金属イオンメカニズムによって触媒されると考えられています。指の習得は、テンプレートのベースを使用して、トリリン酸塩ヌクレオシドを統合するために機能します。プルガードメインは、加工性、転座、およびDNAの位置付けに潜在的な役割を果たします。
加工性 [ 編集します ]
ポリメラーゼの迅速な触媒は、その加工性によるものです。加工性は、高分子基質で機能する酵素の特徴です。 DNAポリメラーゼの場合、酵素がテンプレートに結合するたびに追加されるヌクレオチド数の平均ヌクレオチド数を指します。平均ポリメラーゼDNAには、プライマー /金型を見つけて結合するには、約1秒が必要です。結合すると、非加速ポリメラーゼDNAがヌクレオチド速度でヌクレオチドを1秒間に追加します。ただし、手順ポリメラーゼDNAは1秒あたり複数のヌクレオチドを追加し、DNAの合成速度を劇的に増加させます。加工の程度は、DNA合成率に直接比例します。生細胞のDNA合成率は、バクテリオファージに感染した細菌のDNA伸長率T4として最初に決定されました。 37°CでのDNAの指数増加期間中、速度は毎秒749ヌクレオチドでした。
DNAポリメラーゼがDNAテンプレートに沿ってスライドする能力により、より大きなプロセシビニンが可能になります。複製分岐の加工性の劇的な増加があります。この増加は、DNAポリメラーゼとDNAスライドクランプとして知られるタンパク質との関連によって促進されます。ピンセットは、リングの形で関連するタンパク質の複数のサブユニティです。 ATP加水分解を使用して、スライドクランプとして知られるタンパク質クラスは、DNAスライドクランプの環構造を開き、結合とDNA鎖の放出を可能にします。タンパク質間相互作用クランプは、DNAポリメラーゼがDNAテンプレートから普及することを防ぎます。これにより、酵素は同じプライマー/テンプレートユニオンの1つであることを保証し、複製を継続します。 DNAポリメラーゼは適合を変化させ、それに関連するときにクランプの親和性を高め、クランプの解放を可能にするためにDNAのセクションの複製を完了するときに親和性を低下させます。
ポリメラーゼ連鎖反応 [ 編集します ]
DNAポリメラーゼの所有権は、DNA鎖を複製して、PCRとして知られるポリメラーゼ連鎖反応に使用され、特定のDNAフラグメントの多数のコピーを取得し、研究目的で増幅します。 PCRプロセスでは、DNA分子を変性化するために高温を適用する必要があるため、Taqポリメラーゼなどの熱安定性ポリメラーゼの使用が必要です。
ポリメラーゼDNAファミリー [ 編集します ]
その構造の相同性とアミノ酸配列DNAポリメラーゼは、6つのファミリーに分類できます。 DNAウイルスのDNAポリメラーゼは、ウイルス配列の多様性が高いため、以下のグループで分類することが困難です。
- 家族a :incluye las adn polimerasas eucaryatasγ、θ、ν、la procarita i y la viral t7
- 家族b :Dn Polimas Eucariotsζ、α、Δ、Δ、δ、ε、およびProcariots IIを含む。また、いくつかのDn Pomeras Viralesを含めることによって提案されています。
- 家族d :DNAポリメラーゼprocariota Dを含み、いくつかのウイルスポリメラーゼDNAの包含も提案されています。
- 家族x :incluye las adn polimerasas eucariotsβ、σ、λ、λ、μ、tdt y la procariota IIII。
- 家族と :真核生物のポリメラーゼDNA /を含み、ηおよび原核生物IVおよびVを含む。
- ファミリーRT :真核生物の排他的なテロメラーゼとウイルス逆、真核生物、原核生物転写産物が含まれます。
原核生物ポリメラーゼDNA [ 編集します ]

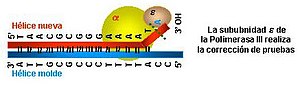
原核生物のポリメラーゼDNA(アーチと細菌)は次のとおりです。ポリシーI、II、V、V、B、D、およびRTはそれぞれ、複製における彼らの役割が何であるかに従って、これらの機能の1つ以上に特化しています。プロダリオ酸性ポリメラーゼは、中心ポリメラーゼとホロ酵素の2つの方法で存在します。中央ポリメラーゼはDNAテンプレートからDNAを合成しますが、合成を単独または正確に開始することはできません。ホロ酵素は精度で合成を開始します。
pol i [ 編集します ]
原核生物ポリメラーゼ科には、Pola遺伝子によってコードされ、原核生物の中で遍在するDNAポリメラーゼI(Pol I)酵素が含まれます。この補償ポリメラーゼは、エキソヌクレアーゼ活性3′-5 ‘および5’-3’による分割補償と、遅延鎖の合成中に生成された岡崎フラグメントの処理に関与します。 [ 初め ] Pol Iは最も豊富なポリメラーゼであり、大腸菌のポリメラーゼ活性の95%以上を表しています。しかし、細胞はポリを欠いていることがわかっており、活性が他の4つのポリメラーゼに置き換えることができることを示唆しています。 pol iは1秒あたり〜15-20ヌクレオチドを追加し、したがって、加工性が低いことを示しています。代わりに、Pol Iは、RNAプライマー:The Replication Origin(ORI)として知られるテンプレートユニオンにヌクレオチドを追加し始めます。起源の下流で約400 pbのホロ酵素Pol IIIが組み立てられ、非常に手続き的な速度と性質で複製を処理します。 [ 2 ]
TAQポリメラーゼは、証拠補正能力を欠いているこのファミリーの熱安定性酵素です。 [ 3 ]
Pol II [ 編集します ]
DNAポリメラーゼIIは、POLB遺伝子によってコードされるファミリーBのポリメラーゼです。 Pol IIはエキソヌクレアーゼ活性3′-5 ‘を持ち、DNAの修復、怪我を避けるための複製の再起動に関与し、その細胞の存在は、SOSの誘導中に細胞Aごとの約200〜300コピーから渡ることができます。また、Pol IIはPol IIIバックアップであると考えられています。これは、ホロエンツィマティックタンパク質と相互作用し、高レベルの加工性を想定できるためです。 Pol IIの主な役割は、複製分岐におけるポリメラーゼの活性を指示する能力であり、Pol III派生の末端の意見の不一致を止めるのに役立つと考えられています。 [ 4 ]
ポリII [ 編集します ]
DNAポリメラーゼIIIのホロ酵素は、原核生物のDNAの複製に関与する主な酵素であり、ポリメラーゼCのファミリーに属します。核は、3つのサブユニットで構成されています:α、ポリメラーゼ活性中心、ɛ、エキソヌクレ石器時代の試験、およびθは、ɛの安定剤として機能します。ベータスライダーの加工係数は、各核に1つずつ重複して存在し、DNAを囲み、高い加工性を可能にするクランプを作成します。 [ 5 ] 3番目のアセンブリは、7つのサブユニット(τ2γΔROCY)のピンセットの複雑な充電器です。
教科書の古い「トロンボーンモデル」は、各複製フォーク(RF)の中心酵素から2つの同等物を持つ伸長複合体、各ストランド、1つ、ラグ、前進を表しています。 [ 4 ] しかし、単一の分子研究の最近の証拠は、Pol IIIとB. subtilis、POLCの対応物の両方について、各RFの中心酵素の3つの化学量論的相当量の平均を示しています。 [ 6 ] 細胞内の蛍光顕微鏡検査により、主鎖の合成は完全に連続していない可能性があることが明らかになり、Pol III*(つまり、スライディングクランプβ2なしでホロ酵素のサブユニットα、ε、τ、δ、χ)が活性rFの解離の周波数を高くしています。 [ 7 ] これらの研究では、複製フォークの回転速度は、Pol III *で約10秒、スライドクランプβ2で47秒、ヘリカーゼDNABで15 mでした。これは、ヘリカーゼDNABがRFで安定したままであり、有能なホロ酵素の核生成点として機能することを示唆しています。単一の分子のin vitro研究では、Pol III *のRF更新速度が過剰であることが示されていますが、濃度が制限されている場合は複製フォークで安定したままです。 [ 7 ] 単一の分子の別の研究では、ADNBのヘリカーゼの活性と鎖の伸長が確率的速度論的分離で進行できることが示されました。 [ 7 ]
Pol IV [ 編集します ]
DNAポリメラーゼIV(POL IV)は、指示されていない突然変異誘発に関与する誤差を起こしやすいDNAポリメラーゼです。 [ 8 ] Pol IVはファミリーポリメラーゼであり、複製分岐における停滞ポリメラーゼによって引き起こされるSOSの誘導によって活性化されるDINB遺伝子によって発現されます。 SOSの誘導中、ポリIVの産生に10を掛けて、この期間中の機能の1つはホロ酵素Pol IIIの加工性を妨げることです。これにより、制御点が作成され、複製を停止し、適切な修復経路を介してDNA病変を修復する時間が与えられます。 [ 9 ] 別のPOL IV機能は、非損傷DNAを横切るよりも速いペースでN2-デソキシグアニンの付加物を回避するなど、停滞複製の分岐におけるトランスリクチャの合成を行うことです。 DINB遺伝子を欠く細胞は、DNAを損傷する薬剤によって引き起こされる変異誘発率が高い。 [ 十 ]
半分 [ 編集します ]
DNAポリメラーゼV(POL V)はファミリーDNAポリメラーゼであり、SOS応答と翻訳合成のDNAを修復するメカニズムに関与しています。 [ 11 ] UMUDC遺伝子を介したPol Vの転写は、細胞に損傷したDNAが存在し、SOSの回答を生成する場合、Pol Vのみを生成するように高度に調節されています。停滞したポリメラーゼはssDNAになめます。これにより、レクサタンパク質が自動的に消化されます。その後、LexaはUMUDC演算子の転写を抑制する能力を失います。集合的な核タンパク質の入札は、umud umudタンパク質タンパク質を修正します。 umudとumud ‘は、umucと相互作用するヘテロダイマーを形成し、それが損傷したDNAにおけるumucポリメラーゼの触媒活性を活性化します。 [ 12番目 ] ポリメラーゼの「ツールベルト」は、両方のポリメラーゼが同時にβクランプに結合される、停止したフォークでPol IVのPol IIIを変更するように提案されています。 [ 13 ] ただし、怪我を避けるために連続して動作する複数のTLSポリメラーゼの参加は、大腸菌ではまだ実証されていません。さらに、POL IVは挿入と高効率の拡張の両方を触媒することができますが、POL Vは主要なSOS TLSポリメラーゼと見なされます。例としては、2つのポリメラーゼの変異片の違いに基づいて、実証された脳胎中ティミナグアニンの交差程度の導出があります。 [ 13 ]
家族d [ 編集します ]
1998年、ポリメラーゼDNAのDNAファミリーが発見されました。 [ 14 ] Pold複合体は2鎖ヘテロダイマーで、それぞれがDP1(小さな補正)とDP2(大きな触媒)によってエンコードされています。他のポリメラーゼDNAとは異なり、DP2触媒核の構造とメカニズムは、複数のサブユニットポリメラーゼの構造に似ています。 DP1-DP2インターフェイスは、クラスB真核ポリメラーゼの亜鉛フィンガーとその小さなサブユニットに似ています。 MRE11とは異なるEX -LIMAであるDP1は、おそらくポリαとεの小さなサブユニットの前駆体であり、真核生物で失われた証明補正能力を提供します。そのN末端HSHドメインは、構造内のAAAタンパク質、特にΔおよびPol III RUVBサブユニットに類似しています。 DP2にはクラスII khドメインがあります。 Poldは、TAQポリメラーゼよりも熱く、より安定しており、より正確ですが、まだ販売されていません。ファミリーDのDNAポリメラーゼが細胞生物で最初に進化したこと、および最後の普遍的な共通の祖先(luca)の複製ポリメラーゼがD. D.に属していることが示唆されています。 [ 15 ]
逆転写酵素(RT) [ 編集します ]
これは、RNAテンプレートからのDNAを合成するRNA依存性ポリメラーゼ(RDDP)DNAです。逆転写酵素ファミリーには、DNAポリメラーゼの機能とHarna Hの機能性の両方が含まれており、DNAと一致するRNAを分解します。逆転写酵素は、研究目的でRNA増幅で一般的に使用されています。 RNAテンプレートを使用して、PCRは逆転写酵素を使用してDNAテンプレートを作成できます。この新しいDNAテンプレートは、典型的なPCR増幅に使用できます。したがって、そのような実験の産物は、RNAからのPCR産物を増幅します。
逆転写には、ゲノムの2つのコピー間のテンプレートの変化(コピーのコピーによる組換え)が伴います。各複製サイクルで5〜14のゲノム組換えイベントが発生します。テンプレートの変化(再結合)は、ゲノムの完全性を維持し、損傷したゲノムを保存するための修復メカニズムとして必要と思われます
真核生物のDNAポリメラーゼ [ 編集します ]
ユーカリオットにはdn polimeraのタイプミスがあります:ユーカリオットはユーカリオットです:ユーカリオットはユーカリオットです:ポルζ、α、α、α、β、β、β、λ、λ、λ、μ、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω、ω ω、η、 Spring(DNA分子分子分子の一部であるα)ARNのSyretiza Cebbatorsと、本のDNのDN DNを使用してolouringを追加します。それは、ポルメーターの変化、次にDn Polimeraσの変化によって生成されます。これは合成を継続します。
ポリエメラサスβ、λ、σ、μ(ベータ、ラムダ、シグマ、MU)y tdt [ 編集します ]
ファミリーXポリメラーゼには、よく知られている真核生物ポリメラーゼPolβ(ベータ)、およびPolσ(Sigma)、Polλ(Lambda)、Polμ(Mu)、Dexinucleogidos末端トランスフェラーゼ(TDT)などの他の真核ポリメラーゼが含まれています。ファミリーXポリメラーゼは主に脊椎動物で、いくつかは植物や菌類に見られます。これらのポリメラーゼには、DNAポリメラの相互作用に不可欠なヘリックスホルキーラヘリックスによる2つの理由を含む非常に保存された領域があります。理由は、下流のDNAと相互作用する8 kDaドメインにあり、理由はプライマーチェーンと相互作用する親指ドメインにあります。 PolB遺伝子によってコードされるPolβは、賃貸または酸化塩基、および非行サイトを修復するために不可欠なDNA修復ルートである短いパッチベーススプリットによる修復に必要です。それぞれ世論調査およびPolM遺伝子によってコードされるpolλおよびPolμは、それぞれ過酸化水素とイオン化放射線によるDNA二重鎖の破損を再分うとするメカニズムである非相同端の結合に関与しています。 TDTはリンパ組織でのみ発現し、免疫の多様性を促進するために再結合v(d)jの間に形成された二重鎖破壊に「nヌクレオチド」を加えます。 [ 16 ]
Polimerasas A、d y e(alfa、deltayépsilon) [ 編集します ]
Polα(Alfa)、PolΔ(Delta)、Polε(Epsilon)はBファミリーのポリメラーゼのメンバーであり、核DNAの複製に関与する主なポリメラーゼです。 Polα複合体(PRIMAPolα-ADN複合体)は、それぞれPola1触媒サブユニット、Pola2調節サブユニット、および大小プリマサブユニットPRIM1およびPRIM2の4つのサブユニットで構成されています。 PRIMAがRNAプライマーを作成すると、Polαは複製を開始し、〜20ヌクレオチドでプライマーを延長します。そのプロセス性が高いため、PolΔは、ポリαの主なおよび遅延polycsの合成を処理します。 polΔはPold1遺伝子によって発現され、触媒サブユニット、Pold2、Pold3、およびPold4を作成します。PolΔがプロセス性を持つDNAクランプである増殖細胞(PCNA)と相互作用する他のサブユニットを作成します。 Polεは、極性サブユニット、極2および極3によってコード化されています。 polε関数は複製中にメインチェーンを拡張することであり、polδは主に遅れの遅れを複製することが報告されています。しかし、最近の証拠は、PolΔが主要なDNA鎖の複製にも役割を果たしている可能性があることを示唆しています。ポリメラーゼ活性には不要であるにもかかわらず、Polε「ポリメラーゼRelic」のC末端領域は、細胞の活力に不可欠であると考えられています。 C末端領域は、後期に入る前に制御点を提供し、ホロ酵素の安定性を提供し、複製の開始に必要なホロ酵素にタンパク質を追加すると考えられています。 POLεには、PCNAに関係なく高い加工性を提供するより大きな「パーム」ドメインがあります。
Bファミリーの他のポリメラーゼと比較して、エキソヌクレアーゼのファミリーは、証拠の補正に関与する専用のファミリーは、ポリαで不活性化されています。 Polεは、亜鉛指と彼のC末端にBファミリーの別のPポリメラーゼの非アクティブコピーを持つ2つのドメインを持っているという意味でユニークです。この亜鉛フィンガーの存在は、ユーカリオタの起源に影響を及ぼします。この場合、これはArcheal Polymerase B3を含むAsgardグループに配置されます。
Polimerasas H、I Y K(Eta、Iota Y Kappa) [ 編集します ]
Polη(ETA)、Polι(IOTA)およびPolκ(Kappa)はファミリーポリメラーゼDNAであり、翻訳合成によるDNAの修復に関与し、それぞれポリ、ポリ、ポーク遺伝子によってエンコードされています。家族と、プライマーの基質と端を統合するのを支援する5つの共通の理由があり、すべてが親指、手のひら、指の典型的なドメイン、右手の指の典型的なドメイン、小指(LF)、ポリメラーゼ(PAD)または人形に関連するドメインなどの骨材ドメインを含む右手の典型的なドメインが含まれています。しかし、活性サイトは、修復された異なる怪我のために家族間で異なります。ファミリーのポリメラーゼは低い忠誠心ポリメラーゼですが、ポリメラーゼに影響を与える変異が皮膚がんやX腸色素症(XPS)の変異体などのさまざまな疾患を引き起こす可能性があるため、より良いことが示されています。これらのポリメラーゼの重要性は、DNAポリメラーゼηをコードする遺伝子がXPVとして知られているという事実によって証明されています。これは、この遺伝子の喪失が疾患のX腸皮症の亜型色素をもたらすためです。紫外線に起因するDNA損傷の正確な翻訳合成を可能にするためには、Polηは特に重要です。 POLκ機能は完全には理解されていませんが、研究者は2つの可能性のある機能を発見しました。 POLκは、特定のDNA病変の特定の塩基の伸筋または挿入物として作用すると考えられています。翻訳合成の3つのポリメラーゼは、Rev1とともに、停滞した複製ポリメラーゼを介して損傷した病変で動員されます。損害を修復する方法は、研究者が選択されたルートは、どの鎖が損傷、メインストランド、または遅れを含んでいるかに依存すると結論付けるように導く2つの方法があります。
PolimerasRev1ζ(ZUT) [ 編集します ]
Bファミリーの他のポリメラーゼは、2つのサブユニットRev3、触媒サブユニット、およびRev7(MAD2L2)によって形成され、ポリメラーゼの触媒機能を増加させ、翻訳の合成に関与します。 polζそれはエキソヌクレアーゼ活性3 ‘から5’を欠いていますが、末端の不一致でプライマーを拡張できるため、ユニークです。 Rev1は、BRCTドメインに関心のある3つの領域、ユビキチン接合部の習得、およびC末端ドメインを持ち、DCMP移動能力を持ち、ポリメラーゼのポリメアスを停止するデキシキシシジンの反対の損傷を追加します。これらの停滞したポリメラーゼは、ユビキチン複合体を活性化し、それが複製ポリメラーゼを解離し、PolζおよびRev1を動員します。一緒に、PolζとRev1はデソキシシチジンを追加し、Polζは病変を超えて伸びます。まだ決定されていないプロセスを通じて、Polζの解離と複製ポリメラーゼが再加工され、複製が継続されます。 PolζとRev1は複製には必要ありませんが、Gemation酵母におけるRev3遺伝子の喪失は、複製ポリメラーゼが停滞している複製フォークの崩壊によりDNAに損傷を与える薬剤に対してより大きな感受性を引き起こす可能性があります。
ポリエメラサγ、θyνν(ガンマ、シータy nu) [ 編集します ]
Polγ(ガンマ)、Polθ(Theta)、およびPolν(Nu)はファミリーAのポリメラーゼです。長い間、PolG遺伝子によってコードされたPolγが唯一のミトコンドリアポリメラーゼであると考えられていました。しかし、最近の研究では、Xファミリーのポリメラーゼである少なくともPolβ(ベータ)がミトコンドリアにも存在することが示されています。機能しない限られたγPOLγを産生する突然変異は、ADNMTに大きな影響を及ぼし、常染色体遺伝性ミトコンドリア障害の最も一般的な原因です。 POLγには、C末端ポリメラーゼのドメインと、リンクされた領域を介して接続されたN末端のEx末端ドメイン3′-5 ‘が含まれており、これはアクセサリーサブユニットに結合します。アクセサリーサブユニットはDNAに結合し、ポリジロの加工性に必要です。リンクされた領域のA467T時間厳守変異は、ポリγに関連するすべてのミトコンドリア障害の3分の1以上の原因です。多くのpolqgenコード化されたホモログは真核生物に見られますが、その機能は明確に理解されていません。最後のcのアミノ酸配列は、polθをファミリーAのポリメラーゼとして分類するものですが、polθ誤差率はファミリーYのポリメラーゼにより密接に関連しています。また、ポリメラーゼのドメインにデオキシリボホスホジエステラーゼ活性(DRPASA)を持ち、ssDNAに近接して密接な活性を示すことができます。 Polν(Nu)は、ポリメラーゼ酵素の最も効果的ではないと考えられています。ただし、Nu DNAは、クロスボンドに対する細胞応答中に相同性を修復する上で積極的な役割を果たします。
逆転写酵素(RT) [ 編集します ]
これは、RNAテンプレートからのDNAを合成するRNA依存性ポリメラーゼ(RDDP)DNAです。逆転写酵素ファミリーには、DNAポリメラーゼの機能とHarna Hの機能性の両方が含まれており、DNAと一致するRNAを分解します。逆転写酵素は、研究目的でRNA増幅で一般的に使用されています。 RNAテンプレートを使用して、PCRは逆転写酵素を使用してDNAテンプレートを作成できます。この新しいDNAテンプレートは、典型的なPCR増幅に使用できます。したがって、そのような実験の産物は、RNAからのPCR産物を増幅します。
逆転写には、ゲノムの2つのコピー間のテンプレートの変化(コピーのコピーによる組換え)が伴います。各複製サイクルで5〜14のゲノム組換えイベントが発生します。テンプレートの変化(再結合)は、ゲノムの完全性を維持し、損傷したゲノムを救うための修復メカニズムとして必要と思われます。
テロメラーゼ [ 編集します ]
テロメラーゼは、通常のポリメラーゼDNAが端やテロメアを再現できないため、線形染色体の端を再現するのに役立つ酵素です。 5′-TTAGGG-3 ‘シーケンスリクルートテロメラーゼを使用した2回の染色体の発信3’単catenary。テロメラーゼは、3 ‘端を伸ばすと他のDNAポリメラーゼのように作用しますが、他のポリメラーゼDNAとは異なり、テロメラーゼはテンプレートを必要としません。逆転写酵素の例であるサブサブユニットは、RNAサブユニットを使用して、テロメラーゼが染色体端の3 ‘末端を伸ばすことを可能にするプライマーとモールド接合部を形成します。生涯を通じて多くの複製の結果としてテロメアのサイズが徐々に減少することは、老化の影響に関連していると考えられています。
ウイルスポリメラーゼDNA [ 編集します ]
DNAウイルスは、細胞ポリメラーゼDNAまたは他のDNAウイルスのものとほとんど関連していないさまざまなDNAポリメラーゼを合成します。 HIVなどのレトロトランススクリプトウイルスは、互いに関連する逆転写酵素を合成することによって特徴付けられ、特に真核生物を使用した細胞逆転写産物を使用します。
ファージφ29は、独自のポリメラーゼDNAを合成します。この酵素は、複数のDNA増幅変位手順の分子生物学で広く使用されており、このアプリケーションに特に適した一連の特性があります。
参照 [ 編集します ]
- ↑ ヒューシャー、ウルリッヒ。 (2010)。 DNAポリメラーゼ:細胞DNAトランザクションの発見、特性評価、および機能 。ワールドサイエンティフィック。 ISBN 978-981-4299-17-6 。 OCLC 670430601 。 2021年1月31日に取得 。
- ↑ キャンプ、マネル;ローブ、ローレンスA.(2004-02)。 «Polがハイギアに入るとき:細胞内のPol Iによる加工DNA合成» 。 細胞周期 (英語で) 3 (2):114-116。 ISSN 1538-4101 。 doi: 10.4161/cc.3.2.651 。 2021年1月31日に取得 。
- ↑ 犬、A。;エドガー、D。B。; Trela、J。M。(1976年のセプティエンブレの1番目)。 «極端なThermophile thermus aquaticusからのデオキシリボ核酸ポリメラーゼ。» 。 Journal of Bacteriology (英語で) 127 (3):1550-1557。 ISSN 0021-9193 。 PMID 8432 。 doi: 10.1128/jb.127.3.1550-1557.1976 。 2021年1月31日に取得 。
- ↑ a b Banach -Orlowska、Magdalena; Fijalkowska、Iwona J。; Schaaper、Roel M。; Jonczyk、Piotr(2005)。 «Escherichia coliの染色体DNA合成の忠実度因子としてのDNAポリメラーゼII» 。 分子微生物学 (英語で) 58 (1):61-70。 ISSN 1365-2958 。 doi: 10.1111/j.1365-2958.2005.04805.x 。 2021年1月31日に取得 。
- ↑ オルソン、マシューW。; Dallmann、H。Garry; Mchenry、Charles S.(1995-12)。 «Escherichia coli DNAポリメラーゼIIIホロエンザイムのDNAX複合体χ・ψ» 。 Journal of Biological Chemistry 270 (49):29570-29577。 ISSN 0021-9258 。 doi: 10.1074/jbc.270.49.29570 。 2021年1月31日に取得 。
- ↑ liao、yi; li、yilai; Schroeder、Jeremy W。; Simmons、Lyle A。; Buteen、Julie S.(20 de diciembre de 2016)。 «生細胞の細菌レプリソームでの単一分子DNAポリメラーゼダイナミクス» 。 生物物理学ジャーナル (英語で) 111 (12):2562-2569。 ISSN 0006-3495 。 PMID 28002733 。 doi: 10.1016/j.bpj.2016.11.006 。 2021年1月31日に取得 。
- ↑ a b c «細菌レプリソーム» 。 構造生物学における現在の意見 (英語で) 53 :159-168。 2018年12月1日。 ISSN 0959-440x 。 doi: 10.1016/j.sbi.2018.09.006 。 2021年1月31日に取得 。
- ↑ グッドマン、マイロンF.(2002年6月1日)。 «原核生物と真核生物の誤差が発生しやすいDNAポリメラーゼ» 。 生化学の年次レビュー 71 (1):17-50。 ISSN 0066-4154 。 doi: 10.1146/annurev.biochem.71.083101.124707 。 2021年1月31日に取得 。
- ↑ Mori, Tetsuya; Nakamura, Tatsuro; Okazaki, Naoto; Furukohri, Asako; Maki, Hisaji; Akiyama, Masahiro Tatsumi (2012). «Escherichia coli DINBは、SOS応答を大幅に誘導することなく複製フォークの進行を阻害します» 。 遺伝子 ^ | ^遺伝システム 八十七 (2):75-87。 ISSN 1341-7568 。 doi: 10.1266/ggs.87.75 。 2021年1月31日に取得 。
- ↑ Jarosz、Daniel F。; Godoy、Veronica G。;ウォーカー、グラハムC.(2007年4月1日)。 «DINB DNAポリメラーゼによる持続性DNA病変の熟練した正確なバイパス» 。 細胞周期 6 (7):817-822。 ISSN 1538-4101 。 PMID 17377496 。 doi: 10.4161/cc.6.7.4065 。 2021年1月31日に取得 。
- ↑ Patel、Meghna; Jiang、Qingfei; Woodgate、Roger; Cox、Michael M。; Goodman、Myron F.(1 De Junio de 2010)。 «SOS誘発性変異誘発の新しいモデル:RECAタンパク質がDNAポリメラーゼVを活性化する方法V» 。 生化学と分子生物学の批判的レビュー 45 (3):171-184。 ISSN 1040-9238 。 PMID 20441441 。 doi: 10,3109/10409238.2010.480968 。 2021年1月31日に取得 。
- ↑ サットン、マークD。;ウォーカー、グラハムC.(17 de Julio de 2001)。 «DNAポリメラーゼの管理:DNA複製、DNA修復、およびDNA組換えの調整» 。 国立科学アカデミーの議事録 (英語で) 98 (15):8342-8349。 ISSN 0027-8424 。 PMID 11459973 。 doi: 10.1073/pnas.111036998 。 2021年1月31日に取得 。
- ↑ a b Raychaudury、パロミータ; Basu、Ashis K.(29 De Marzo de 2011)。 «大腸菌のG [8,5-Me] T架橋の突然変異誘発の遺伝的要件:DNAポリメラーゼIVとVは、エラーが発生しやすいバイパスを競います» 。 生化学 50 (12):2330-2338。 ISSN 0006-2960 。 PMID 21302943 。 doi: 10.1021/bi102064z 。 2021年1月31日に取得 。
- ↑ Ishino, Yoshizumi; Komori, Kayoko; Cann, Isaac K. O.; Koga, Yosuke (15 de abril de 1998). «inarchaeaを発見した新しいDNAポリメラーゼファミリー» 。 Journal of Bacteriology (英語で) 180 (8):2232-2236。 ISSN 0021-9193 。 PMID 9555910 。 doi: 10.1128/jb.180.8.2232-2236.1998 。 2021年1月31日に取得 。
- ↑ Koonin, Eugene V.; Krupovic, Mart; Ishino, Sonoko; Ishino, Yoshizumi (9 de junio de 2020). «ルカの複製機械:DNA複製と転写の一般的な起源» 。 BMC生物学 18 (1):61。 ISSN 1741-7007 。 PMID 32517760 。 doi: 10.1186/s12915-020-00800-9 。 2021年1月31日に取得 。
- ↑ ヤムチッチ、ジェニファー。 Swasy、Joann B.(2010年5月1日)。 «DNAポリメラーゼファミリーX:機能、構造、細胞の役割» 。 Biochimica et Biophysica Acta(BBA) – タンパク質とプロテオミクス 。 DNAポリメラーゼ:構造と機能 (英語で) 1804 (5):1136-1150。 ISSN 1570-9639 。 PMID 19631767 。 doi: 10.1016/j.bbapap。2009.07.008 。 2021年1月31日に取得 。
書誌 [ 編集します ]
‘ 複製 »。
Recent Comments