Genetische Drift – Wikipedia
Die Änderung der Häufigkeit einer vorhandenen Genvariante in einer Population
Genetische Drift (auch bekannt als Alleldrift oder der Sewall Wright-Effekt)[1] ist die Änderung der Häufigkeit einer vorhandenen Genvariante (Allel) in einer Population aufgrund einer zufälligen Stichprobe von Organismen.[2] Die Allele in den Nachkommen sind eine Stichprobe der Allele in den Eltern, und der Zufall spielt eine Rolle bei der Bestimmung, ob ein bestimmtes Individuum überlebt und sich fortpflanzt. Die Allelfrequenz einer Population ist der Anteil der Kopien eines Gens, die eine bestimmte Form haben.[3]
Genetische Drift kann dazu führen, dass Genvarianten vollständig verschwinden und dadurch die genetische Variation verringern.[4] Es kann auch dazu führen, dass anfangs seltene Allele viel häufiger und sogar fixiert werden.
Wenn es nur wenige Kopien eines Allels gibt, ist der Effekt der genetischen Drift größer, und wenn es viele Kopien gibt, ist der Effekt geringer. Mitte des 20. Jahrhunderts gab es heftige Debatten über die relative Bedeutung der natürlichen Selektion gegenüber neutralen Prozessen, einschließlich der genetischen Drift. Ronald Fisher, der die natürliche Selektion mithilfe der Mendelschen Genetik erklärte,[5] vertrat die Ansicht, dass genetische Drift in der Evolution höchstens eine untergeordnete Rolle spielt, und dies blieb über mehrere Jahrzehnte die vorherrschende Ansicht. 1968 entfachte der Populationsgenetiker Motoo Kimura die Debatte mit seiner neutralen Theorie der molekularen Evolution, die besagt, dass die meisten Fälle, in denen sich eine genetische Veränderung über eine Population ausbreitet (obwohl dies nicht unbedingt die Phänotypen ändert), durch genetische Drift verursacht werden, die auf neutrale Mutationen einwirkt.[6][7]
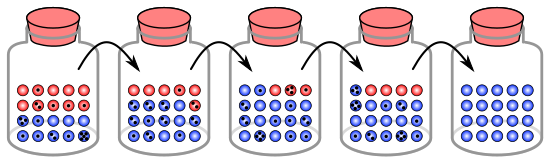
Analogie mit Murmeln im Glas[edit]
Der Prozess der genetischen Drift kann anhand von 20 Murmeln in einem Glas veranschaulicht werden, um 20 Organismen in einer Population darzustellen.[8] Betrachten Sie dieses Glas Murmeln als die Ausgangspopulation. Die Hälfte der Murmeln im Glas ist rot und die Hälfte blau, wobei jede Farbe einem anderen Allel eines Gens in der Population entspricht. In jeder neuen Generation vermehren sich die Organismen zufällig. Um diese Reproduktion darzustellen, wählen Sie zufällig einen Marmor aus dem Originalglas aus und legen Sie einen neuen Marmor mit derselben Farbe in ein neues Glas. Dies ist der “Nachwuchs” des ursprünglichen Marmors, was bedeutet, dass der ursprüngliche Marmor in seinem Glas verbleibt. Wiederholen Sie diesen Vorgang, bis sich 20 neue Murmeln im zweiten Glas befinden. Das zweite Glas enthält jetzt 20 “Nachkommen” oder Murmeln in verschiedenen Farben. Sofern das zweite Gefäß nicht genau 10 rote und 10 blaue Murmeln enthält, ist eine zufällige Verschiebung der Allelfrequenzen aufgetreten.
Wenn dieser Vorgang mehrmals wiederholt wird, schwankt die Anzahl der von jeder Generation gepflückten roten und blauen Murmeln. Manchmal hat ein Glas mehr rote Murmeln als sein “Eltern” -Glas und manchmal mehr blaue. Diese Fluktuation ist analog zur genetischen Drift – eine Änderung der Allelfrequenz der Population, die sich aus einer zufälligen Variation der Allelverteilung von einer Generation zur nächsten ergibt.
Es ist sogar möglich, dass in einer Generation keine Murmeln einer bestimmten Farbe ausgewählt werden, was bedeutet, dass sie keine Nachkommen haben. Wenn in diesem Beispiel keine roten Murmeln ausgewählt sind, enthält das Glas der neuen Generation nur blaue Nachkommen. In diesem Fall ist das rote Allel dauerhaft in der Bevölkerung verloren gegangen, während das verbleibende blaue Allel fixiert ist: Alle zukünftigen Generationen sind vollständig blau. In kleinen Populationen kann die Fixierung in nur wenigen Generationen auftreten.
Wahrscheinlichkeit und Allelfrequenz[edit]
Die Mechanismen der genetischen Drift können anhand eines vereinfachten Beispiels veranschaulicht werden. Stellen Sie sich eine sehr große Bakterienkolonie vor, die in einem Tropfen Lösung isoliert wurde. Die Bakterien sind bis auf ein einziges Gen mit zwei markierten Allelen genetisch identisch EIN und B.. EIN und B. sind neutrale Allele, was bedeutet, dass sie die Überlebens- und Fortpflanzungsfähigkeit der Bakterien nicht beeinträchtigen; Alle Bakterien in dieser Kolonie überleben und vermehren sich gleichermaßen. Angenommen, die Hälfte der Bakterien hat ein Allel EIN und die andere Hälfte hat Allel B.. So EIN und B. jedes hat die Allelfrequenz 1/2.
Der Tropfen der Lösung schrumpft dann, bis er nur noch genug Nahrung hat, um vier Bakterien zu ernähren. Alle anderen Bakterien sterben ab, ohne sich zu vermehren. Unter den vier Überlebenden gibt es 16 mögliche Kombinationen für die EIN und B. Allele:
(AAAA), (BAAA), (ABAA), (BBAA),
(AABA), (BABA), (ABBA), (BBBA),
(AAAB), (BAAB), (ABAB), (BBAB),
(AABB), (BABB), (ABBB), (BBBB).
Da alle Bakterien in der ursprünglichen Lösung gleich wahrscheinlich überleben, wenn die Lösung schrumpft, sind die vier Überlebenden eine Zufallsstichprobe aus der ursprünglichen Kolonie. Die Wahrscheinlichkeit, dass jeder der vier Überlebenden ein bestimmtes Allel hat, beträgt 1/2, und daher ist die Wahrscheinlichkeit, dass eine bestimmte Allelkombination auftritt, wenn die Lösung schrumpft, gleich
(Die ursprüngliche Populationsgröße ist so groß, dass die Probenahme effektiv ersatzlos erfolgt.) Mit anderen Worten, es ist gleich wahrscheinlich, dass jede der 16 möglichen Allelkombinationen mit einer Wahrscheinlichkeit von 1/16 auftritt.
Zählen der Kombinationen mit der gleichen Anzahl von EIN und B.erhalten wir die folgende Tabelle.
| EIN | B. | Kombinationen | Wahrscheinlichkeit |
| 4 | 0 | 1 | 1/16 |
| 3 | 1 | 4 | 4/16 |
| 2 | 2 | 6 | 6/16 |
| 1 | 3 | 4 | 4/16 |
| 0 | 4 | 1 | 1/16 |
Wie in der Tabelle gezeigt, die Gesamtzahl der Kombinationen mit der gleichen Anzahl von EIN Allele ab B. Allele sind sechs und die Wahrscheinlichkeit dieser Kombination ist 6/16. Die Gesamtzahl der anderen Kombinationen beträgt zehn, also die Wahrscheinlichkeit einer ungleichen Anzahl von EIN und B. Allele ist 10/16. So begann zwar die ursprüngliche Kolonie mit einer gleichen Anzahl von EIN und B. Allele, es ist sehr wahrscheinlich, dass die Anzahl der Allele in der verbleibenden Population von vier Mitgliedern nicht gleich ist. Gleiche Zahlen sind weniger wahrscheinlich als ungleiche Zahlen. Im letzteren Fall ist eine genetische Drift aufgetreten, weil sich die Allelfrequenzen der Population aufgrund von Zufallsstichproben geändert haben. In diesem Beispiel schrumpfte die Bevölkerung auf nur vier zufällige Überlebende, ein Phänomen, das als Bevölkerungsengpass bekannt ist.
Die Wahrscheinlichkeiten für die Anzahl der Kopien des Allels EIN (oder B.), die überleben (in der letzten Spalte der obigen Tabelle angegeben), können direkt aus der Binomialverteilung berechnet werden, wobei die “Erfolgs” -Wahrscheinlichkeit (Wahrscheinlichkeit, dass ein gegebenes Allel vorhanden ist) 1/2 beträgt (dh die Wahrscheinlichkeit, dass es vorhanden ist) k Kopien von EIN (oder B.) Allele in der Kombination) ist gegeben durch
wo n = 4 ist die Anzahl der überlebenden Bakterien.
Mathematische Modelle[edit]
Mathematische Modelle der genetischen Drift können entweder unter Verwendung von Verzweigungsprozessen oder einer Diffusionsgleichung entworfen werden, die Änderungen der Allelfrequenz in einer idealisierten Population beschreibt.[9]
Wright-Fisher-Modell[edit]
Betrachten Sie ein Gen mit zwei Allelen, EIN oder B.. In diploiden Populationen bestehend aus N. Einzelpersonen gibt es 2N. Kopien jedes Gens. Ein Individuum kann zwei Kopien desselben Allels oder zwei verschiedene Allele haben. Wir können die Frequenz eines Allels nennen p und die Frequenz des anderen q. Das Wright-Fisher-Modell (benannt nach Sewall Wright und Ronald Fisher) geht davon aus, dass sich Generationen nicht überlappen (zum Beispiel haben einjährige Pflanzen genau eine Generation pro Jahr) und dass jede Kopie des in der neuen Generation gefundenen Gens unabhängig zufällig ausgewählt wird von allen Kopien des Gens in der alten Generation. Die Formel zur Berechnung der Wahrscheinlichkeit des Erhaltens k Kopien eines Allels, das Häufigkeit hatte p in der letzten Generation ist dann[10][11]
wo das Symbol “!“bezeichnet die Fakultätsfunktion. Dieser Ausdruck kann auch unter Verwendung des Binomialkoeffizienten formuliert werden.
Moran Modell[edit]
Das Moran-Modell geht von überlappenden Generationen aus. In jedem Zeitschritt wird ein Individuum ausgewählt, um sich zu reproduzieren, und ein Individuum wird ausgewählt, um zu sterben. In jedem Zeitschritt kann die Anzahl der Kopien eines bestimmten Allels um eins steigen, um eins sinken oder gleich bleiben. Dies bedeutet, dass die Übergangsmatrix tridiagonal ist, was bedeutet, dass mathematische Lösungen für das Moran-Modell einfacher sind als für das Wright-Fisher-Modell. Andererseits sind Computersimulationen mit dem Wright-Fisher-Modell normalerweise einfacher durchzuführen, da weniger Zeitschritte berechnet werden müssen. Im Moran-Modell dauert es N. Zeitschritte, um durch eine Generation zu kommen, wo N. ist die effektive Bevölkerungsgröße. Im Wright-Fisher-Modell ist nur eines erforderlich.[12]
In der Praxis liefern die Modelle Moran und Wright-Fisher qualitativ ähnliche Ergebnisse, aber die genetische Drift läuft im Moran-Modell doppelt so schnell.
Andere Modelle der Drift[edit]
Wenn die Varianz in der Anzahl der Nachkommen viel größer ist als die, die durch die vom Wright-Fisher-Modell angenommene Binomialverteilung gegeben ist, ist die genetische Drift bei gleicher Gesamtgeschwindigkeit der genetischen Drift (der effektiven Populationsgröße der Varianz) eine weniger starke Kraft im Vergleich zur Auswahl.[13] Selbst bei gleicher Varianz wird die Kraft der genetischen Drift erheblich geschwächt, wenn höhere Momente der Verteilung der Nachkommenzahl die der Binomialverteilung überschreiten.[14]
Andere zufällige Effekte als Stichprobenfehler[edit]
Zufällige Änderungen der Allelfrequenzen können auch durch andere Effekte als Abtastfehler verursacht werden, beispielsweise zufällige Änderungen des Selektionsdrucks.[15]
Eine wichtige alternative Quelle für Stochastizität, die vielleicht wichtiger ist als die genetische Drift, ist der genetische Entwurf.[16] Genetischer Entwurf ist die Auswirkung auf einen Ort durch Auswahl an verknüpften Orten. Die mathematischen Eigenschaften des genetischen Entwurfs unterscheiden sich von denen der genetischen Drift.[17] Die Richtung der zufälligen Änderung der Allelfrequenz ist über Generationen hinweg autokorreliert.[2]
Drift und Fixierung[edit]
Das Hardy-Weinberg-Prinzip besagt, dass in ausreichend großen Populationen die Allelfrequenzen von einer Generation zur nächsten konstant bleiben, es sei denn, das Gleichgewicht wird durch Migration, genetische Mutationen oder Selektion gestört.[18]
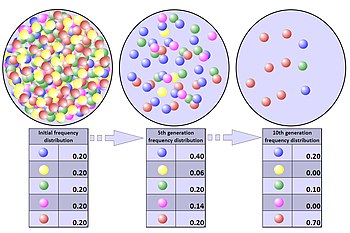
In endlichen Populationen werden jedoch keine neuen Allele aus der zufälligen Stichprobe von Allelen gewonnen, die an die nächste Generation weitergegeben werden. Die Stichprobe kann jedoch dazu führen, dass ein vorhandenes Allel verschwindet. Da zufällige Stichproben ein Allel entfernen, aber nicht ersetzen können und zufällige Abnahmen oder Erhöhungen der Allelfrequenz die erwarteten Allelverteilungen für die nächste Generation beeinflussen, treibt die genetische Drift eine Population im Laufe der Zeit in Richtung genetischer Einheitlichkeit. Wenn ein Allel eine Häufigkeit von 1 (100%) erreicht, wird es in der Population als “fest” bezeichnet, und wenn ein Allel eine Häufigkeit von 0 (0%) erreicht, geht es verloren. Kleinere Populationen erreichen die Fixierung schneller, während im Grenzbereich einer unendlichen Population keine Fixierung erreicht wird. Sobald ein Allel fixiert ist, kommt die genetische Drift zum Stillstand und die Allelfrequenz kann sich nicht ändern, es sei denn, ein neues Allel wird durch Mutation oder Genfluss in die Population eingeführt. Selbst wenn genetische Drift ein zufälliger, richtungsloser Prozess ist, eliminiert sie genetische Variationen im Laufe der Zeit.[19]
Änderungsrate der Allelfrequenz aufgrund von Drift[edit]

Angenommen, genetische Drift ist die einzige evolutionäre Kraft, die danach auf ein Allel wirkt t Generationen in vielen replizierten Populationen, beginnend mit Allelfrequenzen von p und qbeträgt die Varianz der Allelfrequenz über diese Populationen hinweg
- [20]
Zeit zur Fixierung oder zum Verlust[edit]
Unter der Annahme, dass genetische Drift die einzige evolutionäre Kraft ist, die auf ein Allel wirkt, ist die Wahrscheinlichkeit, dass ein Allel irgendwann in der Population fixiert wird, zu einem bestimmten Zeitpunkt einfach seine Häufigkeit in der Population zu diesem Zeitpunkt.[21] Zum Beispiel, wenn die Frequenz p für Allel EIN beträgt 75% und die Häufigkeit q für Allel B. beträgt 25%, dann bei unbegrenzter Zeit die Wahrscheinlichkeit EIN wird letztendlich in der Bevölkerung fixiert wird 75% und die Wahrscheinlichkeit, dass B. wird fest wird 25%.
Die erwartete Anzahl von Generationen für die Fixierung ist proportional zur Populationsgröße, so dass vorausgesagt wird, dass die Fixierung in kleineren Populationen viel schneller erfolgt.[22] Normalerweise wird die effektive Populationsgröße, die kleiner als die Gesamtpopulation ist, verwendet, um diese Wahrscheinlichkeiten zu bestimmen. Die effektive Bevölkerung (N.e) berücksichtigt Faktoren wie das Inzuchtniveau, das Stadium des Lebenszyklus, in dem die Population am kleinsten ist, und die Tatsache, dass einige neutrale Gene genetisch mit anderen verknüpft sind, die ausgewählt werden.[13] Die effektive Populationsgröße ist möglicherweise nicht für jedes Gen in derselben Population gleich.[23]
Eine zukunftsgerichtete Formel zur Annäherung an die erwartete Zeit, bevor ein neutrales Allel durch genetische Drift nach dem Wright-Fisher-Modell fixiert wird, ist
wo T. ist die Anzahl der Generationen, N.e ist die effektive Bevölkerungsgröße und p ist die Anfangsfrequenz für das gegebene Allel. Das Ergebnis ist die Anzahl der Generationen, die voraussichtlich vergehen werden, bevor die Fixierung für ein bestimmtes Allel in einer Population mit einer bestimmten Größe erfolgt (N.e) und Allelfrequenz (p).[24]
Die erwartete Zeit, in der das neutrale Allel durch genetische Drift verloren geht, kann wie folgt berechnet werden:[10]
Wenn eine Mutation nur einmal in einer Population auftritt, die groß genug ist, dass die anfängliche Häufigkeit vernachlässigbar ist, können die Formeln vereinfacht werden[25]
für die durchschnittliche Anzahl von Generationen, die vor der Fixierung einer neutralen Mutation erwartet werden, und
für die durchschnittliche Anzahl von Generationen, die vor dem Verlust einer neutralen Mutation erwartet werden.[26]
Zeit bis zum Verlust sowohl bei Drift als auch bei Mutation[edit]
Die obigen Formeln gelten für ein Allel, das bereits in einer Population vorhanden ist und weder einer Mutation noch einer natürlichen Selektion unterliegt. Wenn ein Allel viel häufiger durch Mutation verloren geht als durch Mutation, kann sowohl die Mutation als auch die Drift die Zeit bis zum Verlust beeinflussen. Wenn das Allel, das zu Mutationsverlust neigt, wie in der Population festgelegt beginnt und durch Mutation mit der Rate m pro Replikation verloren geht, ist die erwartete Zeit in Generationen bis zu seinem Verlust in einer haploiden Population gegeben durch
wo
ist Eulers Konstante.[27] Die erste Annäherung stellt die Wartezeit dar, bis die erste Mutante zum Verlust bestimmt ist, wobei der Verlust dann relativ schnell durch genetische Drift auftritt und Zeit benötigt N.e ≪ 1 /m. Die zweite Näherung stellt die Zeit dar, die für den deterministischen Verlust durch Mutationsakkumulation benötigt wird. In beiden Fällen wird die Zeit bis zur Fixierung von einer Mutation über den Term 1 / dominiertmund ist weniger von der effektiven Bevölkerungsgröße betroffen.
Versus natürliche Auslese[edit]
In natürlichen Populationen wirken genetische Drift und natürliche Selektion nicht isoliert. Beide Phänomene spielen immer eine Rolle, zusammen mit Mutation und Migration. Die neutrale Evolution ist das Produkt von Mutation und Drift, nicht nur von Drift. Auch wenn die Selektion die genetische Drift überwältigt, kann sie nur auf die Variation wirken, die die Mutation bietet.
Während die natürliche Selektion eine Richtung hat, die die Evolution zu vererbbaren Anpassungen an die aktuelle Umgebung führt, hat die genetische Drift keine Richtung und wird nur von der Mathematik des Zufalls geleitet.[28] Infolgedessen wirkt die Drift auf die genotypischen Frequenzen innerhalb einer Population, unabhängig von ihren phänotypischen Wirkungen. Im Gegensatz dazu begünstigt die Selektion die Ausbreitung von Allelen, deren phänotypische Effekte das Überleben und / oder die Reproduktion ihrer Träger erhöhen, senkt die Häufigkeit von Allelen, die ungünstige Eigenschaften verursachen, und ignoriert diejenigen, die neutral sind.[29]
Das Gesetz der großen Zahlen sagt voraus, dass, wenn die absolute Anzahl der Kopien des Allels klein ist (z. B. in kleinen Populationen), die Drift auf den Allelfrequenzen pro Generation größer ist. Die Größe der Drift ist groß genug, um die Selektion bei jeder Allelfrequenz zu überwältigen, wenn der Selektionskoeffizient kleiner als 1 geteilt durch die effektive Populationsgröße ist. Die nicht adaptive Evolution, die aus dem Produkt von Mutation und genetischer Drift resultiert, wird daher als Folgemechanismus des evolutionären Wandels hauptsächlich in kleinen, isolierten Populationen angesehen.[30] Die Mathematik der genetischen Drift hängt von der effektiven Populationsgröße ab, es ist jedoch nicht klar, wie dies mit der tatsächlichen Anzahl von Individuen in einer Population zusammenhängt.[16]Die genetische Verknüpfung mit anderen Genen, die ausgewählt werden, kann die effektive Populationsgröße eines neutralen Allels verringern. Mit einer höheren Rekombinationsrate nimmt die Verknüpfung ab und damit dieser lokale Effekt auf die effektive Populationsgröße.[31][32] Dieser Effekt ist in molekularen Daten als Korrelation zwischen lokaler Rekombinationsrate und genetischer Vielfalt sichtbar.[33] und negative Korrelation zwischen Gendichte und Diversität in nichtkodierenden DNA-Regionen.[34] Die Stochastizität, die mit der Verknüpfung mit anderen Genen verbunden ist, die ausgewählt werden, ist nicht dasselbe wie ein Stichprobenfehler und wird manchmal als genetischer Entwurf bezeichnet, um ihn von der genetischen Drift zu unterscheiden.[16]
Wenn die Allelfrequenz sehr klein ist, kann die Drift auch in großen Populationen die Selektion überwältigen. Während zum Beispiel nachteilige Mutationen in großen Populationen normalerweise schnell eliminiert werden, sind neue vorteilhafte Mutationen fast genauso anfällig für Verluste durch genetische Drift wie neutrale Mutationen. Erst wenn die Allelfrequenz für die vorteilhafte Mutation eine bestimmte Schwelle erreicht, hat die genetische Drift keine Wirkung.[29]
Bevölkerungsengpass[edit]
Ein Bevölkerungsengpass liegt vor, wenn sich eine Bevölkerung aufgrund eines zufälligen Umweltereignisses innerhalb kurzer Zeit zu einer erheblich kleineren Größe zusammenzieht. In einem echten Bevölkerungsengpass sind die Überlebenschancen eines Mitglieds der Bevölkerung rein zufällig und werden durch keinen bestimmten inhärenten genetischen Vorteil verbessert. Der Engpass kann zu radikalen Änderungen der Allelfrequenzen führen, völlig unabhängig von der Auswahl.[35]
Die Auswirkungen eines Bevölkerungsengpasses können auch dann anhalten, wenn der Engpass durch ein einmaliges Ereignis wie eine Naturkatastrophe verursacht wird. Ein interessantes Beispiel für einen Engpass, der zu einer ungewöhnlichen genetischen Verteilung führt, ist der relativ hohe Anteil von Personen mit totaler Stäbchenblindheit (Achromatopsie) auf dem Pingelap-Atoll in Mikronesien. Nach einem Engpass nimmt die Inzucht zu. Dies erhöht den Schaden, der durch rezessive schädliche Mutationen in einem als Inzuchtdepression bekannten Prozess verursacht wird. Die schlimmsten dieser Mutationen werden gegen diese ausgewählt, was zum Verlust anderer Allele führt, die genetisch mit ihnen verbunden sind, und zwar in einem Prozess der Hintergrundauswahl.[2] Bei rezessiven schädlichen Mutationen kann diese Selektion infolge des Engpasses aufgrund genetischer Spülung verstärkt werden. Dies führt zu einem weiteren Verlust der genetischen Vielfalt. Darüber hinaus erhöht eine anhaltende Verringerung der Populationsgröße die Wahrscheinlichkeit weiterer Allelschwankungen aufgrund der Drift in den kommenden Generationen.
Die genetische Variation einer Population kann durch einen Engpass stark reduziert werden, und selbst vorteilhafte Anpassungen können dauerhaft beseitigt werden.[36] Der Variationsverlust macht die überlebende Bevölkerung anfällig für neuen Selektionsdruck wie Krankheit, Klimawandel oder Verschiebung der verfügbaren Nahrungsquelle, da die Anpassung an Umweltveränderungen eine ausreichende genetische Variation in der Bevölkerung erfordert, damit eine natürliche Selektion stattfinden kann.[37][38]
In der jüngeren Vergangenheit sind viele Fälle von Bevölkerungsengpässen bekannt. Vor der Ankunft der Europäer waren nordamerikanische Prärien Lebensraum für Millionen größerer Präriehühner. Allein in Illinois sank ihre Zahl von etwa 100 Millionen Vögeln im Jahr 1900 auf etwa 50 Vögel in den neunziger Jahren. Der Bevölkerungsrückgang resultierte aus der Jagd und der Zerstörung von Lebensräumen. Eine Folge war jedoch der Verlust eines Großteils der genetischen Vielfalt der Arten. Eine DNA-Analyse, bei der Vögel aus der Mitte des Jahrhunderts mit Vögeln aus den neunziger Jahren verglichen wurden, dokumentiert einen starken Rückgang der genetischen Variation in den letzten Jahrzehnten. Derzeit hat das größere Präriehuhn einen geringen Fortpflanzungserfolg.[39]
Der durch Engpass und genetische Drift verursachte genetische Verlust kann jedoch die Fitness erhöhen, wie in Ehrlichia.[40]
Überjagd verursachte im 19. Jahrhundert auch einen schweren Populationsengpass im nördlichen Seeelefanten. Der daraus resultierende Rückgang der genetischen Variation lässt sich durch einen Vergleich mit dem des südlichen Seeelefanten ableiten, der nicht so aggressiv gejagt wurde.[41]
Gründereffekt[edit]
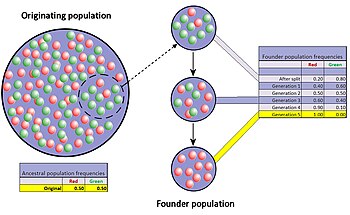
Der Gründereffekt ist ein Sonderfall eines Bevölkerungsengpasses, der auftritt, wenn eine kleine Gruppe in einer Bevölkerung von der ursprünglichen Bevölkerung abplatzt und eine neue bildet. Es wird erwartet, dass die Zufallsstichprobe von Allelen in der gerade gebildeten neuen Kolonie die ursprüngliche Population zumindest in gewisser Hinsicht grob falsch darstellt.[42] Es ist sogar möglich, dass die Anzahl der Allele für einige Gene in der ursprünglichen Population größer ist als die Anzahl der Genkopien in den Gründern, was eine vollständige Darstellung unmöglich macht. Wenn eine neu gebildete Kolonie klein ist, können ihre Gründer das Erbgut der Bevölkerung bis weit in die Zukunft stark beeinflussen.
Ein gut dokumentiertes Beispiel findet sich in der Migration der Amish nach Pennsylvania im Jahr 1744. Zwei Mitglieder der neuen Kolonie teilten sich das rezessive Allel für das Ellis-van-Creveld-Syndrom. Mitglieder der Kolonie und ihre Nachkommen sind in der Regel religiöse Isolate und bleiben relativ insular. Infolge vieler Inzuchtgenerationen ist das Ellis-van-Creveld-Syndrom bei den Amish heute weitaus häufiger als in der Allgemeinbevölkerung.[29][43]
Der Unterschied in den Genfrequenzen zwischen der ursprünglichen Population und der Kolonie kann auch dazu führen, dass die beiden Gruppen im Laufe vieler Generationen signifikant voneinander abweichen. Wenn der Unterschied oder die genetische Distanz zunimmt, können sich die beiden getrennten Populationen sowohl genetisch als auch phänetisch unterscheiden, obwohl nicht nur die genetische Drift, sondern auch die natürliche Selektion, der Genfluss und die Mutation zu dieser Divergenz beitragen. Dieses Potenzial für relativ schnelle Änderungen der Genfrequenz der Kolonie veranlasste die meisten Wissenschaftler, den Gründereffekt (und damit auch die genetische Drift) als wesentliche Triebkraft für die Entwicklung neuer Arten zu betrachten. Sewall Wright war der erste, der diese Bedeutung mit seiner Verschiebungstheorie der Speziation auf zufällige Drift und kleine, neu isolierte Populationen legte.[44] Nach Wright schuf Ernst Mayr viele überzeugende Modelle, um zu zeigen, dass der Rückgang der genetischen Variation und die geringe Populationsgröße nach dem Gründereffekt für die Entwicklung neuer Arten von entscheidender Bedeutung waren.[45] Diese Ansicht wird heute jedoch viel weniger unterstützt, da die Hypothese wiederholt durch experimentelle Untersuchungen überprüft wurde und die Ergebnisse bestenfalls zweideutig waren.[46]
Geschichte[edit]
Die Rolle des Zufalls in der Evolution wurde erstmals 1921 von Arend L. Hagedoorn und AC Hagedoorn-Vorstheuvel La BrandHagedoorn beschrieben.[47] Sie betonten, dass das zufällige Überleben eine Schlüsselrolle beim Verlust der Variation von Populationen spielt. Fisher (1922) antwortete darauf mit der ersten, wenn auch geringfügig falschen mathematischen Behandlung des “Hagedoorn-Effekts”.[48] Insbesondere erwartete er, dass viele natürliche Populationen zu groß waren (N ~ 10.000), als dass die Auswirkungen der Drift wesentlich wären, und dass die Drift eine unbedeutende Auswirkung auf den Evolutionsprozess haben würde. Die korrigierte mathematische Behandlung und der Begriff “genetische Drift” wurden später von einem Begründer der Populationsgenetik, Sewall Wright, geprägt. Seine erste Verwendung des Begriffs “Drift” war 1929,[49] obwohl er es zu der Zeit im Sinne eines gerichteten Veränderungsprozesses oder einer natürlichen Selektion benutzte. Zufällige Drift durch Stichprobenfehler wurde als “Sewall-Wright-Effekt” bekannt, obwohl es ihm nie ganz angenehm war, seinen Namen zu sehen. Wright bezeichnete alle Änderungen der Allelfrequenz entweder als “stetige Drift” (z. B. Auswahl) oder “zufällige Drift” (z. B. Stichprobenfehler).[50] “Drift” wurde ausschließlich als Fachbegriff im stochastischen Sinne übernommen.[51] Heutzutage ist es in der Regel noch enger definiert, was den Stichprobenfehler betrifft.[52] obwohl diese enge Definition nicht universell ist.[53][54] Wright schrieb, dass die “Beschränkung der” zufälligen Drift “oder sogar der” Drift “auf nur eine Komponente, die Auswirkungen von Stichprobenunfällen, zu Verwirrung führt.”[50] Sewall Wright betrachtete den Prozess der zufälligen genetischen Drift anhand eines Stichprobenfehlers, der dem durch Inzucht entspricht, aber spätere Arbeiten haben gezeigt, dass sie unterschiedlich sind.[55]
In den frühen Tagen der modernen Evolutionssynthese begannen Wissenschaftler, die neue Wissenschaft der Populationsgenetik mit Charles Darwins Theorie der natürlichen Selektion zu verbinden. In diesem Rahmen konzentrierte sich Wright auf die Auswirkungen der Inzucht auf kleine, relativ isolierte Populationen. Er führte das Konzept einer adaptiven Landschaft ein, in der Phänomene wie Kreuzung und genetische Drift in kleinen Populationen sie von adaptiven Peaks wegdrücken könnten, was wiederum die natürliche Selektion ermöglicht, sie in Richtung neuer adaptiver Peaks zu treiben.[56] Wright war der Ansicht, dass kleinere Populationen besser für die natürliche Selektion geeignet sind, da “Inzucht ausreichend intensiv war, um neue Interaktionssysteme durch zufällige Drift zu erzeugen, aber nicht intensiv genug, um eine zufällige nichtadaptive Fixierung von Genen zu bewirken”.[57]
Wrights Ansichten über die Rolle der genetischen Drift im Evolutionsschema waren fast von Anfang an umstritten. Einer der lautstärksten und einflussreichsten Kritiker war Kollege Ronald Fisher. Fisher räumte ein, dass genetische Drift eine gewisse Rolle in der Evolution spielte, aber eine unbedeutende. Fisher wurde vorgeworfen, Wrights Ansichten missverstanden zu haben, weil Fisher in seiner Kritik zu argumentieren schien, Wright habe die Auswahl fast vollständig abgelehnt. Für Fisher war es der einzige Weg, die ständig zunehmende Komplexität einfacherer Formen zu erklären, den Evolutionsprozess als einen langen, stetigen, adaptiven Fortschritt zu betrachten. Aber die Debatten zwischen den “Gradualisten” und denen, die sich mehr dem Wright-Modell der Evolution zuwenden, wo Selektion und Drift zusammen eine wichtige Rolle spielen, wurden fortgesetzt.[58]
1968 entfachte Motoo Kimura die Debatte mit seiner neutralen Theorie der molekularen Evolution, die behauptet, dass die meisten genetischen Veränderungen durch genetische Drift verursacht werden, die auf neutrale Mutationen einwirkt.[6][7]
Die Rolle der genetischen Drift durch Stichprobenfehler in der Evolution wurde von John H. Gillespie kritisiert[59] und William B. Provine, der argumentiert, dass die Auswahl auf verknüpften Websites eine wichtigere stochastische Kraft ist.
Siehe auch[edit]
Notizen und Referenzen[edit]
- ^ Gould SJ (2002). “Kapitel 7, Abschnitt” Synthese als Härtung“”“. Die Struktur der Evolutionstheorie.
- ^ ein b c Masel J (Oktober 2011). “Genetische Drift”. Aktuelle Biologie. Cell Press. 21 (20): R837-8. doi:10.1016 / j.cub.2011.08.007. PMID 22032182.
- ^ Futuyma 1998, Glossar
- ^ Stern B, Spencer HG (Mai 2013). “Auswirkungen der genetischen Drift und des Genflusses auf die selektive Aufrechterhaltung der genetischen Variation”. Genetik. 194 (1): 235–44. doi:10.1534 / Genetik.113.149781. PMC 3632471. PMID 23457235.
- ^ Miller 2000, p. 54
- ^ ein b Kimura M (Februar 1968). “Evolutionsrate auf molekularer Ebene”. Natur. Nature Publishing Group. 217 (5129): 624–6. Bibcode:1968Natur.217..624K. doi:10.1038 / 217624a0. PMID 5637732. S2CID 4161261.
- ^ ein b Futuyma 1998, p. 320
- ^ “Stichprobenfehler und Evolution”. Evolution verstehen. Universität von Kalifornien, Berkeley. Archiviert vom Original am 8. Dezember 2015. Abgerufen 1. Dezember 2015.
- ^ Wahl LM (August 2011). “Fixierung, wenn N und s variieren: Klassische Ansätze liefern elegante neue Ergebnisse”. Genetik. Genetics Society of America. 188 (4): 783–5. doi:10.1534 / Genetik.111.131748. PMC 3176088. PMID 21828279.
- ^ ein b Hartl & Clark 2007, p. 112
- ^ Tian 2008, p. 11
- ^ Moran PA (1958). “Zufällige Prozesse in der Genetik”. Mathematische Verfahren der Cambridge Philosophical Society. 54 (1): 60–71. Bibcode:1958PCPS … 54 … 60M. doi:10.1017 / S0305004100033193.
- ^ ein b Charlesworth B (März 2009). “Grundlegende Konzepte in der Genetik: effektive Populationsgröße und Muster der molekularen Evolution und Variation”. Natur Bewertungen. Genetik. Nature Publishing Group. 10 (3): 195–205. doi:10.1038 / nrg2526. PMID 19204717. S2CID 205484393.
- ^ Der R, Epstein CL, Plotkin JB (September 2011). “Verallgemeinerte Populationsmodelle und die Art der genetischen Drift”. Theoretische Populationsbiologie. Elsevier. 80 (2): 80–99. doi:10.1016 / j.tpb.2011.06.004. PMID 21718713.
- ^ Li & Graur 1991, p. 28
- ^ ein b c Gillespie JH (November 2001). “Ist die Populationsgröße einer Art für ihre Entwicklung relevant?”. Evolution; Internationale Zeitschrift für organische Evolution. John Wiley & Sons für die Gesellschaft zur Erforschung der Evolution. 55 (11): 2161–9. doi:10.1111 / j.0014-3820.2001.tb00732.x. PMID 11794777. S2CID 221735887.
- ^ Neher RA, Shraiman BI (August 2011). “Genetischer Entwurf und Quasi-Neutralität in großen fakultativ sexuellen Populationen”. Genetik. Genetics Society of America. 188 (4): 975–96. arXiv:1108.1635. doi:10.1534 / Genetik.111.128876. PMC 3176096. PMID 21625002.
- ^ Ewens 2004
- ^ Li & Graur 1991, p. 29
- ^ Barton et al. 2007, p. 417
- ^ Futuyma 1998, p. 300
- ^ Otto SP, Whitlock MC (Juni 1997). “Die Wahrscheinlichkeit der Fixierung in Populationen mit wechselnder Größe” (PDF). Genetik. Genetics Society of America. 146 (2): 723–33. PMC 1208011. PMID 9178020. Archiviert (PDF) vom Original am 19. März 2015.
- ^ Cutter AD, Choi JY (August 2010). “Natürliche Selektion prägt den Nukleotidpolymorphismus im gesamten Genom des Fadenwurms Caenorhabditis briggsae.”. Genomforschung. Cold Spring Harbor Laboratory Press. 20 (8): 1103–11. doi:10.1101 / gr.104331.109. PMC 2909573. PMID 20508143.
- ^ Hedrick 2005, p. 315
- ^ Li & Graur 1991, p. 33
- ^ Kimura & Ohta 1971
- ^ Masel J, König OD, Maughan H (Januar 2007). “Der Verlust der adaptiven Plastizität während langer Perioden der Umweltstase”. Der amerikanische Naturforscher. University of Chicago Press im Auftrag der American Society of Naturalists. 169 (1): 38–46. doi:10.1086 / 510212. PMC 1766558. PMID 17206583.
- ^ “Natürliche Auslese: Wie Evolution funktioniert”. Actionbioscience. Washington, DC: Amerikanisches Institut für Biowissenschaften. Archiviert vom Original am 6. Januar 2010. Abgerufen 24. November 2009. Ein Interview mit Douglas J. Futuyma. Siehe Antwort auf Frage: Ist natürliche Selektion der einzige Mechanismus der Evolution?
- ^ ein b c Cavalli-Sforza, Menozzi & Piazza 1996
- ^ Zimmer 2001
- ^ Golding 1994, p. 46
- ^ Charlesworth B., Morgan MT, Charlesworth D. (August 1993). “Die Wirkung schädlicher Mutationen auf die neutrale molekulare Variation” (PDF). Genetik. Genetics Society of America. 134 (4): 1289–303. PMC 1205596. PMID 8375663.
- ^ Presgraves DC (September 2005). “Rekombination verbessert die Proteinanpassung in Drosophila melanogaster”. Aktuelle Biologie. Cell Press. 15 (18): 1651–6. doi:10.1016 / j.cub.2005.07.065. PMID 16169487. S2CID 15120927.
- ^ Nordborg M., Hu TT, Ishino Y., Jhaveri J., Toomajian C., Zheng H., Bakker E., Calabrese P., Gladstone J., Goyal R., Jakobsson M., Kim S., Morozov Y., Padhukasahasram B., Plagnol V., Rosenberg NA, Shah C. Wall JD, Wang J., Zhao K., Kalbfleisch T., Schulz V., Kreitman M., Bergelson J. (Juli 2005). “Das Muster des Polymorphismus bei Arabidopsis thaliana”. PLOS Biologie. Öffentliche Wissenschaftsbibliothek. 3 (7): e196. doi:10.1371 / journal.pbio.0030196. PMC 1135296. PMID 15907155.

- ^ Robinson R, ed. (2003). “Bevölkerungsengpass”. Genetik. 3. New York: Macmillan Reference USA. ISBN 0-02-865609-1. LCCN 2002003560. OCLC 614996575. Abgerufen 14. Dezember 2015.
- ^ Futuyma 1998, S. 303–304
- ^ O’Corry-Crowe G (März 2008). “Klimawandel und molekulare Ökologie arktischer Meeressäuger”. Ökologische Anwendungen. Ökologische Gesellschaft von Amerika. 18 (2 Suppl): S56-76. doi:10.1890 / 06-0795.1. PMID 18494363.
- ^ Cornuet JM, Luikart G (Dezember 1996). “Beschreibung und Leistungsanalyse von zwei Tests zur Erkennung der jüngsten Populationsengpässe anhand von Allelfrequenzdaten”. Genetik. Genetics Society of America. 144 (4): 2001–14. PMC 1207747. PMID 8978083.
- ^ Sadava et al. 2008, chpts. 1, 21–33, 52–57
- ^ Dale C, Moran NA (August 2006). “Molekulare Wechselwirkungen zwischen bakteriellen Symbionten und ihren Wirten”. Zelle. 126 (3): 453–65. doi:10.1016 / j.cell.2006.07.014. PMID 16901780. S2CID 15985536.
- ^ “Engpässe und Gründereffekte”. Evolution verstehen. Universität von Kalifornien, Berkeley. Archiviert vom Original am 4. Dezember 2015. Abgerufen 14. Dezember 2015.
- ^ Campbell 1996, p. 423
- ^ “Genetische Drift und der Gründereffekt”. Evolutionsbibliothek (Webressource). Evolution. Boston, MA: WGBH-Bildungsstiftung; Clear Blue Sky Productions, Inc. 2001. OCLC 48165595. Archiviert vom Original am 14. März 2009. Abgerufen 7. April 2009.
- ^ Wolf, Brodie & Wade 2000
- ^ Hey, Fitch & Ayala 2005
- ^ Howard & Berlocher 1998
- ^ Hagedoorn, AL; Hagedoorn-Vorstheuvel La Brand, AC (1921). Der relative Wert der Prozesse, die die Evolution verursachen. Den Haag: Martinus Nijhoff.
- ^ Fisher, RA (1922). “Über das Dominanzverhältnis”. Verfahren der Royal Society of Edinburgh. 42: 321–341. doi:10.1017 / s0370164600023993.
- ^ Wright S (November – Dezember 1929). “Die Entwicklung der Dominanz”. Der amerikanische Naturforscher. Chicago, IL: University of Chicago Press im Auftrag der American Society of Naturalists. 63 (689): 556–561. doi:10.1086 / 280290. ISSN 0003-0147. JSTOR 2456825.
- ^ ein b Wright S (1955). “Klassifikation der Evolutionsfaktoren”. Cold Spring Harbor Symposien zur quantitativen Biologie. Cold Spring Harbor, NY: Laborpresse von Cold Spring Harbor. 20: 16–24. doi:10.1101 / SQB.1955.020.01.004. ISSN 0091-7451. PMID 13433551. Symposium: “Populationsgenetik: Art und Ursachen der genetischen Variabilität in Populationen”.
- ^ Stevenson 1991
- ^ Freeman & Herron 2007
- ^ Masel J (August 2012). “Hardy-Weinberg und genetische Drift in der Bachelor-Biologie neu denken”. BioEssays. John Wiley & Sons. 34 (8): 701–10. doi:10.1002 / bies.201100178. PMID 22576789. S2CID 28513167.
- ^ Lynch 2007
- ^ Crow JF (März 2010). “Wright und Fisher über Inzucht und zufällige Drift”. Genetik. Genetics Society of America. 184 (3): 609–11. doi:10.1534 / Genetik.109.110023. PMC 2845331. PMID 20332416.
- ^ Larson 2004, S. 221–243
- ^ Stevenson 1991: Zitat William B. Provine zugeschrieben in Die Ursprünge der theoretischen Populationsgenetik (1971), p. 162; Chicago: University of Chicago Press.
- ^ Avers 1989
- ^ Gillespie JH (Juni 2000). “Genetische Drift in einer unendlichen Population. Das Pseudohitchhiking-Modell”. Genetik. Genetics Society of America. 155 (2): 909–19. PMC 1461093. PMID 10835409.
Literaturverzeichnis[edit]
- Avers CJ (1989). Prozess und Muster in der Evolution. New York: Oxford University Press. ISBN 0-19-505275-7. LCCN 88005368. OCLC 17677554.
- Barton NH, Briggs DE, Eisen JA, Goldstein DB, Patel NH (2007). Evolution. Cold Spring Harbor, NY: Laborpresse von Cold Spring Harbor. ISBN 978-0-87969-684-9. LCCN 2007010767. OCLC 86090399.
- Campbell NA (1996). Biologie. Benjamin / Cummings-Reihe in den Biowissenschaften (4. Aufl.). Menlo Park, Kalifornien: Benjamin / Cummings Pub. Co. ISBN 0-8053-1940-9. LCCN 95045572. OCLC 33333455.
- Cavalli-Sforza LL, Menozzi P., Piazza A. (1996). Die Geschichte und Geographie menschlicher Gene (Kurzes Taschenbuch ed.). Princeton, NJ: Princeton University Press. ISBN 0-691-02905-9. OCLC 35527063.
- Ewens WJ (2004). Mathematische Populationsgenetik I. Theoretische Einführung. Interdisziplinäre Angewandte Mathematik. 27 (2. Aufl.). New York: Springer-Verlag. ISBN 0-387-20191-2. LCCN 2003065728. OCLC 53231891.
- Freeman S, Herron JC (2007). Evolutionsanalyse (4. Aufl.). Upper Saddle River, New Jersey: Pearson Prentice Hall. ISBN 978-0-13-227584-2. LCCN 2006034384. OCLC 73502978.
- Futuyma D (1998). Evolutionsbiologie (3. Aufl.). Sunderland, MA: Sinauer Associates. ISBN 0-87893-189-9. LCCN 97037947. OCLC 37560100.
- Golding B, hrsg. (1994). Nicht neutrale Evolution: Theorien und molekulare Daten. New York: Chapman & Hall. ISBN 0-412-05391-8. LCCN 93047006. OCLC 29638235. “Beiträge aus einem Workshop, der vom Canadian Institute for Advanced Research gesponsert wurde.”
- Hartl DL, Clark AG (2007). Prinzipien der Populationsgenetik (4. Aufl.). Sunderland, MA: Sinauer Associates. ISBN 978-0-87893-308-2. LCCN 2006036153. OCLC 75087956.
- Hedrick PW (2005). Genetik von Populationen (3. Aufl.). Boston, MA: Jones und Bartlett Publishers. ISBN 0-7637-4772-6. LCCN 2004056666. OCLC 56194719.
- Hey J, Fitch WM, Ayala FJ, Hrsg. (2005). Systematik und Ursprung der Arten: Zum 100. Jahrestag von Ernst Mayr. Washington, DC: National Academies Press. ISBN 978-0-309-09536-5. LCCN 2005017917. OCLC 70745851.
- Howard DJ, Berlocher SH, Hrsg. (1998). Endlose Formen: Spezies und Speziation. New York: Oxford University Press. ISBN 978-0-19-510901-6. LCCN 97031461. OCLC 37545522.
- Kimura M., Ohta T. (1971). Theoretische Aspekte der Populationsgenetik. Monographien in der Populationsbiologie. 4. Princeton, NJ: Princeton University Press. S. 1–219. ISBN 0-691-08096-8. LCCN 75155963. OCLC 299867647. PMID 5162676.
- Larson EJ (2004). Evolution: Die bemerkenswerte Geschichte einer wissenschaftlichen Theorie. Moderne Bibliothekschroniken. 17. New York: Moderne Bibliothek. ISBN 0-679-64288-9. LCCN 2003064888. OCLC 53483597.
- Li W, Graur D (1991). Grundlagen der molekularen Evolution. Sunderland, MA: Sinauer Associates. ISBN 0-87893-452-9. LCCN 90043581. OCLC 22113526.
- Lynch M (2007). Die Ursprünge der Genomarchitektur. Sunderland, MA: Sinauer Associates. ISBN 978-0-87893-484-3. LCCN 2007000012. OCLC 77574049.
- Miller G (2000). Der Paarungsgeist: Wie sexuelle Entscheidungen die Evolution der menschlichen Natur prägten. New York: Doppelter Tag. ISBN 0-385-49516-1. LCCN 00022673. OCLC 43648482.
- Sadava D, Heller HC, Orians GH, Purves WK, Hillis DM (2008). Leben: Die Wissenschaft der Biologie. II: Evolution, Vielfalt und Ökologie (8. Aufl.). Sunderland, MA; Gordonsville, VA: Sinauer Associates; WH Freeman und Company. ISBN 978-0-7167-7674-1. LCCN 2006031320. OCLC 71632224.
- Stevenson JC (1991). Wörterbuch der Konzepte in der physikalischen Anthropologie. Referenzquellen für die Sozial- und Geisteswissenschaften. 10. Westport, CT: Greenwood Press. ISBN 0-313-24756-0. LCCN 90022815. OCLC 22732327.
- Tian JP (2008). Evolutionsalgebren und ihre Anwendungen. Vorlesungsunterlagen in Mathematik. 1921. Berlin; New York: Springer. doi:10.1007 / 978-3-540-74284-5. ISBN 978-3-540-74283-8. LCCN 2007933498. OCLC 173807298. Zbl 1136.17001.
- Wolf JB, Brodie ED, Wade MJ, Hrsg. (2000). Epistase und der Evolutionsprozess. Oxford, Großbritannien; New York: Oxford University Press. ISBN 0-19-512806-0. LCCN 99046515. OCLC 42603105.
- Zimmer C (2001). Evolution: Der Triumph einer Idee. Einführung von Stephen Jay Gould; Vorwort von Richard Hutton (1. Aufl.). New York: HarperCollins. ISBN 0-06-019906-7. LCCN 2001024077. OCLC 46359440.
Externe Links[edit]









![{ displaystyle { bar {T}} _ { text {lost}} approx { begin {case} { dfrac {1} {m}}, { text {if}} mN_ {e} ll 1 \[8pt]{ dfrac { ln {(mN_ {e})} + gamma} {m}} { text {if}} mN_ {e} gg 1 end {case}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0dca9b7dc746a7b2a8d8a63770ac53782d1639e3)
 ist Eulers Konstante.[27] Die erste Annäherung stellt die Wartezeit dar, bis die erste Mutante zum Verlust bestimmt ist, wobei der Verlust dann relativ schnell durch genetische Drift auftritt und Zeit benötigt N.e ≪ 1 /m. Die zweite Näherung stellt die Zeit dar, die für den deterministischen Verlust durch Mutationsakkumulation benötigt wird. In beiden Fällen wird die Zeit bis zur Fixierung von einer Mutation über den Term 1 / dominiertmund ist weniger von der effektiven Bevölkerungsgröße betroffen.
ist Eulers Konstante.[27] Die erste Annäherung stellt die Wartezeit dar, bis die erste Mutante zum Verlust bestimmt ist, wobei der Verlust dann relativ schnell durch genetische Drift auftritt und Zeit benötigt N.e ≪ 1 /m. Die zweite Näherung stellt die Zeit dar, die für den deterministischen Verlust durch Mutationsakkumulation benötigt wird. In beiden Fällen wird die Zeit bis zur Fixierung von einer Mutation über den Term 1 / dominiertmund ist weniger von der effektiven Bevölkerungsgröße betroffen.
Recent Comments